انسانی جینومکس
فطرت جینیات
طویل پڑھنے والی ترتیب نیورونل انٹرا نیوکلیئر انکلوژن بیماری سے وابستہ NOTCH2NLC میں GGC کی دوبارہ توسیع کی نشاندہی کرتی ہے۔
ONT ریسیونسنگ |Illumina |مکمل exome ترتیب |CRISPR-Cas9 ONT ہدف شدہ ترتیب |RNA-seq |ONT 5mC میتھیلیشن کالنگ
جھلکیاں
1. ایک بڑے NIID خاندان پر لنکج تجزیہ کے ذریعے، دو منسلک علاقوں کی نشاندہی کی گئی۔
2.ONT-based long-read sequencing اور Cas-9 ثالثی افزودگی ONT کی ترتیب نے NOTCH2NLC کے 5′ UTR میں NIID، GGC کی دوبارہ توسیع کی ایک ممکنہ جینیاتی وجہ دریافت کی۔اس مطالعہ نے پہلی بار انسانی مخصوص جینوں میں دوبارہ توسیع کی اطلاع دی جو قطعاتی نقلوں کے ذریعے تیار ہوئی۔
3.RNA کی ترتیب نے NOTCH2NLC میں شروع میں یا GGC کے دوبارہ توسیعی علاقوں کے اندر غیر معمولی اینٹی سینس ٹرانسکرپٹس کا انکشاف کیا۔
پس منظر
Nیورونل انٹرا نیوکلیئر انکلوژن بیماری (NIID) ایک ترقی پسند اور مہلک نیوروڈیجینریٹیو بیماری ہے، جو مرکزی اور پردیی اعصابی نظاموں میں eosinophilic hyaline intranuclear inclusions کی موجودگی کی خصوصیت ہے۔اس کے انتہائی متغیر طبی مظاہر جلد کی بایپسی کے تعارف تک تشخیص میں بڑی مشکلات پیدا کرتے ہیں۔تاہم، ہسٹوپیتھولوجی پر مبنی طریقے اب بھی غلط تشخیص کا شکار ہیں، جو NIID کی جینیاتی تفہیم کا مطالبہ کر رہا ہے۔
کامیابیاں
ربط کا تجزیہ
Sہارٹ ریڈ سیکوینسنگ پر مبنی پوری جینوم سیکوینسنگ (WGS) اور پوری exome sequencing (WES) ایک بڑے NIID فیملی (13 متاثرہ اور 7 غیر متاثرہ ممبران) پر کی گئی تھی۔ان اعداد و شمار سے نکالے گئے SNPs پر ربط کے تجزیے سے صرف دو منسلک خطوں کا انکشاف ہوا: 1p36.31-p36.22 پر ایک 3.5 Mb خطہ (زیادہ سے زیادہ LOD=2.32) اور 58.1 Mb خطہ 1p22.1-q21.3 پر (زیادہ سے زیادہ LOD: 4.21 )۔تاہم، ان منسلک علاقوں میں کسی روگجنک SNPs یا CNVs کی شناخت نہیں کی گئی۔
NOTCH2NLC میں GGC کی دوبارہ توسیع
N8 خاندانوں کے 13 متاثرہ اور 4 غیر متاثرہ ارکان پر اینوپور پر مبنی ترتیب پر کارروائی کی گئی تھی (ایک اور متاثرہ رکن کو Pacbio لانگ ریڈ سیکوینسنگ پلیٹ فارم کے ذریعے ترتیب دیا گیا تھا۔)طویل پڑھے جانے والے اعداد و شمار سے پتہ چلتا ہے کہ بیماری سے منسلک GGC کی 5′ UTR میں NOTCH2NLC جین میپنگ کو 58.1 Mb سے منسلک خطہ (شکل 1) میں دہرایا گیا ہے۔RP-PCR کے ذریعہ جانچے گئے تمام 40 چھٹپٹ NIID کیسوں میں بھی ان بار بار توسیع کی نشاندہی کی گئی۔
CNOTCH2NLC ریپیٹ (100 X-1,795 X) پر اعلی پڑھنے کی کوریج حاصل کرنے کے لیے نانو پور پلیٹ فارم پر 9 ثالثی ہدف کی ترتیب کو استعمال کیا گیا تھا۔یہ اتفاق رائے کے سلسلے GGC کی دوبارہ توسیع کے بارے میں پچھلے نتائج سے اچھی طرح متفق ہیں۔مزید برآں، {(GGA)n (GGC)n}n تکرار کی شناخت کمزوری غالب فینوٹائپ (شکل 2) کے لیے ممکنہ جینیاتی مارکر کے طور پر کی گئی۔
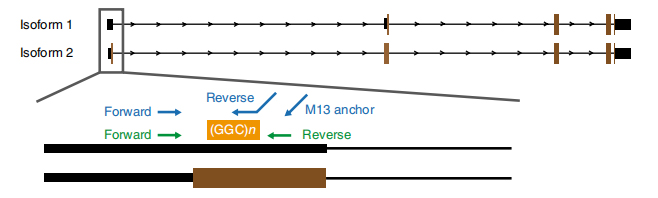
چترا 1. بیماری سے منسلک دوبارہ توسیع NOTCH2NLC isoforms کے exon 1 پر شناخت کی گئی ہے۔
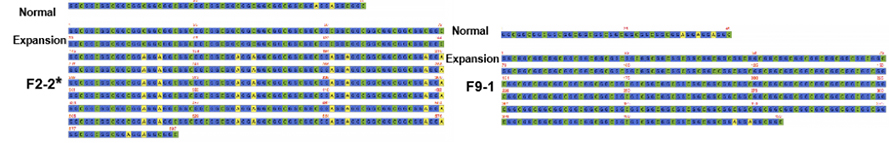
شکل 2. NPTCH2NLC کے متفقہ سلسلے NIID مریضوں میں (*) کے ساتھ یا کمزوری غالب فینوٹائپ کے بغیر دہرائے جاتے ہیں۔
NOTCH2NL جین انسانی مخصوص جینز ہیں، جن کے بارے میں خیال کیا جاتا ہے کہ وہ انسانی دماغی ارتقاء اور اعصابی امراض میں اہم کردار ادا کرتے ہیں۔تاہم، تین NOTCH2 متعلقہ جین (NOTCH2NLA، NOTCH2NLB اور NOTCH2NLC) کے ساتھ >99.1% تسلسل شناخت کے ساتھ تازہ ترین انسانی جینوم اسمبلی تک حل نہیں کیا گیا۔نینو پور پلیٹ فارم پر ترکیب سے پاک اور طویل پڑھنے والی ترتیب نے اعلی مماثلت والے علاقوں کو حل کرنے اور (GGC)n کو 100% GC سے بھرپور کے ساتھ دہرانے میں قابل ذکر فوائد دکھائے ہیں۔
NOTCH2NLC میں GGC کی دوبارہ توسیع
T2 متاثرہ اور 2 غیر متاثرہ اراکین پر ranscriptome کی ترتیب پر کارروائی کی گئی۔NOTCH2NL پیرالاگس کے پہلے ایکسونز کے اپ اسٹریم میں حس اور اینٹی سینس اسٹرینڈز پر معمول کے مطابق پڑھنے کی گہرائی کا حساب لگایا گیا تھا۔غیر معمولی اینٹی سینس ٹرانسکرپٹس صرف متاثرہ صورتوں میں پائی گئیں، جو شروع میں یا دوبارہ پھیلنے والے علاقے کے اندر بیٹھتی ہیں (شکل 3 میں F1-14 اور F1-16 میں جامنی چوٹیاں۔)اس کے علاوہ، 54 ڈی ای جی کی نشاندہی کی گئی تھی اور سبھی کو نیورونل فنکشنز سے متعلق GO اور MPO کی شرائط میں افزودہ کیا گیا تھا۔
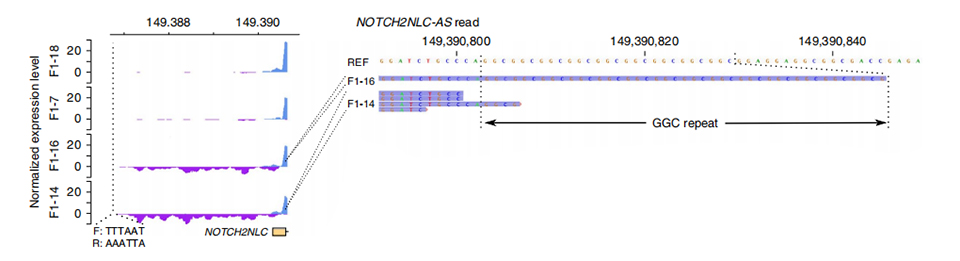
تصویر 3. غیر متاثرہ (اوپر) اور متاثرہ (نیچے) معاملات میں NOTCH2NLC کے پہلے ایکسون کے اوپر کی طرف معمول کے مطابق پڑھنے کی گہرائی۔
ٹیکنالوجی
Oxford Nanopore Teghnologies (ONT)
Nانوپور سیکوینسنگ خود کو دوسرے سیکوینسنگ پلیٹ فارمز سے ممتاز کرتی ہے، اس میں نیوکلیوٹائڈس کو بغیر ڈی این اے کی ترکیب کے عمل کے براہ راست پڑھا جاتا ہے۔جیسا کہ ایک ہی اسٹرینڈ ڈی این اے نینو سائز کے پروٹین پور (نینو پور) سے گزرتا ہے، مختلف نیوکلیوٹائڈز مختلف آئنک کرنٹ پیدا کرتے ہیں، جنہیں پکڑ کر بیس کی ترتیب میں منتقل کیا جا سکتا ہے۔او این ٹی سیکوینسنگ پلیٹ فارم خود ڈی این اے ریڈنگ کی لمبائی پر واضح تکنیکی حد نہیں دکھاتا ہے۔لہذا، الٹرا لانگ ریڈز (ULRs) اعلیٰ معیار کے جینوم اسمبلی کے لیے دستیاب ہیں۔مزید برآں، یہ انتہائی لمبی ریڈز، جو کہ پیچیدہ ترتیب کی خصوصیات یا ساختی تغیرات کو عبور کرنے کے لیے کافی لمبی ہیں، یہاں مختصر پڑھنے کی ترتیب کی حدود کو دور کرنے میں مدد کرتی ہیں۔

نینو پور کی ترتیب
ساخت میں تغیر (SV) کی شناخت
Sترکیب سے پاک ترتیب نے ٹیمپلیٹ پر ڈی این اے میتھیلیشن کی معلومات کو بڑی حد تک محفوظ کیا ہے۔میتھلیٹڈ اے، ٹی، سی اور جی غیر میتھلیٹڈ سے الگ آئنک کرنٹ پیدا کرتے ہیں، جنہیں پلیٹ فارم سے براہ راست پڑھا جا سکتا ہے۔نینو پور کی ترتیب سنگل نیوکلیوٹائڈ ریزولوشن پر 5mC اور 6mA دونوں کی مکمل جینوم پروفائلنگ کو تقویت دیتی ہے۔
حوالہ
جون سون، وغیرہ۔alطویل پڑھنے والی ترتیب نیورونل انٹرا نیوکلیئر انکلوژن بیماری سے وابستہ NOTCH2NLC میں GGC کی دوبارہ توسیع کی نشاندہی کرتی ہے۔نیچر جینیٹکس (2019)
ٹیک اور ہائی لائٹس جس کا مقصد مختلف تحقیقی میدانوں میں مختلف ہائی تھرو پٹ سیکوینسنگ ٹیکنالوجیز کے حالیہ کامیاب اطلاق کے ساتھ ساتھ تجرباتی ڈیزائن اور ڈیٹا مائننگ میں شاندار خیالات کا اشتراک کرنا ہے۔
پوسٹ ٹائم: جنوری 06-2022

