Повне секвенування транскриптомів – Illumina
особливості
● Подвійна бібліотека для секвенування повного транскриптому: виснаження рРНК з подальшим приготуванням бібліотеки PE150 і вибором розміру з наступним приготуванням бібліотеки SE50
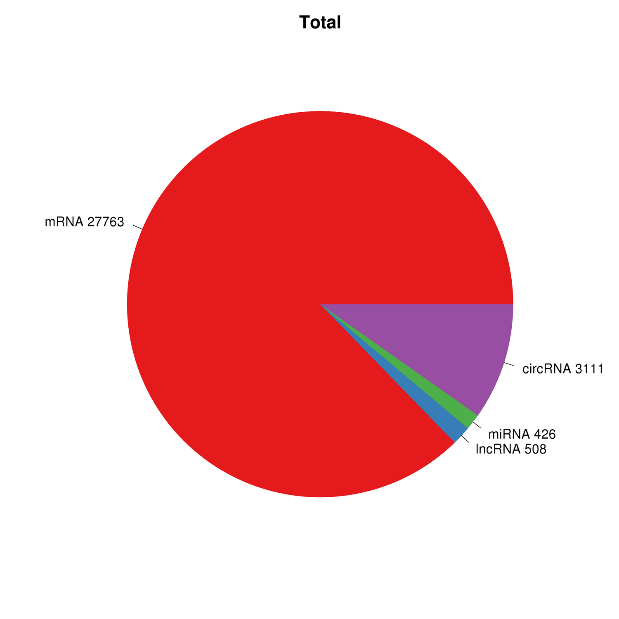
● Повний біоінформаційний аналіз мРНК, lncRNA, circRNA та miRNA в окремих біоінформаційних звітах
● Спільний аналіз усієї експресії РНК у комбінованому звіті, включаючи аналіз мереж ceRNA.
Переваги сервісу
●Поглиблений аналіз регуляторних мереж: мережевий аналіз ceRNA можливий завдяки спільному секвенуванню мРНК, lncRNA, circRNA та miRNA і вичерпному біоінформаційному робочому процесу.
●Вичерпна анотація: ми використовуємо численні бази даних для функціонального анотування диференціально експресованих генів (DEG) і виконання відповідного аналізу збагачення, надаючи розуміння клітинних і молекулярних процесів, що лежать в основі відповіді транскриптома.
●Великий досвід: завдяки досвіду успішного закриття понад 2000 цілих транскриптомних проектів у різних дослідницьких областях наша команда вносить багатий досвід у кожен проект.
●Суворий контроль якості: ми впроваджуємо основні контрольні точки на всіх етапах, від підготовки зразків і бібліотеки до секвенування та біоінформатики.Такий ретельний моніторинг забезпечує стабільно високу якість результатів.
● Вичерпна анотація: ми використовуємо численні бази даних для функціонального анотування диференціально експресованих генів (DEG) і виконання відповідного аналізу збагачення, надаючи розуміння клітинних і молекулярних процесів, що лежать в основі відповіді транскриптома.
●Післяпродажна підтримка: Наше зобов’язання поширюється на період після завершення проекту з 3-місячним періодом післяпродажного обслуговування.Протягом цього часу ми пропонуємо подальше спостереження за проектом, допомогу з усунення несправностей і сеанси запитань і відповідей, щоб відповісти на будь-які запитання, пов’язані з результатами
Вимоги до зразків і доставка
| Бібліотека | Стратегія секвенування | Рекомендовані дані | Контроль якості |
| виснажена рРНК | Illumina PE150 | 16 Гб | Q30≥85% |
| Вибраний розмір | Illumina SE50 | 10-20 млн читань |
Вимоги до зразків:
Нуклеотиди:
| Конц. (нг/мкл) | Кількість (мкг) | Чистота | Цілісність |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 На гелі виявлено обмежене забруднення білком або ДНК або його відсутність. | Рослини: RIN≥6,5 Тварина: RIN≥7,0 5,0≥28S/18S≥1,0; обмежене підвищення або відсутність базової лінії |
Рекомендована доставка зразків
Контейнер:
Центрифужна пробірка на 2 мл (фольга не рекомендована)
Зразок маркування: Група + копія, наприклад, A1, A2, A3;B1, B2, B3... ...
Відвантаження:
1. Сухий лід: Зразки потрібно упакувати в мішки та закопати в сухий лід.
2. РНК-стабільні пробірки: зразки РНК можна висушити в пробірці для стабілізації РНК (наприклад, RNAstable®) і відправити при кімнатній температурі.
Потік роботи служби

Дизайн експерименту

Доставка зразків

екстракція РНК

Будівництво бібліотеки

Секвенування

Аналіз даних

Післяпродажне обслуговування
Біоінформатика
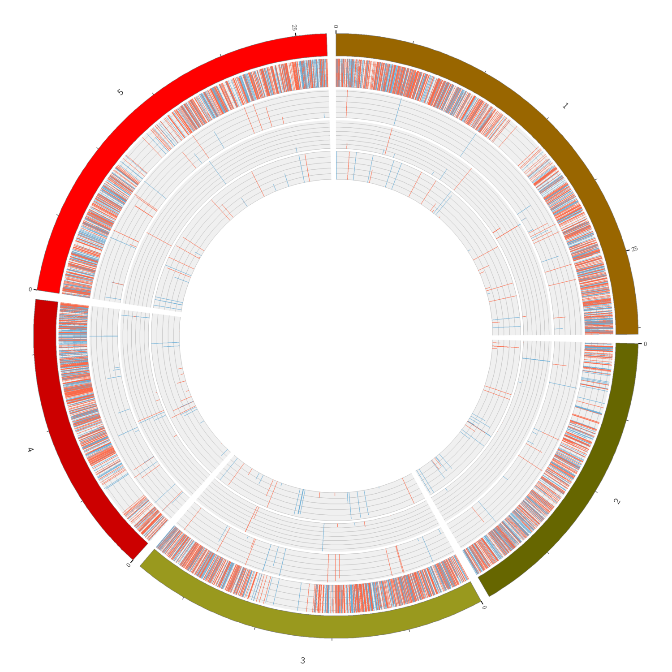
Огляд експресії РНК
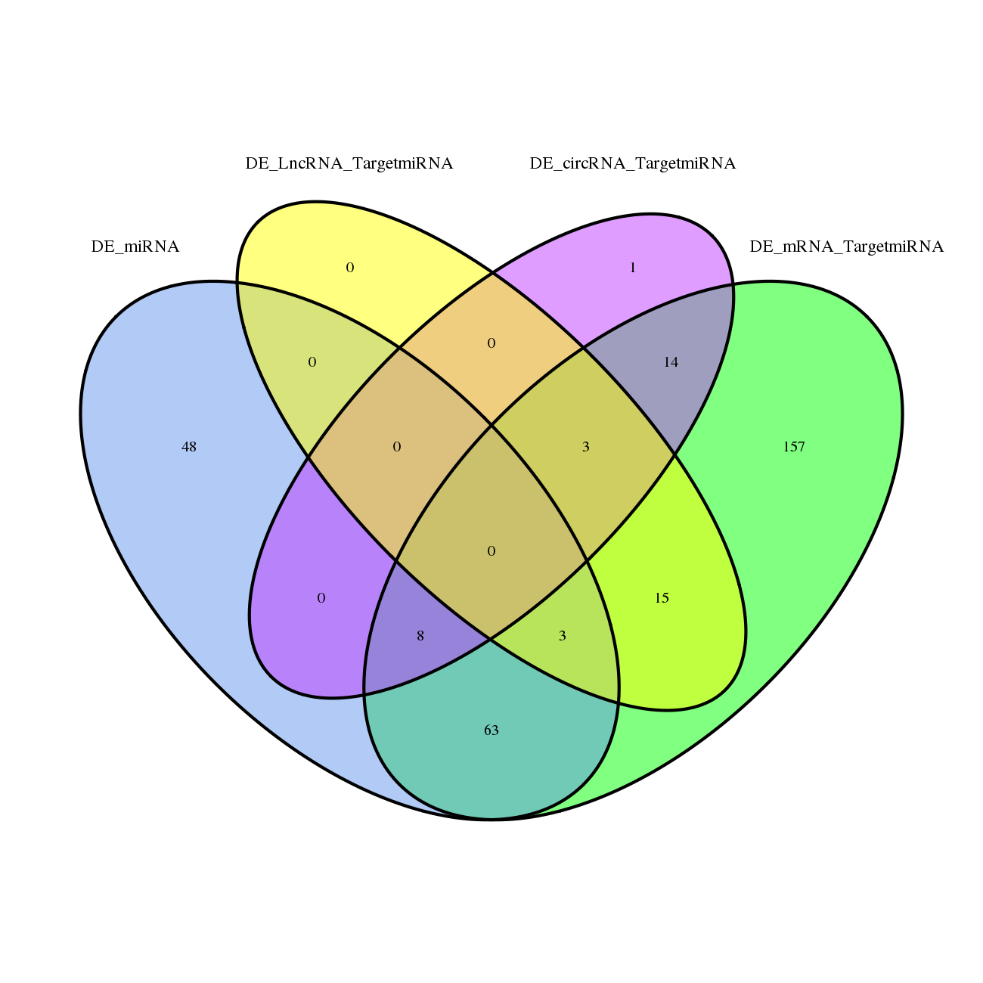
Гени з диференціальною експресією
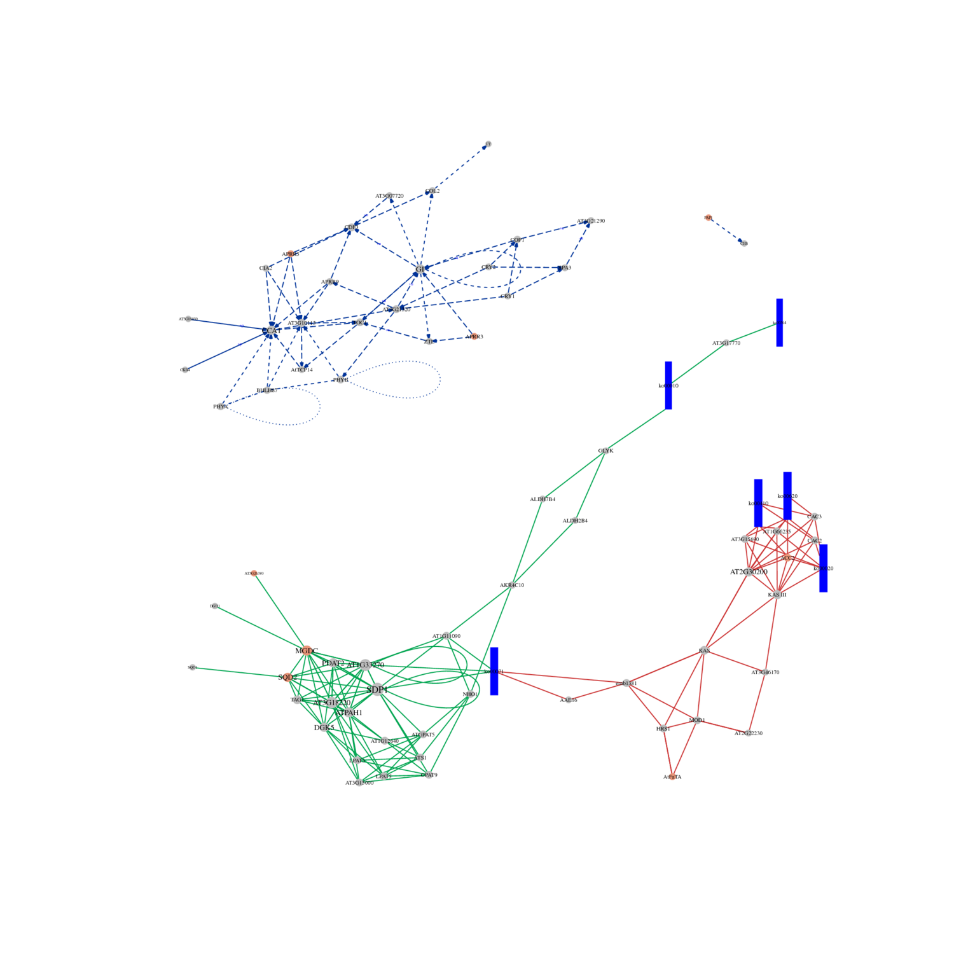
аналіз цеРНК
Ознайомтеся з досягненнями в дослідженнях, які сприяють послуги секвенування повних транскриптомів BMKGene, завдяки підібраній колекції публікацій.
Dai, Y. та ін.(2022) «Комплексні профілі експресії мРНК, lncRNA та мікроРНК при хворобі Кашина-Бека, ідентифіковані за допомогою РНК-секвенування», Molecular Omics, 18(2), стор. 154–166.doi: 10.1039/D1MO00370D.
Лю, Н. нан та ін.(2022) «Аналіз повної довжини транскриптомів холодостійкості Apis cerana в горі Чанбай під час періоду зимівлі». Gene, 830, стор. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ та ін.(2022) «Пріоритезація на основі інтеграції Multi-Omics конкуруючих ендогенних мереж регуляції РНК у дрібноклітинному раку легенів: молекулярні характеристики та препарати-кандидати», Frontiers in Oncology, 12, стор.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. та ін.(2022) «Інтегрований аналіз профілів експресії lncRNA/circRNA-miRNA-mRNA відкриває нове розуміння потенційних механізмів у відповідь на нематоди кореневих вузлів в арахісі», BMC Genomics, 23(1), стор. 1–12.doi: 10.1186/S12864-022-08470-3/FIGURES/7.
Ян, З. та ін.(2022) «Секвенування повної транскриптомної РНК висвітлює молекулярні механізми, пов’язані з підтримкою якості післязбирального врожаю брокколі за допомогою червоного світлодіодного опромінення», Postharvest Biology and Technology, 188, с.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.