TRANSKRİPTOMİKLER
doğa
İLETİŞİM
Kronik lenfositik lösemide SF3B1 mutasyonunun tam uzunlukta transkript karakterizasyonu, tutulan intronların aşağı regülasyonunu ortaya koyuyor
Tam uzunlukta transkriptler|Nanogözenek dizilimi|Alternatif izoform analizi
Arka plan
SSF3B1 birleştirme faktöründeki omatik mutasyonların, kronik lenfositik lösemi (CLL), uveal melanom, meme kanseri vb. dahil olmak üzere çeşitli kanserlerle ilişkili olduğu geniş çapta rapor edilmiştir. Ayrıca, kısa okumalı transkriptomik çalışmalar, SF3B1 mutasyonlarının neden olduğu anormal birleştirme modellerini ortaya çıkarmıştır.Bununla birlikte, bu alternatif birleştirme modelleri üzerindeki çalışmalar uzun süredir olay düzeyiyle sınırlıydı ve kısa okunan birleştirilmiş transkriptlerin sınırlılığı nedeniyle izoform düzeyinde bilgi eksikliği vardı.Burada, AS izoformlarının araştırılmasını güçlendiren tam uzunlukta transkriptler oluşturmak için nanopore sıralama platformu tanıtıldı.
Deneysel tasarım
Deneyler
Gruplandırma:1. CLL-SF3B1(WT) 2. CLL-SF3B1(K700E mutasyonu);3. Normal B hücreleri
Sıralama stratejisi:MinION 2D kitaplık sıralaması, PromethION 1D kitaplık sıralaması;Aynı örneklerden kısa okuma verileri
Sıralama platformu:ONT Minion;ONT Promethion;
Biyoinformatik Analiz
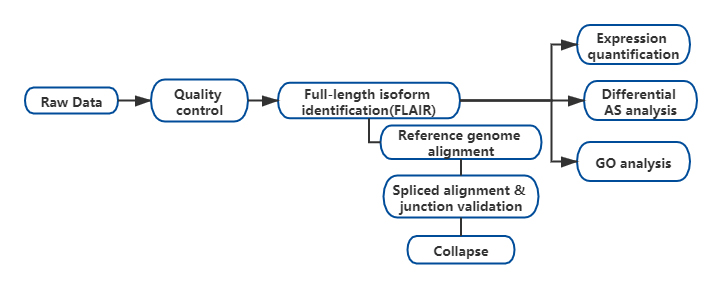
Sonuçlar
A6 CLL örneğinden ve 3 B hücresinden toplam 257 milyon okuma üretildi.Bu okumaların ortalama %30,5'i tam uzunlukta transkript olarak tanımlandı.
FRNA'nın tam uzunlukta alternatif izoform analizi (FLAIR), bir dizi yüksek güvenirli izoform oluşturmak için geliştirildi.FLAIR şu şekilde özetlenebilir:
Nanopore hizalamayı okur: referans genomuna dayalı olarak genel transkript yapısını tanımlar;
Skat bağlantı düzeltmesi: açıklamalı intronlardan, kısa okunan verilerden veya her ikisinden gelen ekleme bölgesi ile dizi hatalarını (kırmızı) düzeltin;
Collapse: ek bağlantı zincirlerine (ilk geçiş kümesi) dayalı temsili izoformları özetleyin.Destekleyici okuma sayısına göre yüksek güvenilirliğe sahip isofrom'u seçin (Eşik: 3).
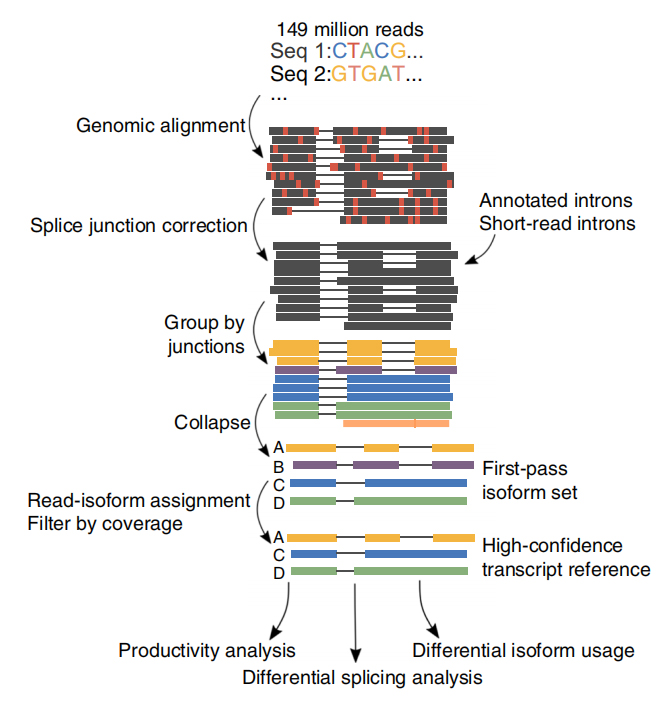
Şekil 1. CLL'de SF3B1 mutasyonuyla ilişkili tam uzunluktaki izoformları tanımlamak için FLAIR analizi
FLAIR, %90'ı yeni izoformlar olan 326.699 yüksek güvenliğe sahip eklenmiş izoform tanımladı.Bu açıklanmamış izoformların çoğunun, bilinen ekleme bağlantılarının (142.971) yeni kombinasyonları olduğu, geri kalan yeni izoformların ise ya tutulan intron (21.700) ya da yeni ekson (3594) içerdiği bulundu.
LOng-read dizileri, mutant SF3B1-K700E ile değiştirilmiş ekleme bölgelerinin izoform düzeyinde tanımlanmasını güçlendirir.35 alternatif 3'SS ve 10 alternatif 5'SS'nin, SF3B1-K700E ve SF3B1-WT arasında önemli ölçüde diferansiyel olarak eklendiği bulundu.35 değişikliğin 33'ü uzun okuma dizileri tarafından yeni keşfedildi.Nanopore verilerinde, SF3B1-K700E ile değiştirilmiş 3'SS'ler ile kanonik konum zirveleri arasındaki mesafenin dağılımı -20 bp civarındadır; bu, CLL kısa okuma dizilerinde rapor edilene benzer şekilde bir kontrol dağılımından önemli ölçüde farklıdır.ERGIC3 geninin izoformları analiz edildi; burada proksimal birleşme bölgesini içeren yeni bir izoformun SF3B1-K700E'de daha bol olduğu bulundu.Hem proksimal hem de distal 3'SS, çoklu izoformlar üreten farklı AS modelleri ile ilişkilendirildi.
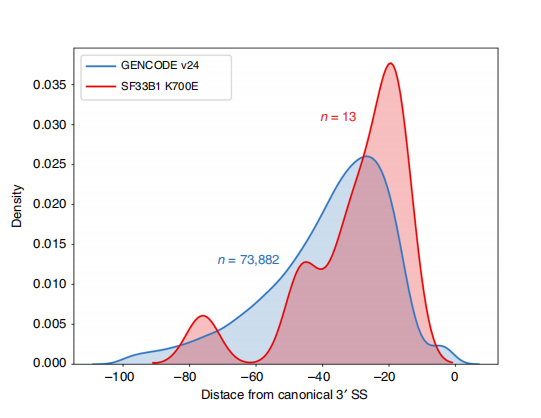
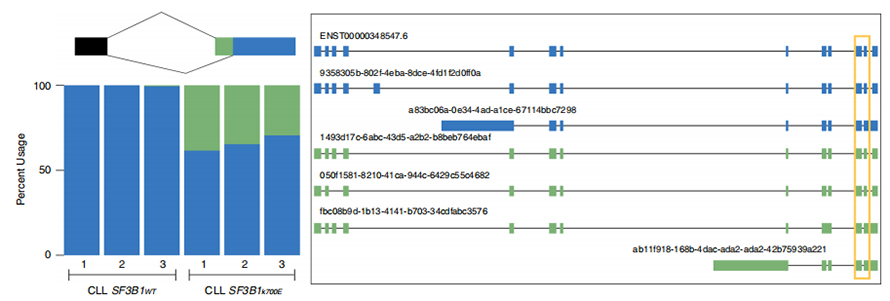
Şekil 2. Nano gözenek sıralama verileriyle tanımlanan alternatif 3' birleştirme desenleri
IR olay kullanım analizi, IR tanımlama ve ölçümüne olan güven nedeniyle kısa okuma tabanlı analizde uzun süredir sınırlıdır.SF3B1-K700E ve SF3B1-WT'de IR izoformlarının ekspresyonu, nano-gözenek dizilerine dayalı olarak ölçüldü ve SF3B1-K700E'de IR izoformlarının küresel bir aşağı regülasyonunu ortaya çıkardı.
Şekil 4. Üç tarım sistemi (A ve B) genelinde tarım yoğunluğu ve ağ bağlantısı;Rastgele orman analizi(C) ve tarımsal yoğunluk ile AMF kolonizasyonu arasındaki ilişki (D)
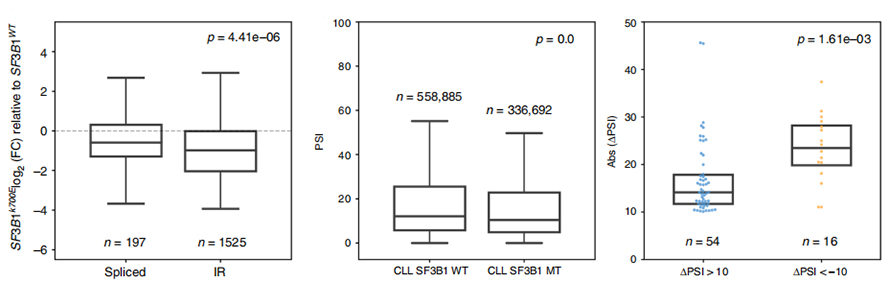
Şekil 3. İntron kiralama olayları CLL SF3B1-K700E'de daha güçlü bir şekilde aşağı doğru düzenlenmiştir.
Teknoloji
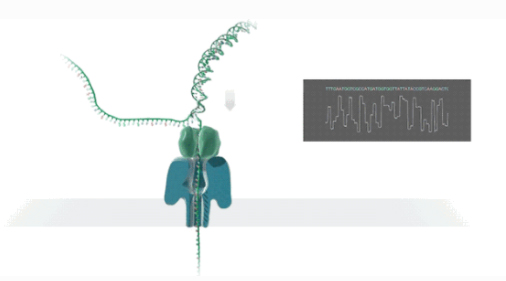
Nanopore Uzun Okumalı Sıralama
Nanopore sıralama, tek moleküllü gerçek zamanlı elektrik sinyali sıralama teknolojisidir.
DÇift sarmallı DNA veya RNA, biyofilme gömülü nano gözenekli proteine bağlanacak ve motor proteinin öncülüğünde çözülecektir.
DNA/RNA iplikçikleri, voltaj farkının etkisi altında belirli bir oranda nanopore kanal proteininden geçer.
MOleküller kimyasal yapılarına göre farklı elektrik sinyalleri üretirler.
RDizilerin gerçek zamanlı tespiti baz araması ile gerçekleştirilir.
Tam uzunlukta transkriptom sıralamasının performansı
√ Veri Doygunluğu
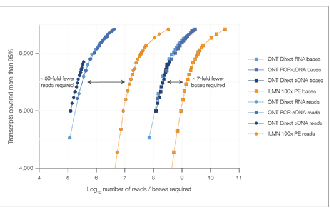
Karşılaştırılabilir veri doygunluğuna ulaşmak için 7 kat daha az okuma gerekir.
√ Transkript Yapısının Belirlenmesi
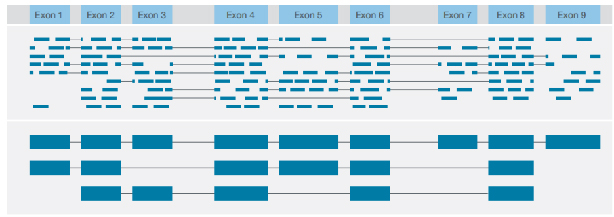
Her transkriptin tam uzunlukta okunmasıyla çeşitli yapısal varyantların tanımlanması
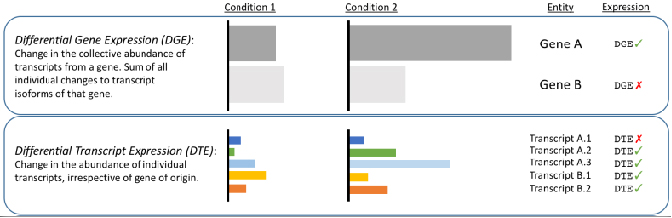
√ Transkript düzeyinde diferansiyel analiz -Kısa okumalarla gizlenen değişiklikleri ortaya çıkarın
Referans
Tang AD, Soulette CM, Baren MJV ve diğerleri.Kronik lenfositik lösemide SF3B1 mutasyonunun tam uzunlukta transkript karakterizasyonu, tutulan intronların[J] aşağı regülasyonunu ortaya koymaktadır.Doğa İletişimi.
Teknoloji ve Öne Çıkanlar farklı yüksek verimli sıralama teknolojilerinin çeşitli araştırma alanlarındaki en son başarılı uygulamalarının yanı sıra deneysel tasarım ve veri madenciliğindeki parlak fikirleri paylaşmayı amaçlamaktadır.
Gönderim zamanı: Ocak-08-2022

