การจัดลำดับการถอดเสียงทั้งหมด - อิลลูมินา
คุณสมบัติ
● ไลบรารีคู่เพื่อจัดลำดับทรานสคริปโตมที่สมบูรณ์: การสูญเสีย rRNA ตามด้วยการเตรียมไลบรารี PE150 และการเลือกขนาด ตามด้วยการเตรียมไลบรารี SE50
● วิเคราะห์ชีวสารสนเทศสมบูรณ์ของ mRNA, lncRNA, circRNA และ miRNA ในรายงานชีวสารสนเทศที่แยกจากกัน
● การวิเคราะห์ร่วมของการแสดงออก RNA ทั้งหมดในรายงานแบบรวม รวมถึงการวิเคราะห์เครือข่าย ceRNA
ข้อดีของการบริการ
●การวิเคราะห์เชิงลึกของเครือข่ายการกำกับดูแล: การวิเคราะห์เครือข่าย ceRNA เปิดใช้งานได้โดยการจัดลำดับร่วมของ mRNA, lncRNA, circRNA และ miRNA และโดยขั้นตอนการทำงานทางชีวสารสนเทศที่ละเอียดถี่ถ้วน
●คำอธิบายประกอบที่ครอบคลุม: เราใช้ฐานข้อมูลหลายฐานข้อมูลเพื่ออธิบายประกอบยีนที่แสดงออกแตกต่างกัน (DEG) และดำเนินการวิเคราะห์การเพิ่มคุณค่าที่สอดคล้องกัน โดยให้ข้อมูลเชิงลึกเกี่ยวกับกระบวนการระดับเซลล์และโมเลกุลที่เป็นรากฐานของการตอบสนองของการถอดเสียง
●ความเชี่ยวชาญที่กว้างขวาง: ด้วยประวัติความสำเร็จในการปิดโครงการถอดเสียงทั้งหมดมากกว่า 2,000 โครงการในขอบเขตการวิจัยต่างๆ ทีมงานของเรานำประสบการณ์มากมายมาสู่ทุกโครงการ
●การควบคุมคุณภาพอย่างเข้มงวด: เราใช้จุดควบคุมหลักในทุกขั้นตอน ตั้งแต่การเตรียมตัวอย่างและห้องสมุด ไปจนถึงการจัดลำดับและชีวสารสนเทศศาสตร์การตรวจสอบอย่างพิถีพิถันนี้ช่วยให้มั่นใจได้ถึงผลลัพธ์คุณภาพสูงอย่างสม่ำเสมอ
● คำอธิบายประกอบที่ครอบคลุม: เราใช้ฐานข้อมูลหลายฐานข้อมูลเพื่ออธิบายประกอบยีนที่แสดงออกแตกต่างกัน (DEG) และดำเนินการวิเคราะห์การเพิ่มคุณค่าที่สอดคล้องกัน โดยให้ข้อมูลเชิงลึกเกี่ยวกับกระบวนการระดับเซลล์และโมเลกุลที่เป็นรากฐานของการตอบสนองของการถอดเสียง
●การสนับสนุนหลังการขาย: ความมุ่งมั่นของเราขยายไปไกลกว่าการเสร็จสิ้นโครงการด้วยระยะเวลาบริการหลังการขาย 3 เดือนในช่วงเวลานี้ เราเสนอการติดตามผลโครงการ ความช่วยเหลือในการแก้ไขปัญหา และเซสชันถามและตอบเพื่อตอบข้อสงสัยใด ๆ ที่เกี่ยวข้องกับผลลัพธ์
ข้อกำหนดตัวอย่างและการจัดส่ง
| ห้องสมุด | กลยุทธ์การจัดลำดับ | ข้อมูลที่แนะนำ | ควบคุมคุณภาพ |
| rRNA หมดลง | อิลลูมิน่า พีอี150 | 16 กิกะไบต์ | Q30≥85% |
| เลือกขนาดแล้ว | อิลลูมินา SE50 | อ่าน 10-20M |
ข้อกำหนดตัวอย่าง:
นิวคลีโอไทด์:
| ความเข้มข้น(ng/μl) | ปริมาณ (ไมโครกรัม) | ความบริสุทธิ์ | ความซื่อสัตย์ |
| ≥ 100 | ≥ 1 | OD260/280=1.7-2.5 โอดี260/230=0.5-2.5 มีการปนเปื้อนของโปรตีนหรือ DNA อย่างจำกัดหรือไม่มีเลยบนเจล | พืช: RIN≥6.5 สัตว์: RIN≥7.0 5.0≥28S/18S≥1.0; ระดับความสูงพื้นฐานที่จำกัดหรือไม่มีเลย |
การจัดส่งตัวอย่างที่แนะนำ
คอนเทนเนอร์:
หลอดหมุนเหวี่ยงขนาด 2 มล. (ไม่แนะนำให้ใช้ฟอยล์ดีบุก)
การติดฉลากตัวอย่าง: กลุ่ม+ทำซ้ำ เช่น A1, A2, A3;บี1 บี2 บี3......
การจัดส่ง:
1.น้ำแข็งแห้ง: ตัวอย่างจะต้องบรรจุในถุงและฝังในน้ำแข็งแห้ง
2.หลอด RNAstable: ตัวอย่าง RNA สามารถทำให้แห้งในหลอดรักษาเสถียรภาพ RNA (เช่น RNAstable®) และจัดส่งในอุณหภูมิห้อง
ขั้นตอนการทำงานบริการ

การออกแบบการทดลอง

การจัดส่งตัวอย่าง

การสกัดอาร์เอ็นเอ

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
ชีวสารสนเทศศาสตร์
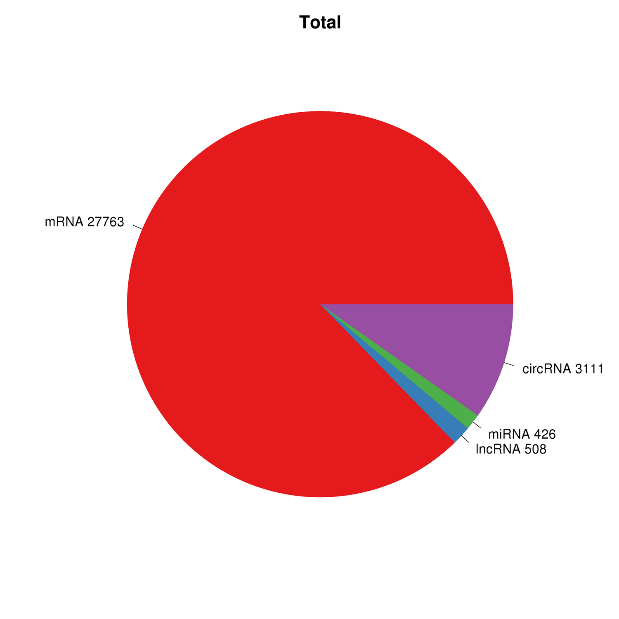
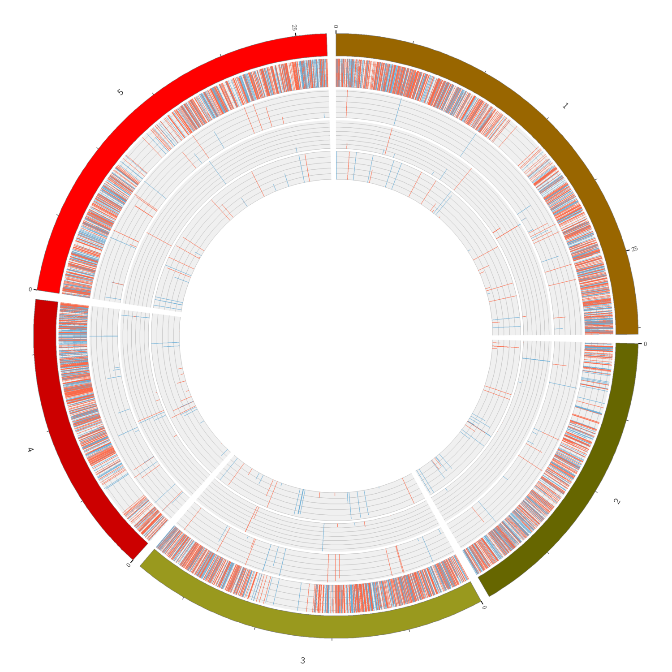
ภาพรวมการแสดงออกของ RNA
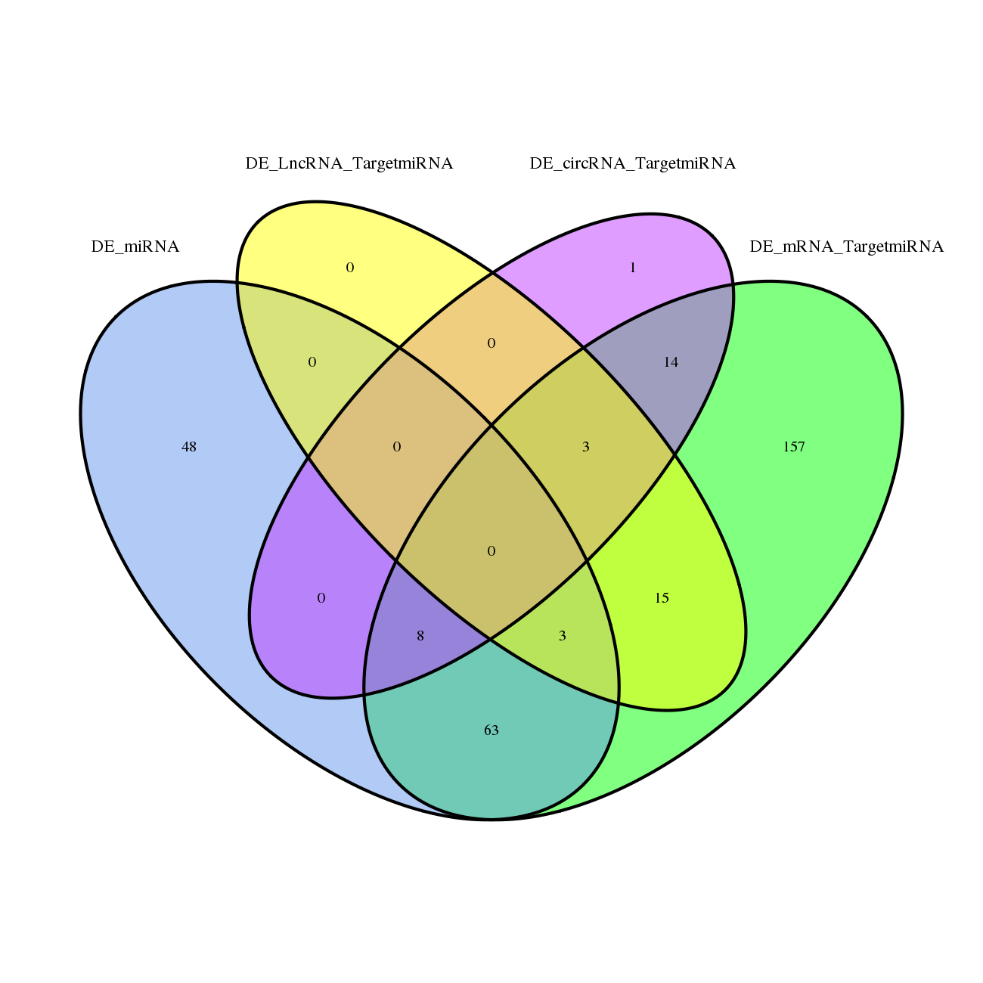
ยีนที่แสดงออกอย่างแตกต่าง
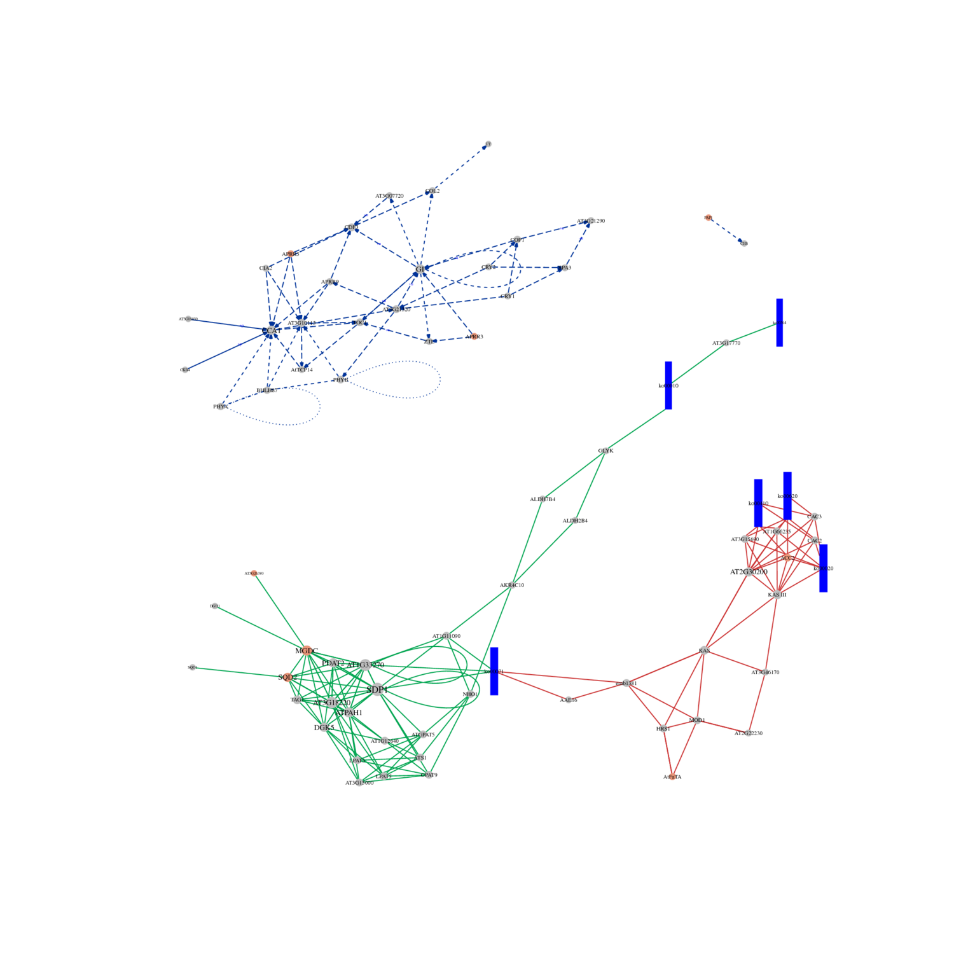
การวิเคราะห์ซีอาร์เอ็นเอ
สำรวจความก้าวหน้าทางการวิจัยที่อำนวยความสะดวกโดยบริการจัดลำดับการถอดเสียงทั้งหมดของ BMKGene ผ่านคอลเลคชันสิ่งพิมพ์ที่คัดสรรมาอย่างดี
Dai, Y. และคณะ(2022) 'โปรไฟล์การแสดงออกที่ครอบคลุมของ mRNAs, lncRNAs และ miRNAs ในโรค Kashin-Beck ที่ระบุโดยลำดับ RNA', Molecular Omics, 18(2), หน้า 154–166ดอย: 10.1039/D1MO00370D.
หลิว น. น่าน และคณะ(2022) 'การวิเคราะห์ทรานสคริปต์โตมแบบเต็มความยาวของการต้านทานความเย็นของ Apis cerana ในภูเขาฉางไป๋ในช่วงฤดูหนาว', Gene, 830, หน้า 146503–146503ดอย: 10.1016/J.GENE.2022.146503.
วัง XJ และคณะ(2022) 'การจัดลำดับความสำคัญตามการบูรณาการหลาย Omics ของเครือข่ายการควบคุม RNA ภายนอกที่แข่งขันกันในมะเร็งปอดในเซลล์ขนาดเล็ก: ลักษณะทางโมเลกุลและผู้สมัครยา', พรมแดนในด้านเนื้องอกวิทยา, 12, p.904865. ดอย: 10.3389/FONC.2022.904865/BIBTEX.
ซู พี และคณะ(2022) 'การวิเคราะห์แบบบูรณาการของโปรไฟล์การแสดงออก lncRNA/circRNA-miRNA-mRNA เผยให้เห็นข้อมูลเชิงลึกใหม่เกี่ยวกับกลไกที่เป็นไปได้ในการตอบสนองต่อไส้เดือนฝอยรากปมในถั่วลิสง', BMC Genomics, 23(1), หน้า 1–12ดอย: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. และคณะ(2022) 'การจัดลำดับ RNA ทั้งยีนเน้นย้ำกลไกระดับโมเลกุลที่เกี่ยวข้องกับการรักษาคุณภาพหลังการเก็บเกี่ยวในบรอกโคลีโดยการฉายรังสี LED สีแดง', ชีววิทยาและเทคโนโลยีหลังการเก็บเกี่ยว, 188, p.111878. ดอย: 10.1016/J.POSTHARVBIO.2022.111878.