จีโนมิกส์ของมนุษย์
พันธุศาสตร์ธรรมชาติ
การจัดลำดับแบบอ่านยาวระบุการขยายตัวซ้ำของ GGC ใน NOTCH2NLC ที่เกี่ยวข้องกับโรคการรวมในสมองของเซลล์ประสาท
ลำดับ ONT |อิลลูมินา |ลำดับภายนอกทั้งหมด |CRISPR-Cas9 ONT ลำดับเป้าหมาย |RNA-seq |การเรียกเมทิลเลชั่น ONT 5mC
ไฮไลท์
1. โดยการวิเคราะห์การเชื่อมโยงในกลุ่ม NIID ขนาดใหญ่ มีการระบุบริเวณที่เชื่อมโยงกันสองแห่ง
2. การจัดลำดับการอ่านแบบยาวโดยใช้ ONT และการเพิ่มคุณค่าแบบสื่อกลาง Cas-9 การจัดลำดับ ONT ค้นพบสาเหตุทางพันธุกรรมที่เป็นไปได้ของ NIID, GGC การขยายซ้ำใน 5 ′UTR ของ NOTCH2NLCการศึกษานี้รายงานการขยายตัวซ้ำในยีนเฉพาะของมนุษย์เป็นครั้งแรกที่วิวัฒนาการผ่านการทำซ้ำแบบปล้อง
3. การจัดลำดับ RNA เปิดเผยการถอดเสียง antisense ที่ผิดปกติในจุดเริ่มต้นหรือภายในขอบเขตการขยายซ้ำของ GGC ใน NOTCH2NLC
พื้นหลัง
Nโรคการรวมตัวของนิวเคลียร์ในยูโรนัล (NIID) เป็นโรคที่เกิดจากความเสื่อมของระบบประสาทที่ลุกลามและเป็นอันตรายถึงชีวิต ซึ่งมีลักษณะเฉพาะคือการมีอยู่ของการรวมตัวของไฮยาลีนในนิวเคลียร์ eosinophilic ในระบบประสาทส่วนกลางและอุปกรณ์ต่อพ่วงอาการทางคลินิกที่มีความแปรปรวนสูงทำให้เกิดความยากในการวินิจฉัยจนกระทั่งมีการตัดชิ้นเนื้อผิวหนังอย่างไรก็ตาม วิธีการทางจุลพยาธิวิทยายังคงประสบปัญหาจากการวินิจฉัยผิดพลาด ซึ่งเรียกร้องให้มีความเข้าใจทางพันธุกรรมของ NIID
ความสำเร็จ
การวิเคราะห์การเชื่อมโยง
Sการจัดลำดับแบบอ่านง่ายตามการจัดลำดับจีโนมทั้งหมด (WGS) และการจัดลำดับภายนอกทั้งหมด (WES) ได้ดำเนินการในตระกูล NIID ขนาดใหญ่ (ได้รับผลกระทบ 13 รายและสมาชิกที่ไม่ได้รับผลกระทบ 7 ราย)การวิเคราะห์การเชื่อมโยงบน SNP ที่ดึงมาจากข้อมูลเหล่านี้เผยให้เห็นเพียงสองขอบเขตที่เชื่อมโยง: ขอบเขต 3.5 Mb ที่ 1p36.31-p36.22 (LOD สูงสุด=2.32) และขอบเขต 58.1 Mb ที่ 1p22.1-q21.3 (LOD สูงสุด: 4.21 ).อย่างไรก็ตาม ไม่มีการระบุ SNP หรือ CNV ที่ทำให้เกิดโรคในภูมิภาคที่เชื่อมโยงเหล่านี้
GGC ขยายซ้ำใน NOTCH2NLC
Nการจัดลำดับแบบ anopore ได้รับการประมวลผลใน 13 คนที่ได้รับผลกระทบและสมาชิกที่ไม่ได้รับผลกระทบ 4 คนจาก 8 ตระกูล (สมาชิกที่ได้รับผลกระทบอีกคนถูกจัดลำดับโดยแพลตฟอร์มการจัดลำดับการอ่านแบบยาวของ Pacbio)ข้อมูลที่อ่านแบบยาวเผยให้เห็นโรคที่เกี่ยวข้องกับการขยาย GGC ซ้ำใน 5 ′UTR ของการทำแผนที่ยีน NOTCH2NLC ไปยังภูมิภาคที่เชื่อมโยง 58.1 Mb (รูปที่ 1)การขยายตัวซ้ำเหล่านี้ยังถูกระบุในกรณี NIID ประปรายทั้ง 40 กรณีที่ทดสอบโดย RP-PCR
Cการจัดลำดับเป้าหมายที่เป็นสื่อกลาง as-9 บนแพลตฟอร์ม nanopore ถูกนำมาใช้เพื่อให้เกิดความครอบคลุมการอ่านที่สูงขึ้นในการทำซ้ำ NOTCH2NLC (100 X-1,795 X)ลำดับฉันทามติเหล่านี้ตกลงกันได้ดีกับการค้นพบครั้งก่อนเกี่ยวกับการขยาย GGC ซ้ำยิ่งไปกว่านั้น {(GGA) n (GGC) n} n การทำซ้ำถูกระบุว่าเป็นเครื่องหมายทางพันธุกรรมที่เป็นไปได้สำหรับฟีโนไทป์ที่มีความอ่อนแอ (รูปที่ 2)
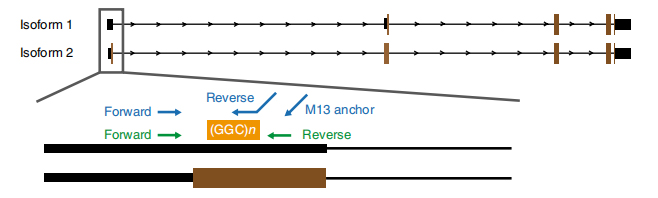
รูปที่ 1 การขยายตัวซ้ำที่เกี่ยวข้องกับโรคที่ระบุใน exon 1 ของไอโซฟอร์ม NOTCH2NLC
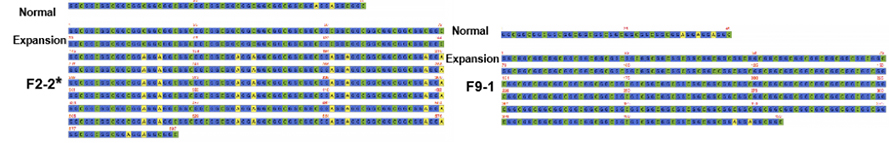
รูปที่ 2 ลำดับฉันทามติของการทำซ้ำ NPTCH2NLC ในผู้ป่วย NIID ที่มี (*) หรือไม่มีฟีโนไทป์ที่มีความอ่อนแอ
Nยีน OTCH2NL เป็นยีนเฉพาะของมนุษย์ ซึ่งเชื่อกันว่ามีบทบาทสำคัญในวิวัฒนาการของสมองของมนุษย์และโรคทางระบบประสาทอย่างไรก็ตาม ยีนที่เกี่ยวข้องกับ NOTCH2 สามยีน (NOTCH2NLA, NOTCH2NLB และ NOTCH2NLC) ที่มีความเป็นเอกลักษณ์ของลำดับ> 99.1% ไม่ได้รับการแก้ไขจนกว่าการประกอบจีโนมมนุษย์ล่าสุดการจัดลำดับที่ไม่มีการสังเคราะห์และอ่านได้นานบนแพลตฟอร์มนาโนพอร์ได้แสดงให้เห็นถึงข้อได้เปรียบที่โดดเด่นในการแก้ไขบริเวณที่มีความคล้ายคลึงกันสูงและ (GGC) n ซ้ำด้วย GC ที่อุดมด้วย 100%
GGC ขยายซ้ำใน NOTCH2NLC
Tการจัดลำดับ ranscriptome ได้รับการประมวลผลกับสมาชิกที่ได้รับผลกระทบ 2 รายและสมาชิกที่ไม่ได้รับผลกระทบ 2 รายความลึกในการอ่านที่ถูกทำให้เป็นมาตรฐานถูกคำนวณตามความรู้สึกและแอนตี้เซนส์ในต้นน้ำของ exons แรกของพาราล็อก NOTCH2NLการถอดเสียงต่อต้านการรับรู้ที่ผิดปกติพบเฉพาะในกรณีที่ได้รับผลกระทบ ซึ่งอยู่ที่จุดเริ่มต้นหรือภายในขอบเขตการขยายตัวซ้ำ (ยอดสีม่วงใน F1-14 และ F1-16 ในรูปที่ 3)นอกจากนี้ยังมีการระบุ 54 DEG และทั้งหมดได้รับการเสริมคุณค่าในเงื่อนไข GO และ MPO ที่เกี่ยวข้องกับการทำงานของเซลล์ประสาท
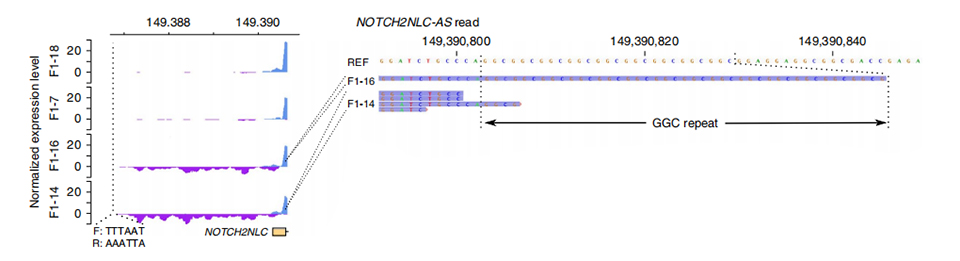
รูปที่ 3 ความลึกในการอ่านแบบปกติบนอัพสตรีมของ exon แรกของ NOTCH2NLC ในกรณีที่ไม่ได้รับผลกระทบ (ด้านบน) และได้รับผลกระทบ (ด้านล่าง)
เทคโนโลยี
เทคโนโลยี Oxford Nanopore (ONT)
Nการหาลำดับอะพอร์แตกต่างจากแพลตฟอร์มการหาลำดับอื่นๆ ตรงที่นิวคลีโอไทด์จะถูกอ่านโดยตรงโดยไม่ต้องใช้กระบวนการสังเคราะห์ DNAเมื่อ DNA สายเดี่ยวผ่านรูพรุนโปรตีนขนาดนาโน (นาโนพอร์) นิวคลีโอไทด์ที่แตกต่างกันจะสร้างกระแสไอออนิกที่แตกต่างกัน ซึ่งสามารถจับและถ่ายโอนไปยังลำดับเบสได้แพลตฟอร์มการจัดลำดับ ONT เองไม่ได้แสดงขีดจำกัดทางเทคนิคที่ชัดเจนเกี่ยวกับความยาวของการอ่าน DNAดังนั้นจึงมีการอ่านค่า Ultra-long (ULR) สำหรับการประกอบจีโนมคุณภาพสูงนอกจากนี้ การอ่านที่ยาวมากเหล่านี้ ซึ่งยาวพอที่จะข้ามคุณลักษณะลำดับที่ซับซ้อนหรือการแปรผันของโครงสร้าง ช่วยเอาชนะข้อจำกัดของการจัดลำดับการอ่านแบบสั้นที่นี่

ลำดับนาโนพอร์
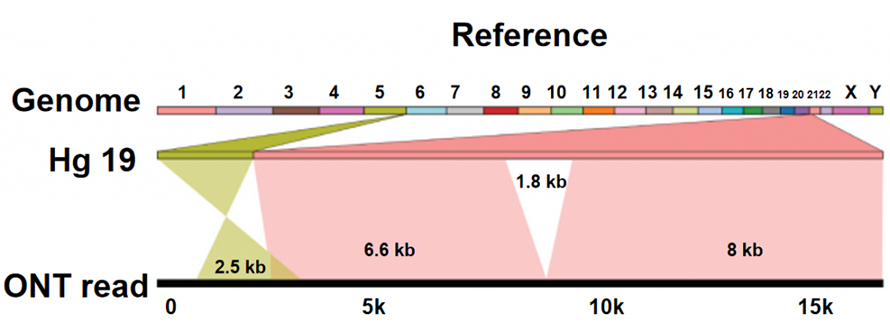
การระบุการเปลี่ยนแปลงโครงสร้าง (SV)
Sการจัดลำดับที่ไม่มีการสังเคราะห์จะรักษาข้อมูล DNA methylation ไว้ในเทมเพลตเป็นส่วนใหญ่เมทิลเลต A, T, C และ G สร้างกระแสไอออนิกที่แตกต่างจากกระแสที่ไม่มีเมทิลเลต ซึ่งสามารถอ่านได้โดยตรงจากแพลตฟอร์มการจัดลำดับนาโนพอร์ช่วยให้สามารถสร้างโปรไฟล์จีโนมทั้ง 5mC และ 6mA ที่ความละเอียดนิวคลีโอไทด์เดี่ยว
อ้างอิง
จุน โซน และ.อัลการจัดลำดับแบบอ่านยาวจะระบุการขยายตัวซ้ำของ GGC ใน NOTCH2NLC ที่เกี่ยวข้องกับโรคการรวมตัวของเซลล์ประสาทในสมองพันธุศาสตร์ธรรมชาติ (2019)
เทคโนโลยีและไฮไลท์ มุ่งหวังที่จะแบ่งปันการประยุกต์ใช้เทคโนโลยีการจัดลำดับความเร็วสูงต่างๆ ที่ประสบความสำเร็จล่าสุดในขอบเขตการวิจัยต่างๆ รวมถึงแนวคิดที่ยอดเยี่ยมในการออกแบบการทดลองและการขุดข้อมูล
เวลาโพสต์: Jan-06-2022

