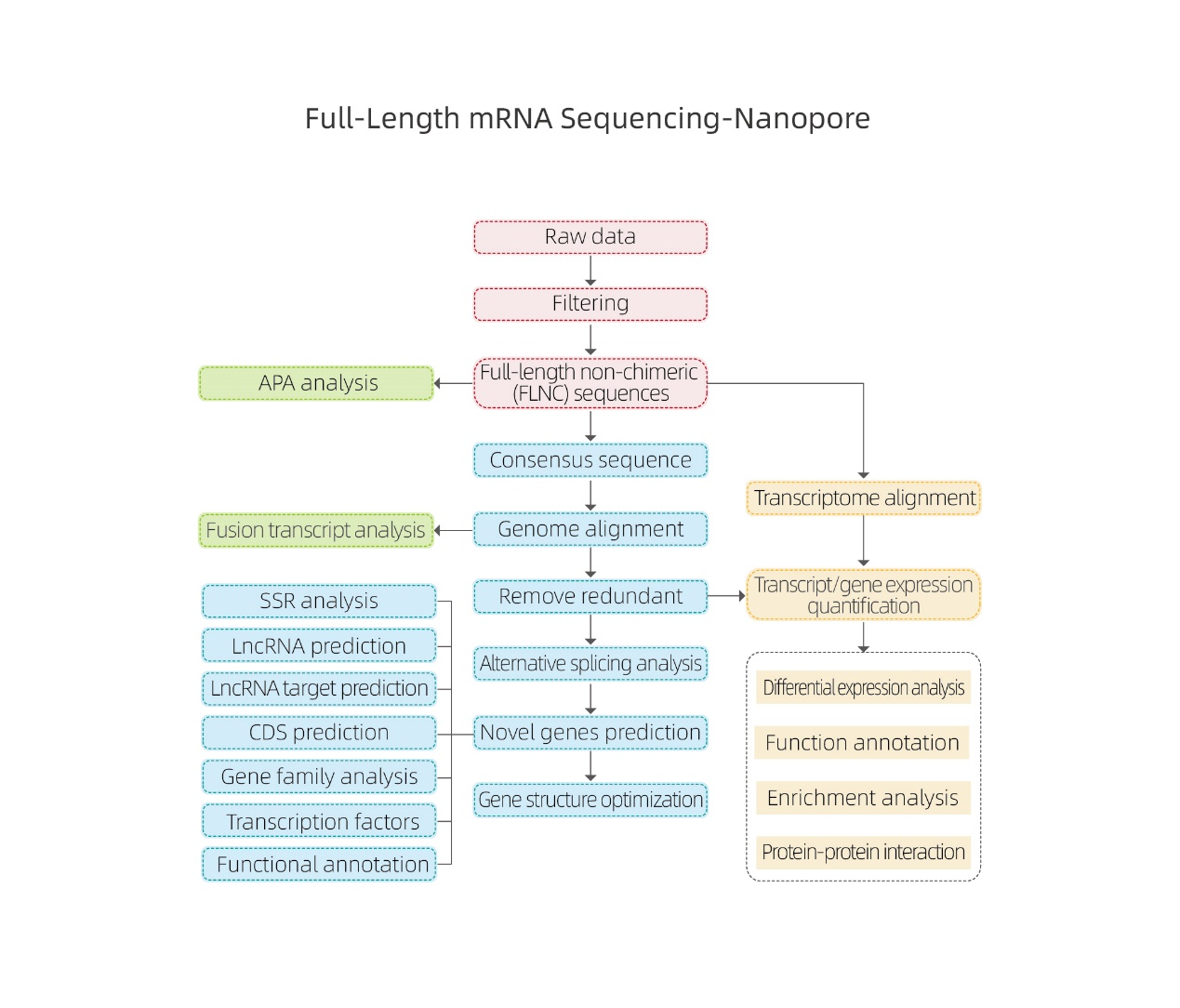
ลำดับ mRNA ความยาวเต็ม-นาโนพอร์
ข้อดีของการบริการ
● อคติในลำดับต่ำ
● เผยโมเลกุล cDNA แบบเต็มความยาว
● ต้องใช้ข้อมูลน้อยลงเพื่อให้ครอบคลุมการถอดเสียงจำนวนเท่าเดิม
● การระบุไอโซฟอร์มหลายแบบต่อยีน
● ปริมาณการแสดงออกในระดับไอโซฟอร์ม
ข้อมูลจำเพาะของบริการ
| ห้องสมุด | แพลตฟอร์ม | ปริมาณข้อมูลที่แนะนำ (Gb) | ควบคุมคุณภาพ |
| cDNA-PCR (เสริมโพลี-เอ) | นาโนพอร์ โพรเมทไอออน P48 | 6 Gb/ตัวอย่าง (ขึ้นอยู่กับสายพันธุ์) | อัตราส่วนเต็มความยาว > 70% คะแนนคุณภาพเฉลี่ย: Q10
|
การวิเคราะห์ทางชีวสารสนเทศ
●การประมวลผลข้อมูลดิบ
● บัตรประจำตัวการถอดเสียง
● การประกบทางเลือก
● ปริมาณการแสดงออกในระดับยีนและระดับไอโซฟอร์ม
● การวิเคราะห์นิพจน์เชิงอนุพันธ์
● คำอธิบายประกอบฟังก์ชันและการตกแต่ง (DEG และ DET)
ข้อกำหนดตัวอย่างและการจัดส่ง
ข้อกำหนดตัวอย่าง:
นิวคลีโอไทด์:
| ความเข้มข้น(ng/μl) | ปริมาณ (ไมโครกรัม) | ความบริสุทธิ์ | ความซื่อสัตย์ |
| ≥ 100 | ≥ 0.6 | OD260/280=1.7-2.5 โอดี260/230=0.5-2.5 มีการปนเปื้อนของโปรตีนหรือ DNA อย่างจำกัดหรือไม่มีเลยบนเจล | สำหรับพืช: RIN≥7.0; สำหรับสัตว์: RIN≥7.5; 5.0≥28S/18S≥1.0; ระดับความสูงพื้นฐานที่จำกัดหรือไม่มีเลย |
เนื้อเยื่อ: น้ำหนัก(แห้ง): ≥1 g
*สำหรับเนื้อเยื่อที่มีขนาดเล็กกว่า 5 มก. เราแนะนำให้ส่งตัวอย่างเนื้อเยื่อแช่แข็งแบบแฟลช (ในไนโตรเจนเหลว)
การระงับเซลล์: จำนวนเซลล์ = 3 × 106- 1×107
*เราแนะนำให้จัดส่งไลเซทเซลล์แช่แข็งในกรณีที่เซลล์นั้นนับน้อยกว่า 5×105แนะนำให้ใช้แฟลชแช่แข็งในไนโตรเจนเหลว ซึ่งเหมาะสำหรับการสกัดแบบไมโคร
ตัวอย่างเลือด: ปริมาตร≥1มล
การจัดส่งตัวอย่างที่แนะนำ
ภาชนะบรรจุ: หลอดหมุนเหวี่ยงขนาด 2 มล. (ไม่แนะนำให้ใช้ฟอยล์ดีบุก)
การติดฉลากตัวอย่าง: กลุ่ม+ทำซ้ำ เช่น A1, A2, A3;บี1 บี2 บี3......
การจัดส่ง: 2、น้ำแข็งแห้ง: ตัวอย่างจะต้องบรรจุในถุงและฝังในน้ำแข็งแห้ง
- หลอด RNAstable: ตัวอย่าง RNA สามารถทำให้แห้งในหลอดรักษาเสถียรภาพ RNA (เช่น RNAstable®) และจัดส่งในอุณหภูมิห้อง
ขั้นตอนการทำงานบริการ
นิวคลีโอไทด์:

การจัดส่งตัวอย่าง

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
ขั้นตอนการทำงานบริการ
เนื้อเยื่อ:

การออกแบบการทดลอง

การจัดส่งตัวอย่าง

การสกัดอาร์เอ็นเอ

การก่อสร้างห้องสมุด

การเรียงลำดับ

การวิเคราะห์ข้อมูล

บริการหลังการขาย
1.การวิเคราะห์การแสดงออกเชิงอนุพันธ์ - โครงเรื่องภูเขาไฟ
การวิเคราะห์การแสดงออกที่แตกต่างกันสามารถประมวลผลได้ทั้งในระดับยีนเพื่อระบุยีนที่แสดงออกแตกต่างกัน (DEG) และในระดับไอโซฟอร์มเพื่อระบุยีนที่แตกต่างกัน
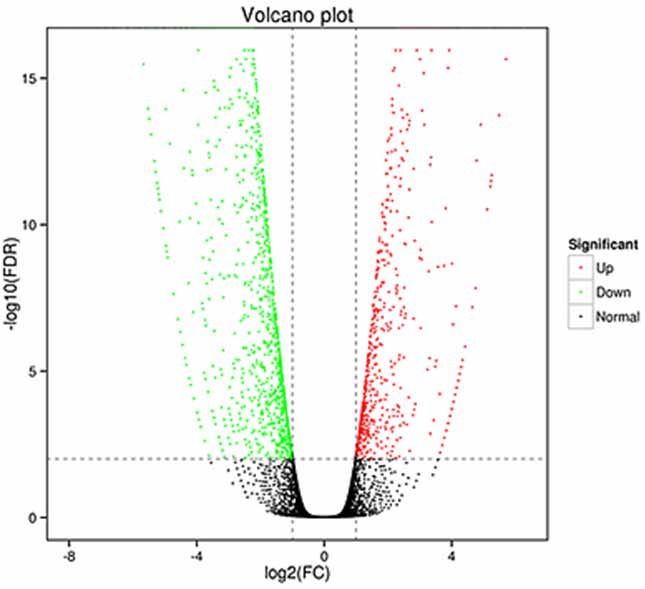
ใบรับรองผลการเรียนที่แสดง (DET)
2.แผนที่ความร้อนการจัดกลุ่มแบบลำดับชั้น
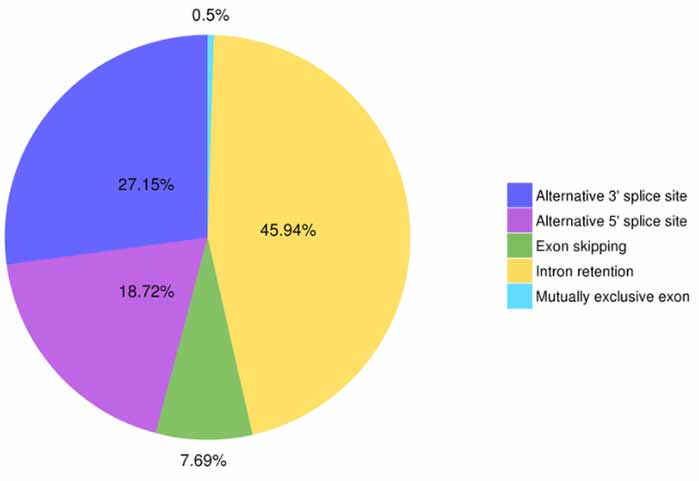
3. การระบุและการจำแนกประเภทประกบทางเลือก
Astalavista สามารถคาดการณ์เหตุการณ์การประกบทางเลือกได้ห้าประเภท
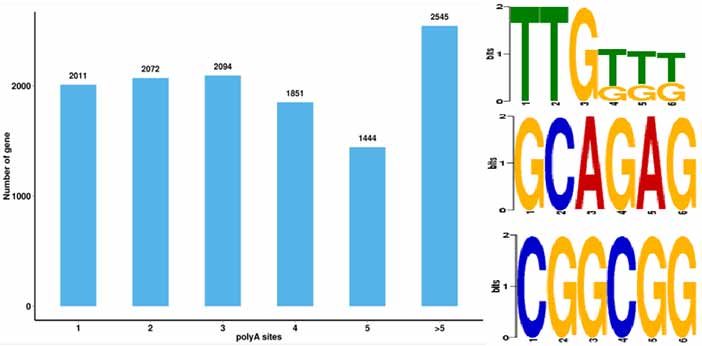
4.การระบุเหตุการณ์โพลีอะดีนีเลชันทางเลือก (APA) และ Motif ที่ 50 bp ต้นทางของโพลี-A
คดีบีเอ็มเค
การระบุการประกบทางเลือกและการหาปริมาณระดับไอโซฟอร์มโดยการจัดลำดับทรานสคริปโตมแบบเต็มความยาวระดับนาโนพอร์
ที่ตีพิมพ์:การสื่อสารธรรมชาติ 2563
กลยุทธ์การจัดลำดับ:
การจัดกลุ่ม: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (การกลายพันธุ์ K700E);3. บีเซลล์ปกติ
กลยุทธ์การจัดลำดับ: การจัดลำดับไลบรารี MiniION 2D, การจัดลำดับไลบรารี PromethION 1D;ข้อมูลอ่านสั้นจากตัวอย่างเดียวกัน
แพลตฟอร์มการหาลำดับ: Nanopore Minion;นาโนพอร์ โพรเมธไอออน;
ผลลัพธ์ที่สำคัญ
1. การระบุรอยต่อทางเลือกระดับไอโซฟอร์ม
ลำดับการอ่านแบบยาวช่วยให้สามารถระบุ SF3B1 กลายพันธุ์ได้K700E- แก้ไขจุดต่อรอยที่ระดับไอโซฟอร์มพบว่า 35 ทางเลือก 3′SSs และ 10 ทางเลือก 5′SSs พบว่ามีความแตกต่างกันอย่างมีนัยสำคัญระหว่าง SF3B1K700Eและ SF3B1WT.การเปลี่ยนแปลง 33 รายการจากทั้งหมด 35 รายการถูกค้นพบใหม่โดยลำดับที่อ่านมานาน
2. การหาปริมาณการประกบทางเลือกระดับไอโซฟอร์ม
การแสดงออกของไอโซฟอร์มการกักเก็บอินตรอน (IR) ใน SF3B1K700Eและ SF3B1WTถูกหาปริมาณตามลำดับนาโนพอร์ ซึ่งเผยให้เห็นการควบคุมทั่วโลกของไอโซฟอร์ม IR ใน SF3B1K700E.
อ้างอิง
Tang AD, Soulette CM, บาเรน MJV และคณะการแสดงลักษณะการถอดเสียงแบบเต็มความยาวของการกลายพันธุ์ SF3B1 ในมะเร็งเม็ดเลือดขาวชนิดลิมโฟไซติกเรื้อรังเผยให้เห็นการลดลงของอินตรอนที่คงอยู่ [J]การสื่อสารธรรมชาติ