TRANSKRIPTOMI
natur
KOMMUNIKATIONER
Fullängdstranskriptkarakterisering av SF3B1-mutation vid kronisk lymfatisk leukemi avslöjar nedreglering av kvarhållna introner
Avskrifter i full längd|Nanopore-sekvensering|Alternativ isoformanalys
Bakgrund
Somatiska mutationer i splitsningsfaktor SF3B1 har i stor utsträckning rapporterats associera med olika cancerformer, inklusive kronisk lymfatisk leukemi (CLL), uveal melanom, bröstcancer, etc. Dessutom har kortlästa transkriptomiska studier avslöjat avvikande splitsningsmönster inducerade av SF3B1-mutationer.Studier av dessa alternativa splitsningsmönster har dock länge varit begränsade till händelsenivå och bristande kunskap på isoformnivå på grund av begränsningen av kortlästa sammansatta transkript.Här introducerades nanopore-sekvenseringsplattform för att generera transkript i full längd, vilket bemyndigade inverstigation på AS-isoformer.
Experimentell design
Experiment
Gruppering:1. CLL-SF3B1(WT) 2. CLL-SF3B1(K700E-mutation);3. Normala B-celler
Sekvenseringsstrategi:MinION 2D-bibliotekssekvensering, PromethION 1D-bibliotekssekvensering;kortlästa data från samma prover
Sekvenseringsplattform:ONT MinION;ONT PromethION;
Bioinformatisk analys
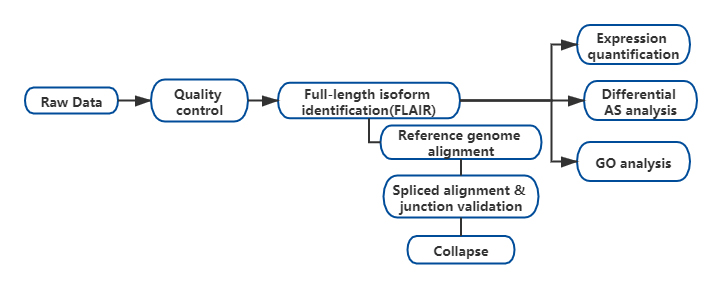
Resultat
Atotalt 257 miljoner avläsningar genererades från 6 CLL-prover och 3 B-celler.I genomsnitt identifierades 30,5 % av dessa läsningar som fullängdsavskrifter.
FAlternativ isoformanalys i full längd av RNA(FLAIR) utvecklades för att generera en uppsättning isoformer med hög konfidens.FLAIR kan sammanfattas som:
Nanopore läser anpassning: identifiera allmän transkriptstruktur baserat på referensgenom;
Splice junction correction: korrigera sekvensfel (röda) med splitsningsställe från antingen kommenterade introner, introner från kortlästa data eller båda;
Collapse: sammanfatta representativa isoformer baserade på skarvövergångskedjor (första passageuppsättning).Välj högsäkerhets isofrom baserat på antalet stödjande läsningar (Tröskel: 3).
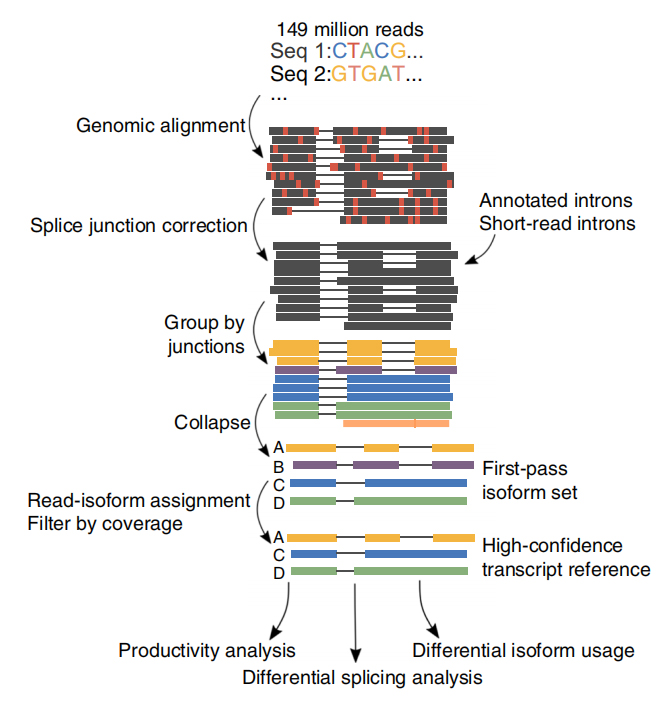
Figur 1. FLAIR-analys för att identifiera fulllängdsisoformer associerade med SF3B1-mutation i KLL
FLAIR identifierade 326 699 högsäkerhetsskarvade isoformer, varav 90 % är nya isoformer.De flesta av dessa okommenterade isoformer visade sig vara nya kombinationer av kända splitsningsövergångar (142 971), medan resten av nya isoformer innehöll antingen bibehållen intron (21 700) eller ny exon (3594).
Long-read-sekvenser möjliggör identifiering av mutant SF3B1-K700E-förändrade splitsningsställen på isoformnivå.35 alternativ 3'SS och 10 alternativ 5'SS visade sig vara signifikant differentiellt splitsade mellan SF3B1-K700E och SF3B1-WT.33 av de 35 förändringarna upptäcktes nyligen av långlästa sekvenser.I Nanopore-data är fördelningen av avståndet mellan SF3B1-K700E-förändrade 3'SS:er till toppar på kanoniska platser runt -20 bp, vilket skiljer sig signifikant från en kontrollfördelning, liknande vad som har rapporterats i KLL-kortlästa sekvenser.Isoformer av ERGIC3-genen analyserades, där en ny isoform innehållande det proximala splitsningsstället hittades mer rikligt i SF3B1-K700E.Både proximala och distala 3'SS associerades med distinkta AS-mönster som genererade flera isoformer.
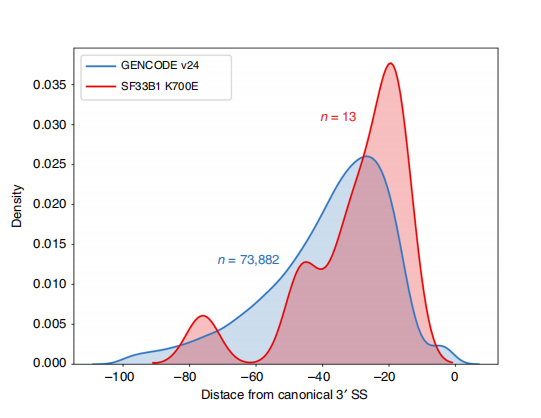
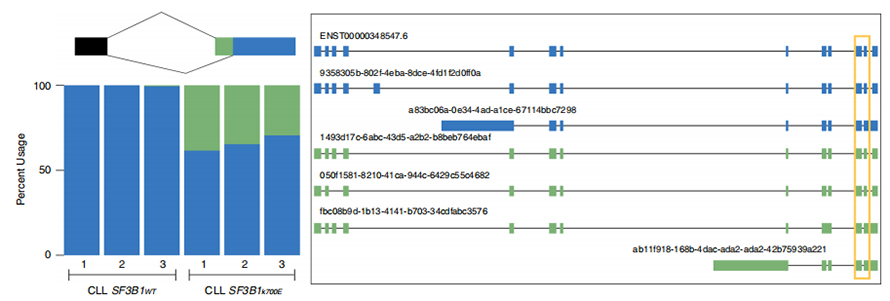
Figur 2. Alternativa 3′-skarvningsmönster identifierade med nanopore-sekvenseringsdata
IR-händelseanvändningsanalys har länge varit begränsad i kortläsningsbaserad analys på grund av förtroende för IR-identifiering och kvantifiering.Uttryck av IR-isoformer i SF3B1-K700E och SF3B1-WT kvantifierades baserat på nanoporsekvenser, vilket avslöjar en global nedreglering av IR-isoformer i SF3B1-K700E.
Figur 4. Jordbruksintensitet och nätverksanslutning mellan tre jordbrukssystem (A och B);Slumpmässig skogsanalys(C) och samband mellan jordbruksintensitet och AMF-kolonisering (D)
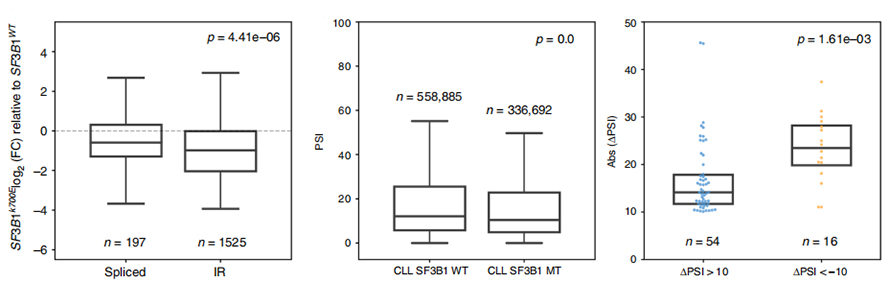
Figur 3. Intronrententionshändelser är kraftigare nedreglerade i CLL SF3B1-K700E
Teknologi
Nanopore Långläst Sequencing
Nanopore-sekvensering är en sekvenseringsteknik för elektrisk signalsekvens i realtid med en enda molekyl.
Ddubbelsträngat DNA eller RNA kommer att binda till nanoporöst protein inbäddat i biofilmen och varva ner under ledning av motorprotein.
DNA/RNA-strängar passerar genom nanoporkanalprotein med viss hastighet under inverkan av spänningsskillnad.
Molekuler genererar olika elektriska signaler beroende på kemisk struktur.
Real-time detektering av sekvenser uppnås genom basanrop.
Utförande av transkriptomsekvensering i full längd
√ Datamättnad
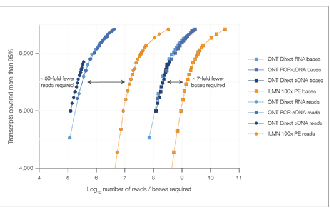
7 gånger färre läsningar krävs för att nå jämförbar datamättnad.
√ Identifiering av transkriptionsstruktur
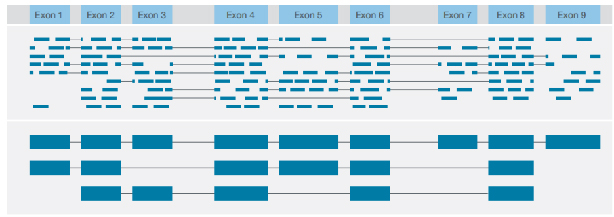
Identifiering av olika strukturella varianter med konsensusfullängdsavläsning av varje transkript
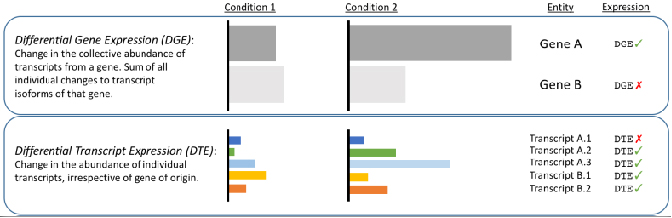
√ Differentialanalys på transkriptionsnivå - Avslöja förändringar som är dolda av kortläsningar
Referens
Tang AD, Soulette CM, Baren MJV, et al.Fullängdstranskriptkarakterisering av SF3B1-mutation vid kronisk lymfatisk leukemi avslöjar nedreglering av kvarhållna introner [J].Naturkommunikation.
Teknik och höjdpunkter syftar till att dela den senaste framgångsrika tillämpningen av olika högkapacitetssekvenseringsteknologier på olika forskningsarenor samt briljanta idéer inom experimentell design och datautvinning.
Posttid: Jan-08-2022

