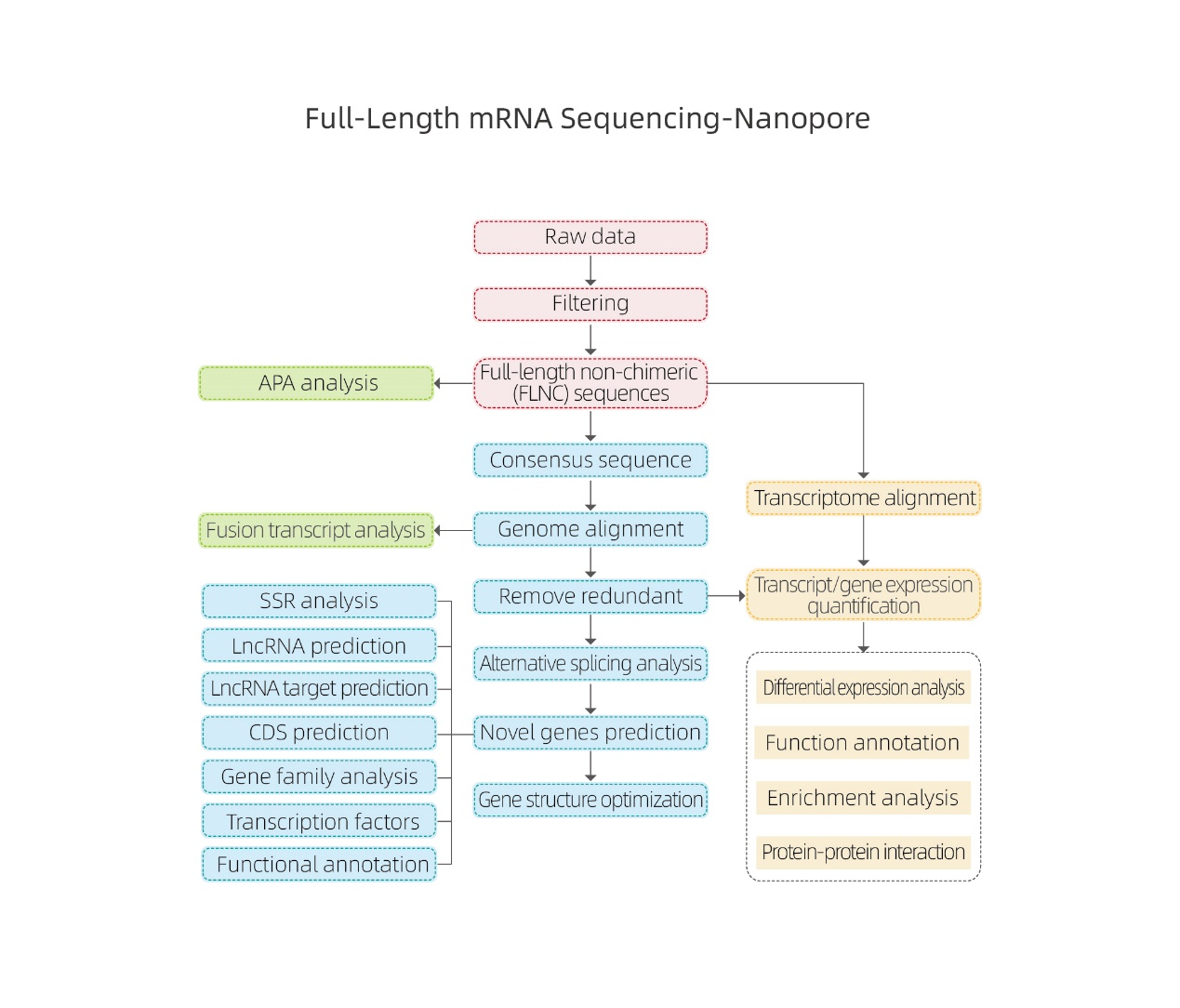
Fullängds mRNA-sekvensering-Nanopore
Servicefördelar
● Låg sekvensbias
● Avslöjar fullängds cDNA-molekyler
● Mindre data krävs för att täcka samma antal avskrifter
● Identifiering av flera isoformer per gen
● Kvantifiering av uttryck i isoformnivå
Servicespecifikationer
| Bibliotek | Plattform | Rekommenderat datautbyte (Gb) | Kvalitetskontroll |
| cDNA-PCR(Poly-A-berikad) | Nanopore PromethION P48 | 6 Gb/prov (beroende på art) | Fullängdsförhållande>70 % Genomsnittligt kvalitetspoäng: Q10
|
Bioinformatikanalyser
●Bearbetning av rådata
● Avskriftsidentifiering
● Alternativ skarvning
● Expressionskvantifiering i gennivå och isoformnivå
● Analys av differentiella uttryck
● Funktionskommentarer och berikning (DEG och DET)
Exempel på krav och leverans
Exempelkrav:
Nukleotider:
| Konc.(ng/μl) | Mängd (μg) | Renhet | Integritet |
| ≥ 100 | ≥ 0,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Begränsad eller ingen protein- eller DNA-kontamination visas på gelen. | För växter: RIN≥7,0; För djur: RIN≥7,5; 5,0≥28S/18S≥1,0; begränsad eller ingen baslinjehöjning |
Vävnad: Vikt (torr): ≥1 g
*För vävnad som är mindre än 5 mg rekommenderar vi att du skickar snabbfryst (i flytande kväve) vävnadsprov.
Cellsuspension: Cellantal = 3×106- 1×107
*Vi rekommenderar att du skickar fruset cellysat.Om cellen räknas mindre än 5×105, snabbfryst i flytande kväve rekommenderas, vilket är att föredra för mikroextraktion.
Blodprover: Volym≥1 ml
Rekommenderad provleverans
Behållare: 2 ml centrifugrör (tunnfolie rekommenderas inte)
Provmärkning: Grupp+replikat t.ex. A1, A2, A3;B1, B2, B3... ...
Leverans: 2、Torris: Proverna måste packas i påsar och grävas ner i torris.
- RNAstabila rör: RNA-prover kan torkas i RNA-stabiliseringsrör (t.ex. RNAstable®) och skickas i rumstemperatur.
Service Arbetsflöde
Nukleotider:

Provleverans

Byggande av bibliotek

Sekvensering

Dataanalys

Service efter försäljning
Service Arbetsflöde
Vävnad:

Experimentdesign

Provleverans

RNA-extraktion

Byggande av bibliotek

Sekvensering

Dataanalys

Service efter försäljning
1.Differentiell uttrycksanalys -Vulkanplot
Differentiell expressionsanalys kan bearbetas på både gennivå för att identifiera differentiellt uttryckta gener (DEG) och på isoformnivå för att identifiera differentiellt
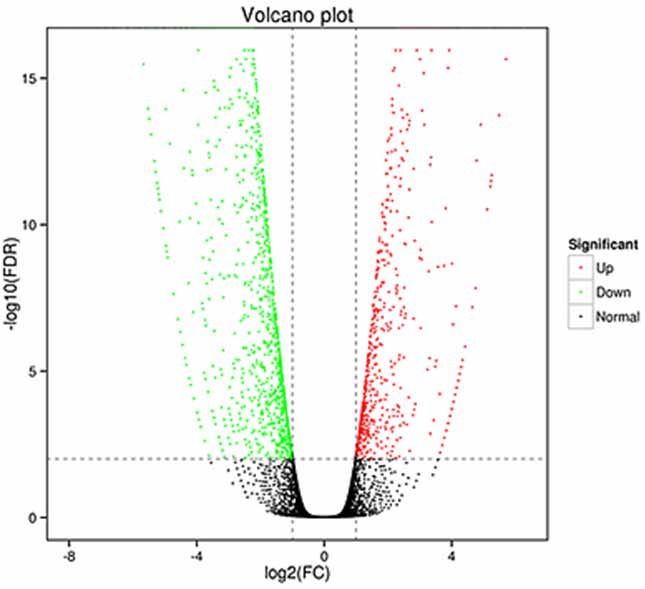
uttryckta transkriptioner (DET)
2.Hierarkisk klustringsvärmekarta
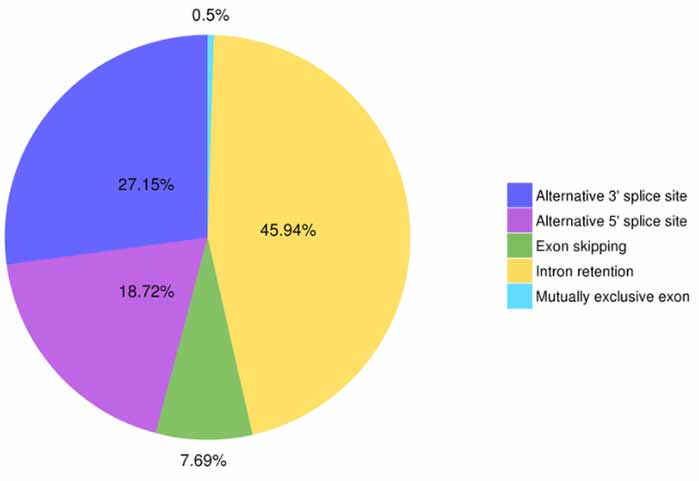
3.Alternativ skarvningsidentifiering och klassificering
Fem typer av alternativa splitsningshändelser kan förutsägas av Astalavista.
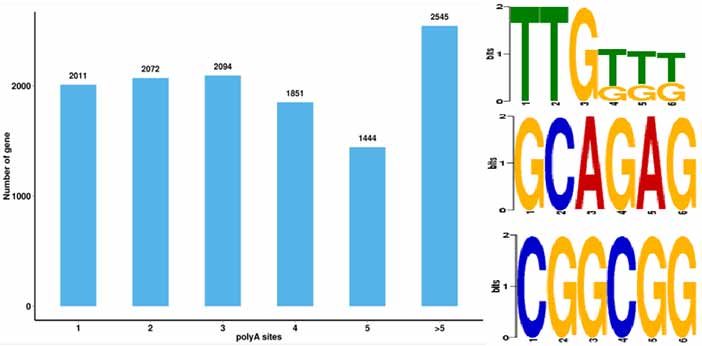
4.Identifiering av alternativ polyadenylering (APA) händelser och motiv vid 50 bp uppströms poly-A
BMK Fall
Alternativ splitsningsidentifiering och kvantifiering på isoformnivå genom nanopore fullängds transkriptomsekvensering
Publicerad:Naturkommunikation, 2020
Sekvenseringsstrategi:
Gruppering: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (K700E-mutation);3. Normala B-celler
Sekvenseringsstrategi: MinION 2D-bibliotekssekvensering, PromethION 1D-bibliotekssekvensering;kortlästa data från samma prover
Sekvenseringsplattform: Nanopore MinION;Nanopore PromethION;
Nyckelresultat
1. Alternativ skarvningsidentifiering på isoformnivå
Långlästa sekvenser möjliggör identifiering av mutant SF3B1K700E-förändrade skarvställen på isoformnivå.35 alternativa 3'SS och 10 alternativa 5'SS visade sig vara signifikant differentiellt splitsade mellan SF3B1K700Eoch SF3B1WT.33 av de 35 förändringarna upptäcktes nyligen av långlästa sekvenser.
2. Kvantifiering av alternativ skarvning på isoformnivå
Uttryck av intronretention (IR) isoformer i SF3B1K700Eoch SF3B1WTkvantifierades baserat på nanoporsekvenser, vilket avslöjar en global nedreglering av IR-isoformer i SF3B1K700E.
Referens
Tang AD, Soulette CM, Baren MJV, et al.Fullängdstranskriptkarakterisering av SF3B1-mutation vid kronisk lymfatisk leukemi avslöjar nedreglering av kvarhållna introner [J].Naturkommunikation.