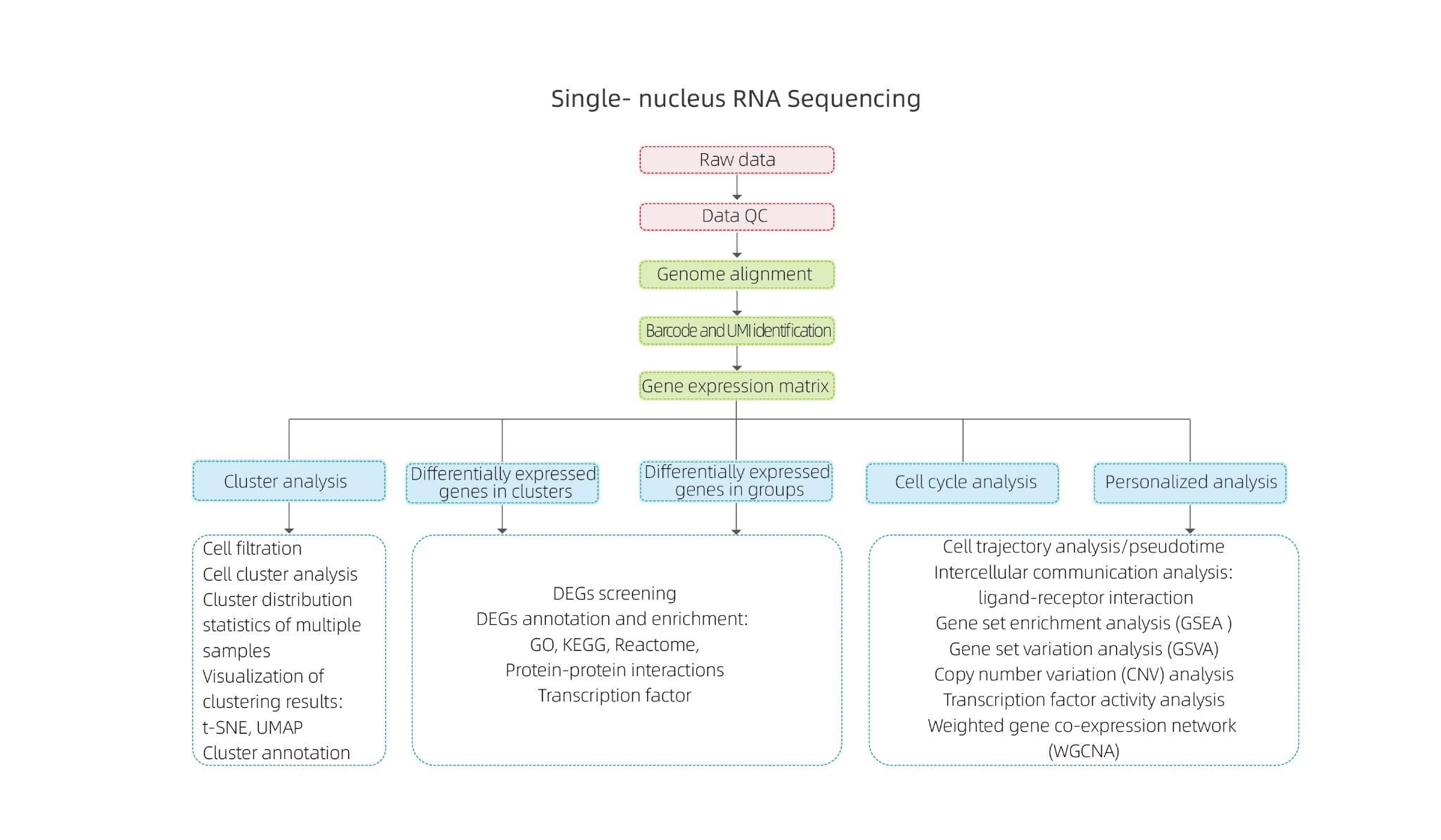
Sequenciamento de RNA de núcleo único
Princípio técnico
O isolamento dos núcleos é conseguido pelo 10× Genomics ChromiumTM, que consiste em um sistema microfluídico de oito canais com cruzamentos duplos.Neste sistema, esferas de gel com códigos de barras e primer, enzimas e um único núcleo são encapsuladas em uma gota de óleo do tamanho de um nanolitro, gerando Gel Bead-in-Emulsion (GEM).Uma vez formado o GEM, a lise celular e a liberação dos códigos de barras são realizadas em cada GEM.O mRNA é transcrito reversamente em moléculas de cDNA com códigos de barras 10× e UMI, que estão ainda sujeitos à construção de biblioteca de sequenciamento padrão.
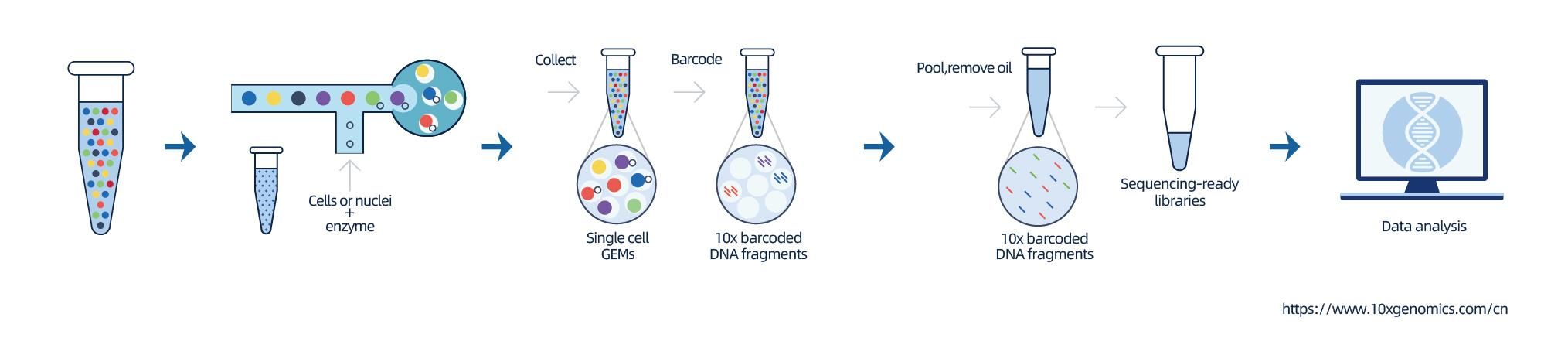
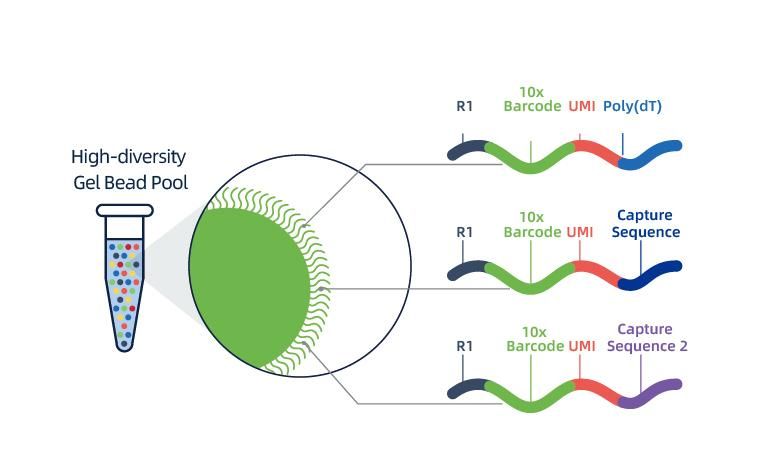
Tecido não adequado para preparação de suspensão unicelular
| Célula / Tecido | Razão |
| Tecido congelado não fresco | Não é possível obter organizações novas ou salvas há muito tempo |
| Célula muscular, megacariócito, gordura… | O diâmetro da célula é muito grande para entrar no instrumento |
| Fígado… | Muito frágil para quebrar, incapaz de distinguir células individuais |
| Célula neuronal, cérebro… | Mais sensível, fácil de estressar, mudará os resultados do sequenciamento |
| Pâncreas, Tireóide… | Rico em enzimas endógenas, afetando a produção de suspensão unicelular |
Núcleo único vs célula única
| Núcleo único | Célula única |
| Diâmetro celular ilimitado | Diâmetro celular: 10-40 μm |
| O material pode ser tecido congelado | O material deve ser tecido fresco |
| Baixo estresse de células congeladas | O tratamento enzimático pode causar reação de estresse celular |
| Nenhum glóbulo vermelho precisa ser removido | Os glóbulos vermelhos precisam ser removidos |
| Nuclear expressa bioinformação | A célula inteira expressa bioinformação |
Especificações de serviço
| Biblioteca | Estratégia de sequenciamento | Volume de dados | Requisitos de amostra | Tecido |
| 10× biblioteca de núcleo único genômica | 10x Genômica -Illumina PE150 | 100.000 leituras/célula aprox.100-200 GB | Número de células: >2× 105 Conc. celularem 700-1.200 células/μL | ≥ 200 mg |
Para obter mais detalhes sobre orientações de preparação de amostras e fluxo de trabalho de serviço, sinta-se à vontade para falar com umEspecialista BMKGENE
Fluxo de trabalho de serviço

Projeto de experimento

Entrega de amostra
Isolamento de núcleos

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
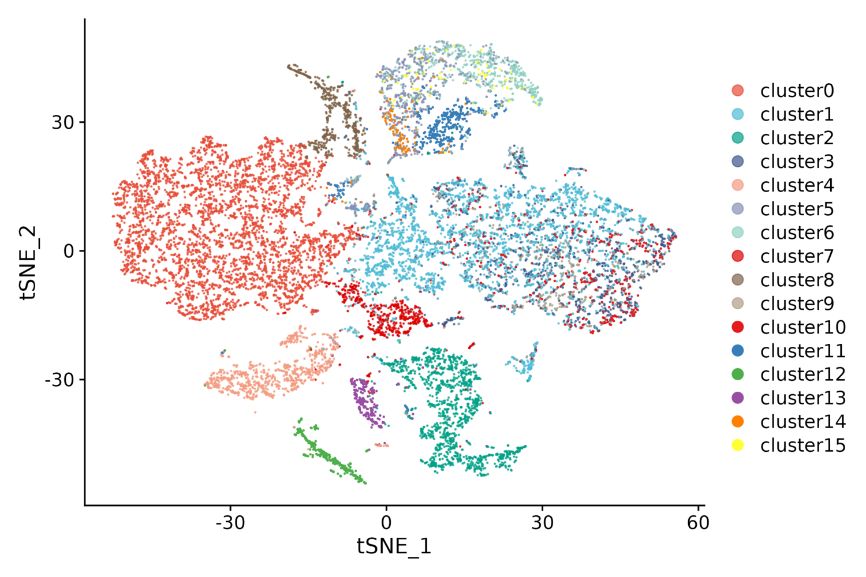
1. Agrupamento pontual
2.Mapa de calor de agrupamento de abundância de expressão de marcador
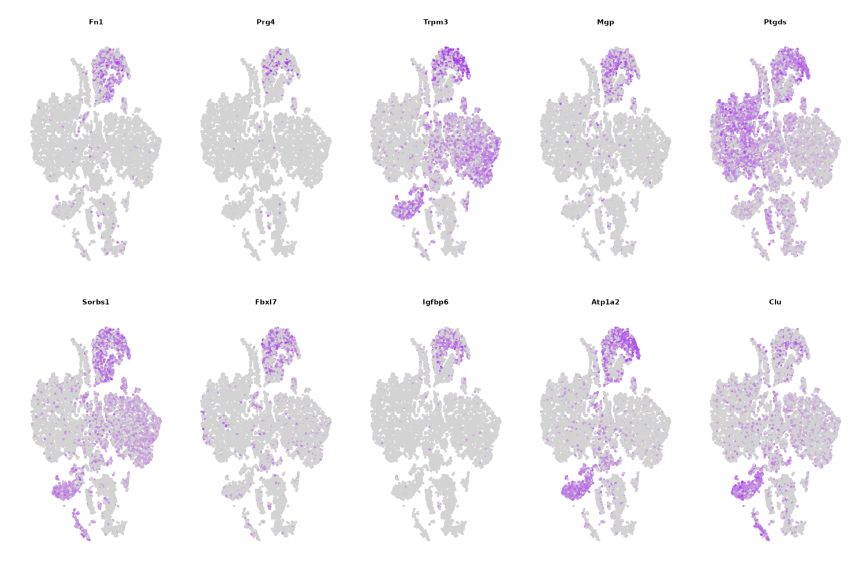
 3.Distribuição do gene Maker em diferentes clusters
3.Distribuição do gene Maker em diferentes clusters
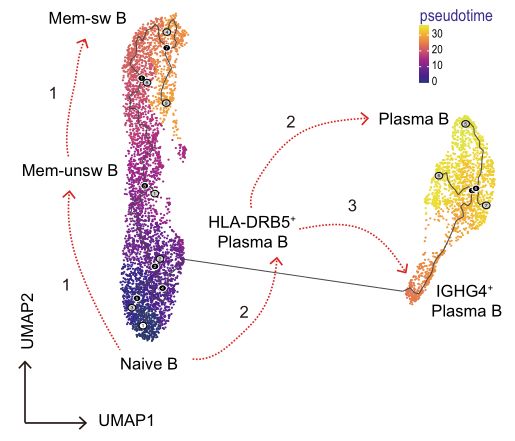
4. Análise de trajetória celular/pseudotempo