GENÔMICA HUMANA
genética da natureza
Sequenciamento de leitura longa identifica expansões repetidas de GGC em NOTCH2NLC associadas à doença de inclusão intranuclear neuronal
Resequenciamento ONT |Iluminar |Sequenciamento completo do exoma |Sequenciamento direcionado CRISPR-Cas9 ONT |RNA-seq |Chamada de metilação ONT 5mC
Destaques
1.Através da análise de ligação numa grande família NIID, foram identificadas duas regiões ligadas.
2. Sequenciamento de leitura longa baseado em ONT e enriquecimento mediado por Cas-9 O sequenciamento ONT descobriu uma causa genética potencial de NIID, expansões de repetição GGC em 5 ′ UTR de NOTCH2NLC.Este estudo relatou pela primeira vez expansões repetidas em genes específicos de humanos que evoluíram através de duplicações segmentares.
3. O sequenciamento de RNA revelou transcrições antisense anormais no início ou dentro das regiões de expansão repetidas de GGC em NOTCH2NLC.
Fundo
NA doença de inclusão intranuclear euronal (NIID) é uma doença neurodegenerativa progressiva e fatal, caracterizada pela presença de inclusões intranucleares hialinas eosinofílicas nos sistemas nervoso central e periférico.Suas manifestações clínicas altamente variáveis levantam grandes dificuldades no diagnóstico até a introdução da biópsia de pele.No entanto, os métodos baseados em histopatologia ainda sofrem de erros de diagnóstico, o que exige uma compreensão genética do NIID.
Conquistas
Análise de ligação
SO sequenciamento hort-read baseado no sequenciamento do genoma completo (WGS) e no sequenciamento completo do exoma (WES) foi realizado em uma grande família NIID (13 membros afetados e 7 não afetados).A análise de ligação em SNPs extraídos desses dados revelou apenas duas regiões ligadas: uma região de 3,5 Mb em 1p36.31-p36.22 (LOD máximo = 2,32) e uma região de 58,1 Mb em 1p22.1-q21.3 (LOD máximo: 4,21 ).No entanto, nenhum SNP ou CNV patogênico foi identificado nessas regiões ligadas.
Expansões de repetição GGC em NOTCH2NLC
NO sequenciamento baseado em anóporo foi processado em 13 membros afetados e 4 não afetados de 8 famílias (outro membro afetado foi sequenciado pela plataforma de sequenciamento de leitura longa Pacbio).Dados de leitura longa revelaram expansões de repetição de GGC associadas a doenças no mapeamento do gene 5′ UTR de NOTCH2NLC para a região ligada a 58,1 Mb (Figura 1).Estas expansões repetidas também foram identificadas em todos os 40 casos esporádicos de NIID testados por RP-PCR.
CO sequenciamento de alvo mediado por as-9 na plataforma nanopore foi empregado para obter maior cobertura de leitura na repetição NOTCH2NLC (100 X-1.795 X).Estas sequências de consenso concordaram bem com descobertas anteriores sobre expansões repetidas de GGC.Além disso, repetições {(GGA)n (GGC)n}n foram identificadas como um potencial marcador genético para o fenótipo dominante de fraqueza (Figura 2).
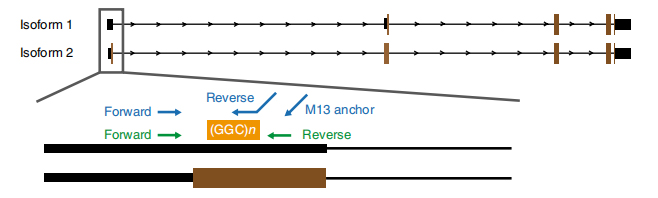
Figura 1. Expansão repetida associada à doença identificada no exon 1 das isoformas NOTCH2NLC.
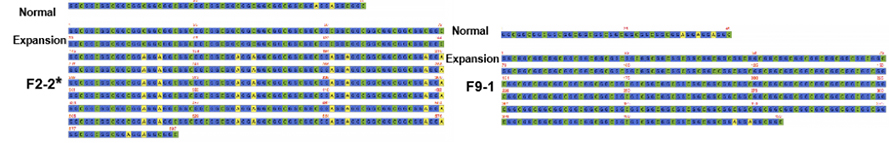
Figura 2. Sequências de consenso de repetição de NPTCH2NLC em pacientes NIID com (*) ou sem fenótipo dominante de fraqueza
NOs genes OTCH2NL são genes específicos de humanos, que se acredita desempenharem um papel vital na evolução do cérebro humano e nas doenças neurológicas.No entanto, três genes relacionados com NOTCH2 (NOTCH2NLA, NOTCH2NLB e NOTCH2NLC) com> 99,1% de identidade de sequência não foram resolvidos até a última montagem do genoma humano.O sequenciamento livre de síntese e de leitura longa na plataforma nanopore mostrou vantagens notáveis na resolução de regiões de alta similaridade e repetições (GGC)n com 100% rico em GC.
Expansões de repetição GGC em NOTCH2NLC
To sequenciamento do ranscriptoma foi processado em 2 membros afetados e 2 não afetados.A profundidade de leitura normalizada foi calculada nas cadeias sense e antisense a montante dos primeiros exons dos parálogos NOTCH2NL.Transcrições anti-sentido anormais foram encontradas apenas nos casos afetados, que ficam no início ou dentro da região de expansão repetida (picos roxos em F1-14 e F1-16 na Figura 3).Além disso, foram identificados 54 DEGs e todos foram enriquecidos em termos GO e MPO relacionados às funções neuronais.
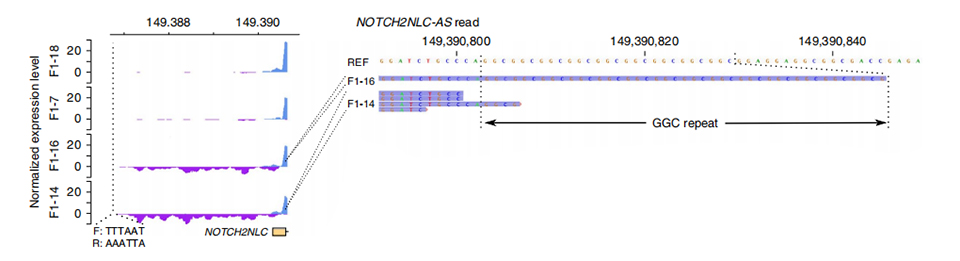
Figura 3. Profundidade de leitura normalizada a montante do primeiro exon de NOTCH2NLC em casos não afetados (acima) e afetados (abaixo).
Tecnologia
Tecnologias Oxford Nanopore (ONT)
NO sequenciamento anopore se diferencia de outras plataformas de sequenciamento, pois os nucleotídeos são lidos diretamente, sem processo de síntese de DNA.À medida que uma fita simples de DNA passa por um poro proteico de tamanho nanométrico (nanopore), diferentes nucleotídeos geram diferentes correntes iônicas, que podem ser capturadas e transferidas em sequências de bases.A própria plataforma de sequenciamento ONT não mostra limite técnico aparente no comprimento da leitura do DNA.Portanto, leituras ultralongas (ULRs) estão disponíveis para montagem de genoma de alta qualidade.Além disso, essas leituras extremamente longas, que são longas o suficiente para cruzar características de sequências complexas ou variações estruturais, ajudam a superar as limitações do sequenciamento de leitura curta aqui.

Sequenciamento de nanoporos
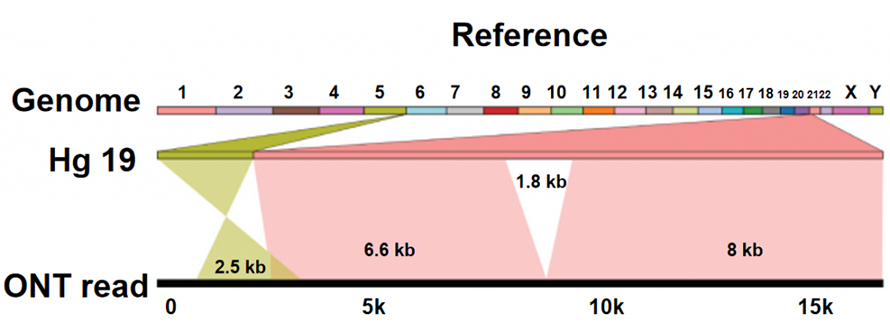
Identificação da variação da estrutura (SV)
So sequenciamento livre de síntese preservou amplamente as informações de metilação do DNA no modelo.A, T, C e G metilados geram correntes iônicas distintas das não metiladas, que podem ser lidas diretamente pela plataforma.O sequenciamento de nanoporos permite o perfil do genoma completo de 5mC e 6mA com resolução de nucleotídeo único.
Referência
Jun Sone, et.al.O sequenciamento de leitura longa identifica expansões repetidas de GGC em NOTCH2NLC associadas à doença de inclusão intranuclear neuronal.Genética da Natureza (2019)
Tecnologia e destaques visa compartilhar as mais recentes aplicações bem-sucedidas de diferentes tecnologias de sequenciamento de alto rendimento em diversas áreas de pesquisa, bem como ideias brilhantes em design experimental e mineração de dados.
Horário da postagem: 06/01/2022

