Sequenciamento completo de mRNA-Nanopore
Vantagens do serviço
● Viés de sequência baixo
● Revelando moléculas de cDNA completas
● Menos dados necessários para cobrir o mesmo número de transcrições
● Identificação de múltiplas isoformas por gene
● Quantificação de expressão em nível de isoforma
Especificações de serviço
| Biblioteca | Plataforma | Rendimento de dados recomendado (Gb) | Controle de qualidade |
| cDNA-PCR (enriquecido com Poli-A) | Nanopore PromethION P48 | 6 Gb/amostra (dependendo da espécie) | Proporção de comprimento total> 70% Índice de qualidade médio: Q10
|
Análises de bioinformática
●Processamento de dados brutos
● Identificação da transcrição
● Emenda alternativa
● Quantificação de expressão em nível de gene e nível de isoforma
● Análise de expressão diferencial
● Anotação e enriquecimento de função (DEGs e DETs)
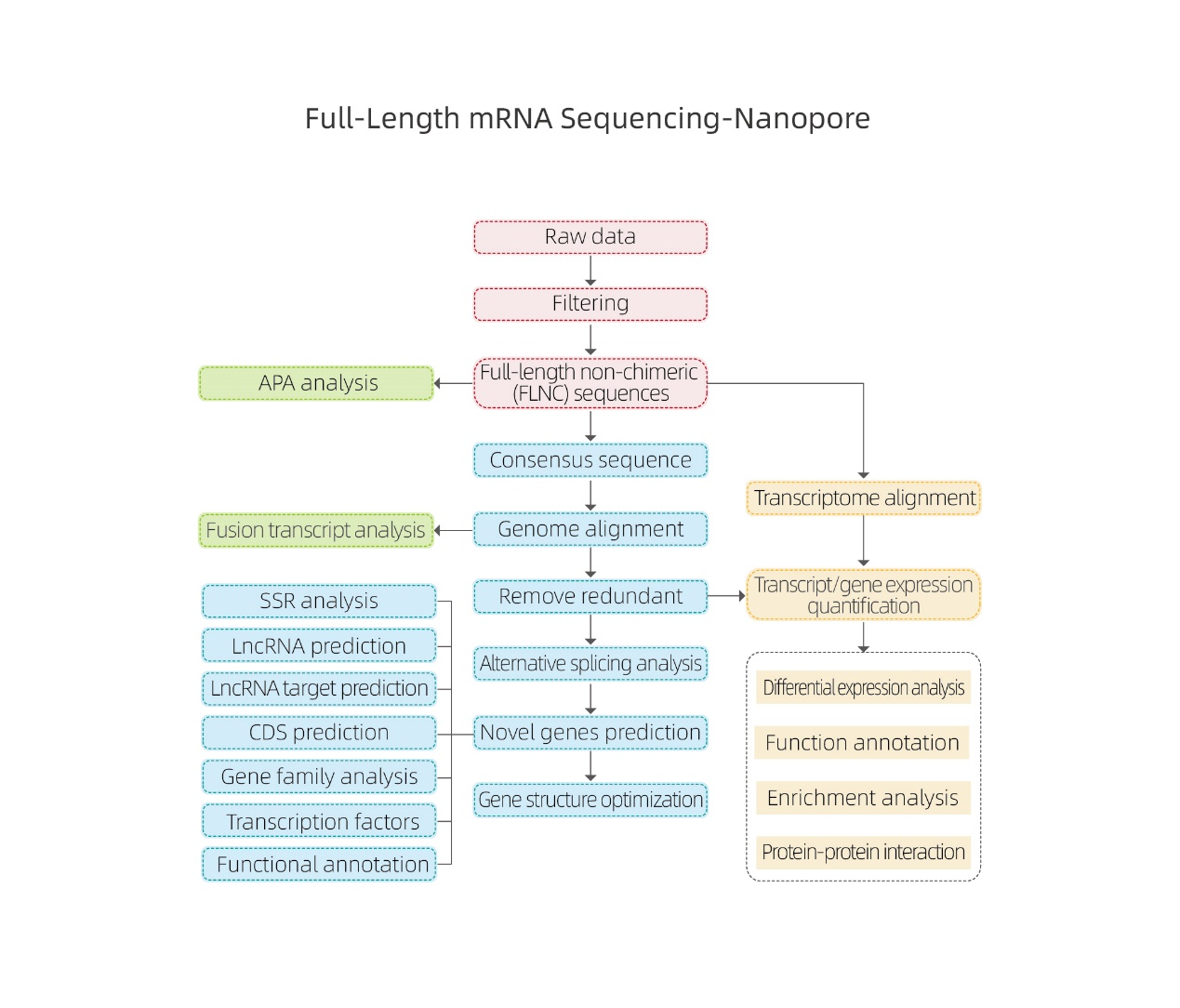
Requisitos de amostra e entrega
Requisitos de amostra:
Nucleotídeos:
| Conc.(ng/μl) | Quantidade (μg) | Pureza | Integridade |
| ≥ 100 | ≥ 0,6 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contaminação limitada ou inexistente de proteínas ou DNA mostrada no gel. | Para plantas: RIN≥7,0; Para animais: RIN≥7,5; 5,0≥28S/18S≥1,0; elevação limitada ou nenhuma elevação da linha de base |
Tecido: Peso (seco): ≥1g
*Para tecidos menores que 5 mg, recomendamos o envio de amostra de tecido congelado (em nitrogênio líquido).
Suspensão celular: Contagem de células = 3×106- 1×107
*Recomendamos enviar lisado celular congelado.Caso a contagem de células seja menor que 5×105, recomenda-se o congelamento instantâneo em nitrogênio líquido, que é preferível para microextração.
Amostras de sangue: Volume≥1 ml
Entrega de amostra recomendada
Recipiente: tubo de centrífuga de 2 ml (folha de estanho não é recomendada)
Rotulagem da amostra: Grupo+réplica, por exemplo, A1, A2, A3;B1, B2, B3... ...
Remessa: 2、Gelo seco: as amostras precisam ser embaladas em sacos e enterradas em gelo seco.
- Tubos RNAstable: As amostras de RNA podem ser secas em tubo de estabilização de RNA (por exemplo, RNAstable®) e enviadas em temperatura ambiente.
Fluxo de trabalho de serviço
Nucleotídeos:

Entrega de amostra

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
Fluxo de trabalho de serviço
Tecido:

Projeto de experimento

Entrega de amostra

Extração de RNA

Construção de biblioteca

Sequenciamento

Análise de dados

Serviços pós-venda
1.Análise de expressão diferencial - gráfico do vulcão
A análise de expressão diferencial pode ser processada tanto no nível do gene para identificar genes diferencialmente expressos (DEGs) quanto no nível da isoforma para identificar diferencialmente
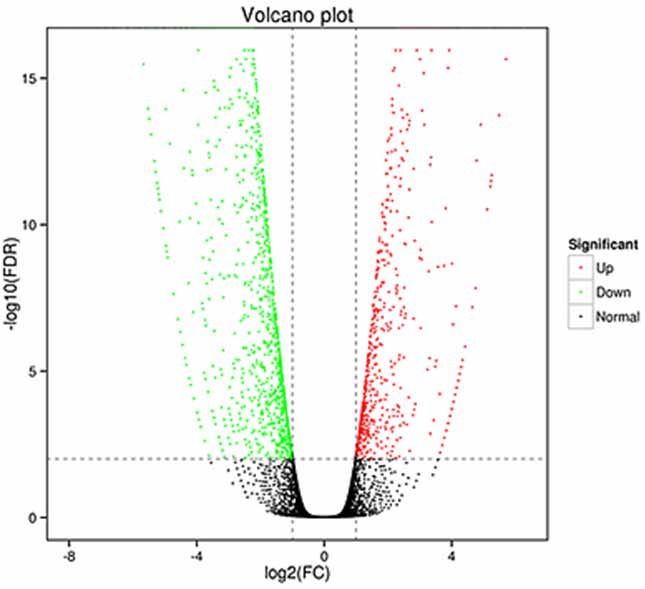
transcrições expressas (DETs)
2.Mapa de calor de cluster hierárquico
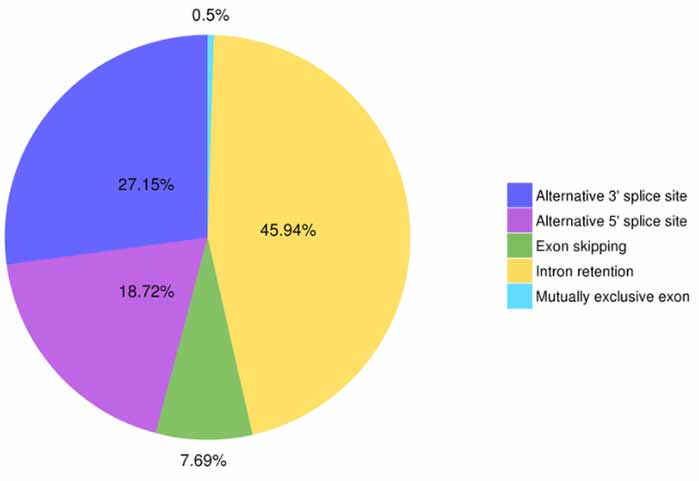
3. Identificação e classificação de emenda alternativa
Cinco tipos de eventos de splicing alternativo podem ser previstos pelo Astalavista.
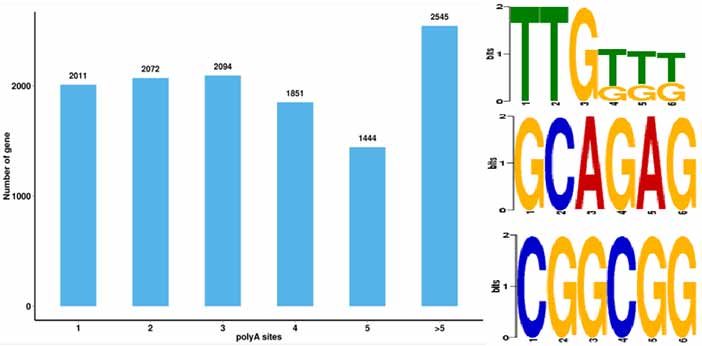
4.Identificação alternativa de eventos de poliadenilação (APA) e motivo a 50 pb a montante de poli-A
Caso BMK
Identificação de splicing alternativo e quantificação em nível de isoforma por sequenciamento de transcriptoma completo de nanoporos
Publicados:Comunicações da Natureza, 2020
Estratégia de sequenciamento:
Agrupamento: 1. CLL-SF3B1(WT);2. LLC-SF3B1 (mutação K700E);3. Células B normais
Estratégia de sequenciamento: sequenciamento da biblioteca MinION 2D, sequenciamento da biblioteca PromethION 1D;dados de leitura curta das mesmas amostras
Plataforma de sequenciamento: Nanopore MinION;Nanopore PromethION;
Principais resultados
1. Identificação de emenda alternativa em nível de isoforma
Sequências de leitura longa permitem a identificação do mutante SF3B1K700E- locais de splice alterados no nível da isoforma.Descobriu-se que 35 3′SSs alternativos e 10 5′SSs alternativos foram emendados significativamente diferencialmente entre SF3B1K700Ee SF3B1WT.33 das 35 alterações foram descobertas recentemente por sequências de leitura longa.
2.Quantificação de emenda alternativa em nível de isoforma
Expressão de isoformas de retenção de íntrons (IR) em SF3B1K700Ee SF3B1WTforam quantificados com base em sequências de nanoporos, revelando uma regulação negativa global das isoformas de IR em SF3B1K700E.
Referência
Tang AD, Soulette CM, Baren MJV, et al.A caracterização completa do transcrito da mutação SF3B1 na leucemia linfocítica crônica revela regulação negativa de íntrons retidos [J].Comunicações da Natureza.