Sequenciamento de DNA/RNA – Sequenciador Nanopore
Recursos de detalhes do serviço
| Plataforma | Tamanho da biblioteca | Rendimento teórico de dados (por célula) | Precisão de base única | Formulários |
| Nanoporo | 8Kb, 10kb, 20kb, Ultralongo, cDNA-PCR | 70-90 Gb/célula | 85-92% | Chamada SV, De novo, Sequenciamento completo, Iso-Seq, Anotação genética, Detecção de metilação do DNA |
Vantagens do serviço
● Mais de 5 anos de experiência na plataforma de sequenciamento PacBio com milhares de projetos fechados com diversas espécies.
● A BMKGENE é parceira oficial da Oxford Nanopore, com certificação de dupla plataforma RNA/DNA.
● Existem modelos convencionais de sequenciadores com equipamento completo e capacidade de sequenciamento suficiente.
● Com base na plataforma Nanopore, mais de 10 pesquisas Denovo em animais e plantas foram publicadas em revistas de renome internacional.
Requisitos de amostra
| Tipo de amostra | Quantia | Concentração(Qubit ®) | Volume | Pureza | Outros |
| DNA genômico | Depende da exigência de dados | ≥20ng/μl | ≥15μl | DO260/280=1,7-2,2; OD260/230≥1,5; Pico claro em 260 nm, sem contaminações | A concentração precisa ser medida por Qubit e Qubit/Nanopore ≤ 2 |
| ARN total | ≥1,2 μg | ≥100μg/μl | ≥15μl | DO260/280=1,7-2,5; OD260/230=0,5-2,5;sem contaminações | Valor RIN ≥7,5 |
Fluxo de trabalho de serviço
Preparação de amostra

Construção de biblioteca

Sequenciamento

Análise de dados

Entrega de projeto
Avaliação da qualidade dos dados da amostra de DNA
Tabela 1. Estatísticas sobre dados limpos.
| BMKID | rawSeqNum | rawSumBase | limparSeqNum | limparSumBase | limpoN50Len | limpoN90Len | cleanMeanLen | cleanMaxLen | cleanMeanQual |
| DNA_BMK01 | 1.218.239 | 26.37 | 1.121.736 | 25,90 | 28.014 | 15.764 | 23.090 | 143.181 | 9 |
Avaliação da qualidade dos dados da amostra de RNA
Tabela 1. Estatísticas sobre dados limpos.
| Nome do arquivo | ID do Cliente | LerNum | BaseNum | N50 | Comprimento Médio | Comprimento máximo | MédiaQscore |
| RNA_BMK001 | C2 | 8.947.708 | 4.047.230.083 | 398 | 452 | 129.227 | Q12 |
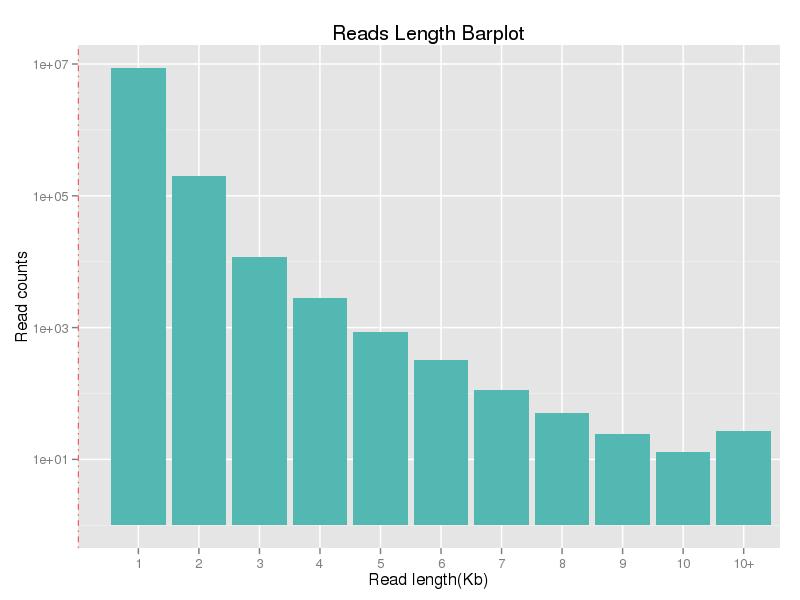
Figura 1. Distribuição do comprimento de leitura
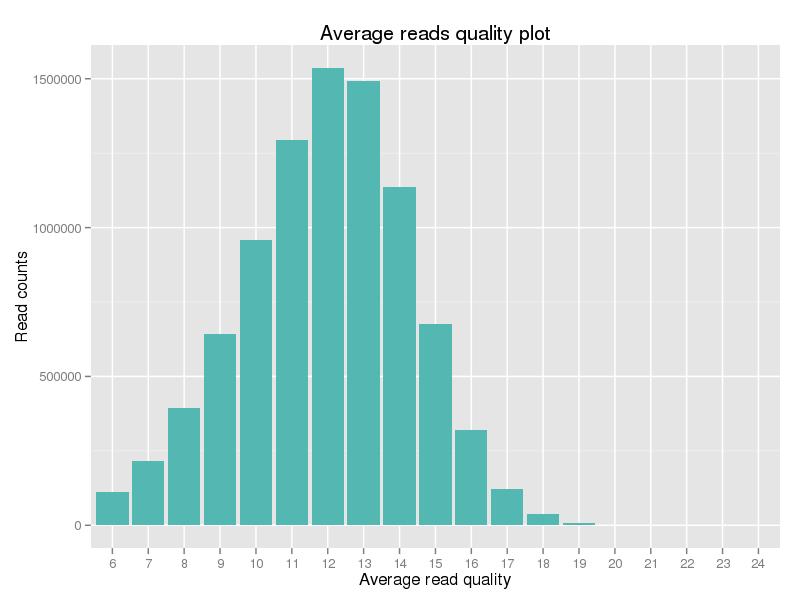
Figura 2. Distribuição do índice de qualidade de dados limpos
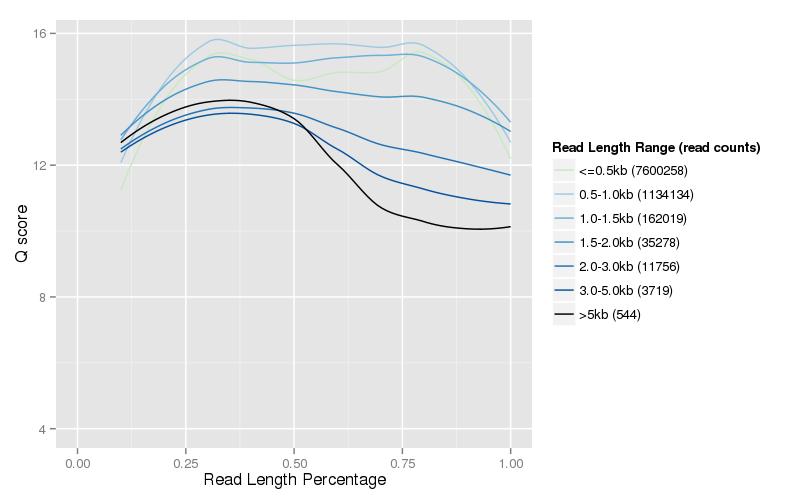
Figura 3. Distribuição de comprimento e pontuação de qualidade de dados limpos