Illumina e BGI
Características
●Plataformas:Illumina NovaSeq 6000, NovaSeq, HiSeq X Ten e BGI-DNB-T7
●Modos de sequenciamento:PE50, PE100, PE150, PE250
●Controle de qualidade de bibliotecas antes do sequenciamento
●Sequenciamento de entrega de dados e controle de qualidade:entrega de relatório de controle de qualidade e dados brutos em formato fastq após demultiplexação e filtragem de leituras Q30.
Vantagens do serviço
●Versatilidade dos serviços de sequenciamento:o cliente pode optar por sequenciar por pista, célula de fluxo ou quantidade de dados.
●Versatilidade de plataformas:Bibliotecas DNB podem ser transferidas para plataformas Illumina
●Ampla experiência na plataforma de sequenciamento Illumina:com milhares de projetos fechados com diversas espécies.
●Entrega do relatório de CQ de sequenciamento:com métricas de qualidade, precisão dos dados e desempenho geral do projeto de sequenciamento.
●Processo de sequenciamento maduro:com curto tempo de resposta.
●Rigoroso controle de qualidade: implementamos requisitos rigorosos de controle de qualidade para garantir a entrega de resultados consistentemente de alta qualidade.
Requisitos de amostra*
Sequenciamento parcial de pista
| Quantidade de dados (X) | Concentração (qPCR/nM) | Volume |
| X ≤ 50GB | ≥ 2nM | ≥ 20 μl |
| 50 GB ≤ X < 100 GB | ≥ 3nM | ≥ 20 μl |
| X ≥ 100 GB | ≥ 4nM | ≥ 20 μl |
Faixa única (Illumina)
| Plataforma | Concentração (qPCR/nM) | Volume |
| HiSeq X Dez | ≥ 2nM | ≥ 20 μl |
| NovaSeq 6000SP | ≥ 1nM | ≥ 25 μl |
| NovaSeq 6000 S4 | ≥ 1,5nM | ≥ 25 μl |
| NovaSeq X | ≥ 1,5nM | ≥ 25 μl |
| BGI-DNBSEQ-T7 | ≥ 1,5nM | ≥ 25 μl |
Além da concentração e da quantidade total, também é necessário um padrão de pico adequado.
Entre em contato conosco se suas amostras não atenderem aos requisitos de material inicial.
Fluxo de trabalho de serviço
Controle de qualidade da biblioteca

Sequenciamento

Controle de qualidade de dados

Entrega de projeto
Relatório de controle de qualidade da biblioteca
Um relatório sobre a qualidade da biblioteca é fornecido antes do sequenciamento, avaliando a quantidade e a fragmentação da biblioteca.
Relatório de CQ de sequenciamento
Tabela 1. Estatísticas sobre dados de sequenciamento.
| ID da amostra | BMKID | Leituras brutas | Dados brutos (pb) | Leituras limpas (%) | Q20(%) | Q30(%) | GC(%) |
| C_01 | BMK_01 | 22.870.120 | 6.861.036.000 | 96,48 | 99,14 | 94,85 | 36,67 |
| C_02 | BMK_02 | 14.717.867 | 4.415.360.100 | 96,00 | 98,95 | 93,89 | 37.08 |
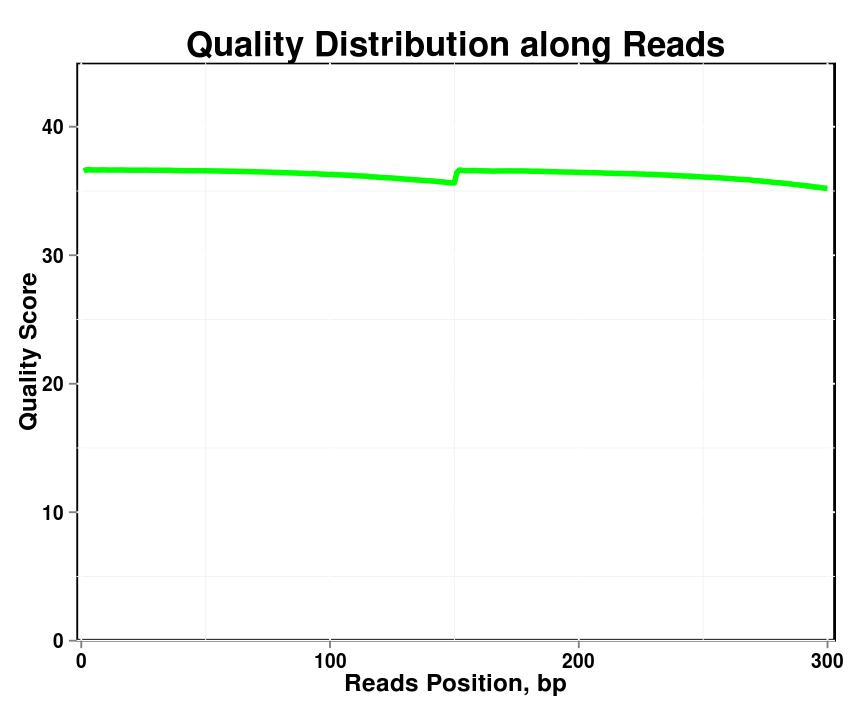
Figura 1. Distribuição de qualidade ao longo das leituras em cada amostra
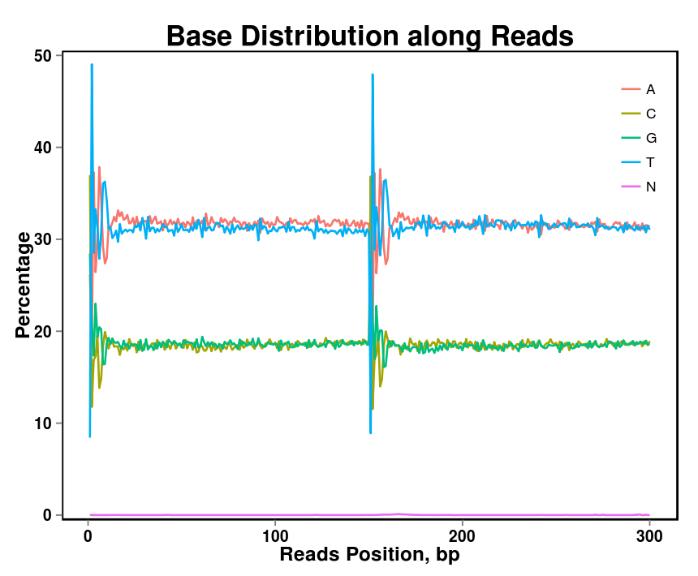
Figura 2. Distribuição de conteúdo base
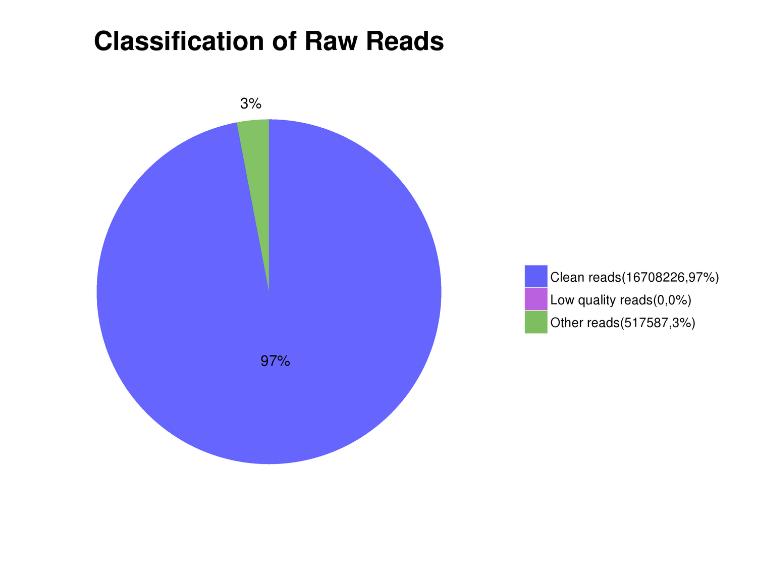
Figura 3. Distribuição do conteúdo lido nos dados de sequenciamento