Sekwencjonowanie całego transkryptomu – Illumina
Cechy
● Podwójna biblioteka do sekwencjonowania całego transkryptomu: wyczerpywanie rRNA, następnie przygotowanie biblioteki PE150 i selekcja wielkości, a następnie przygotowanie biblioteki SE50
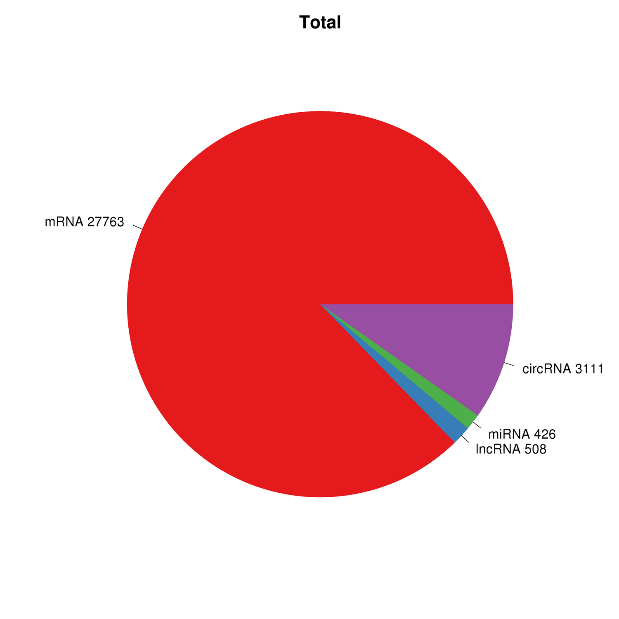
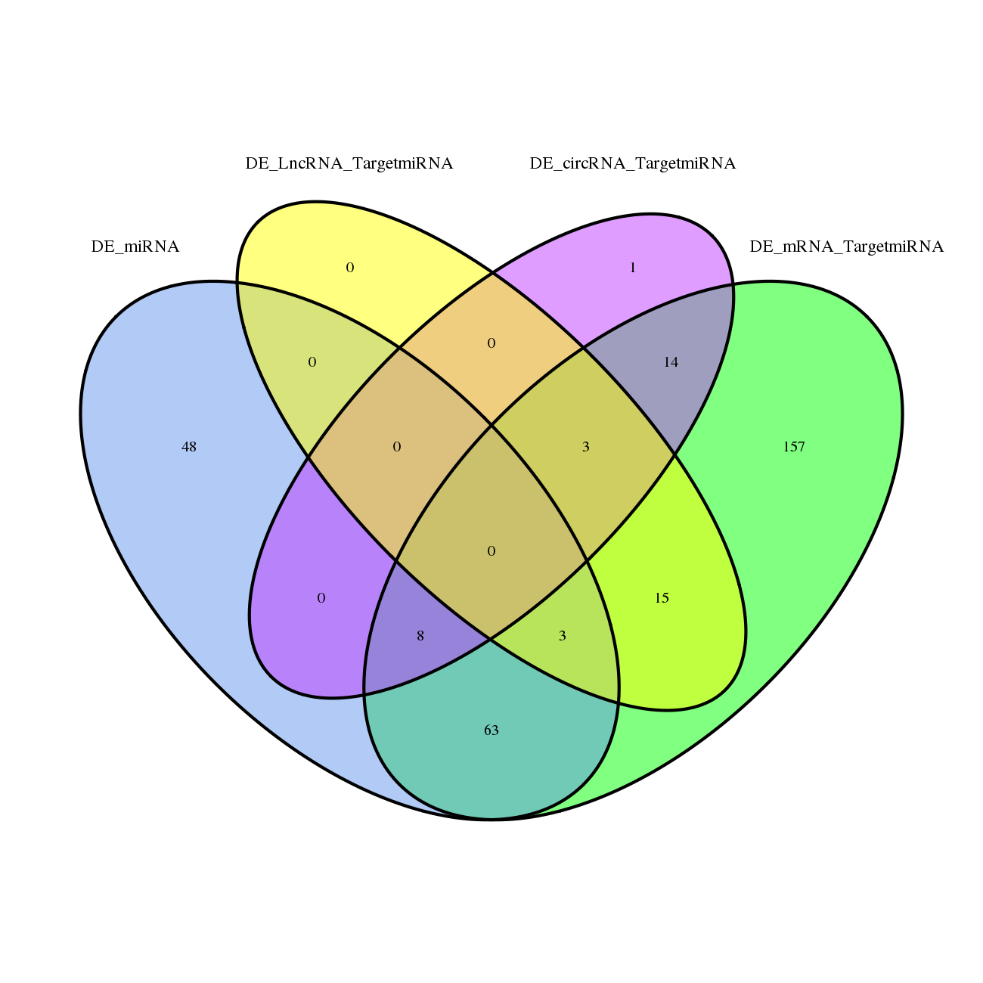
● Pełna analiza bioinformatyczna mRNA, lncRNA, circRNA i miRNA w oddzielnych raportach bioinformatycznych
● Wspólna analiza całej ekspresji RNA w połączonym raporcie, w tym analiza sieci ceRNA.
Zalety serwisu
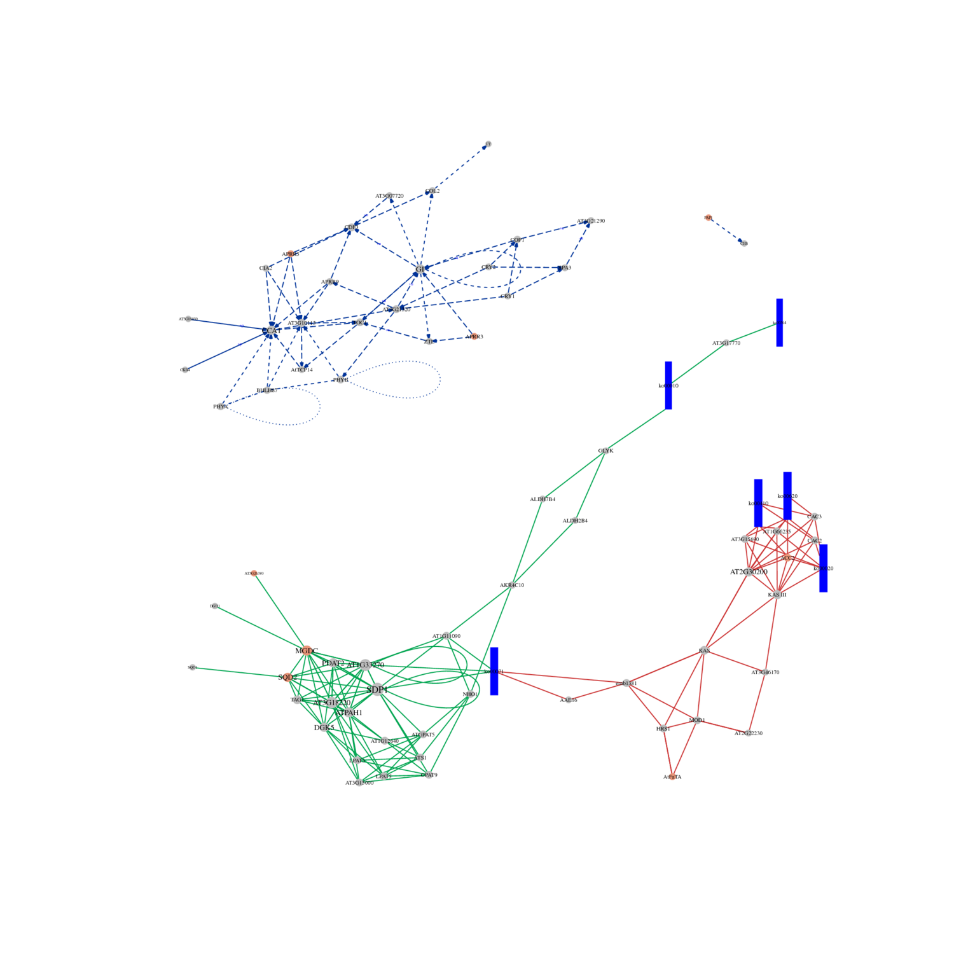
●Dogłębna analiza sieci regulacyjnych: Analiza sieci ceRNA jest możliwa dzięki wspólnemu sekwencjonowaniu mRNA, lncRNA, circRNA i miRNA oraz wyczerpującemu procesowi bioinformatycznemu.
●Obszerna adnotacja: używamy wielu baz danych do funkcjonalnego opisywania genów o zróżnicowanej ekspresji (DEG) i przeprowadzamy odpowiednią analizę wzbogacania, zapewniając wgląd w procesy komórkowe i molekularne leżące u podstaw odpowiedzi transkryptomu.
●Rozległa wiedza specjalistyczna: z doświadczeniem w pomyślnym zamknięciu ponad 2000 projektów całych transkryptomów w różnych dziedzinach badań, nasz zespół wnosi bogate doświadczenie do każdego projektu.
●Rygorystyczna kontrola jakości: wdrażamy podstawowe punkty kontroli na wszystkich etapach, od przygotowania próbek i bibliotek po sekwencjonowanie i bioinformatykę.To skrupulatne monitorowanie zapewnia niezmiennie wysoką jakość wyników.
● Obszerna adnotacja: używamy wielu baz danych do funkcjonalnego opisywania genów o zróżnicowanej ekspresji (DEG) i przeprowadzamy odpowiednią analizę wzbogacania, zapewniając wgląd w procesy komórkowe i molekularne leżące u podstaw odpowiedzi transkryptomu.
●Wsparcie posprzedażowe: Nasze zaangażowanie wykracza poza ukończenie projektu i obejmuje 3-miesięczny okres obsługi posprzedażnej.W tym czasie oferujemy monitorowanie projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby odpowiedzieć na wszelkie pytania związane z wynikami
Przykładowe wymagania i dostawa
| Biblioteka | Strategia sekwencjonowania | Zalecane dane | Kontrola jakości |
| rRNA wyczerpane | Illumina PE150 | 16 giga bajtów | Q30≥85% |
| Wybrany rozmiar | Ilumina SE50 | 10-20 milionów odczytów |
Przykładowe wymagania:
Nukleotydy:
| Stężenie (ng/μl) | Ilość (µg) | Czystość | Uczciwość |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Na żelu widoczne jest ograniczone lub żadne zanieczyszczenie białkiem lub DNA. | Rośliny: RIN≥6,5 Zwierzę: RIN≥7,0 5,0 ≥ 28 S/18 S ≥ 1,0; ograniczone lub żadne wzniesienie linii bazowej |
Zalecana dostawa próbek
Pojemnik:
Probówka wirówkowa o pojemności 2 ml (nie zaleca się stosowania folii aluminiowej)
Przykładowe oznakowanie: Grupa+replika, np. A1, A2, A3;B1, B2, B3... ...
Wysyłka:
1.Suchy lód: Próbki należy zapakować do worków i zakopać w suchym lodzie.
2. Probówki RNAstable: Próbki RNA można suszyć w probówkach do stabilizacji RNA (np. RNAstable®) i przesyłać w temperaturze pokojowej.
Przebieg prac serwisowych

Projekt eksperymentu

Dostawa próbek

Ekstrakcja RNA

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
Bioinformatyka
Przegląd ekspresji RNA
Geny o zróżnicowanej ekspresji
analiza ceRNA
Zapoznaj się z postępami badawczymi, jakie umożliwiły usługi sekwencjonowania całego transkryptomu BMKGene, poprzez wyselekcjonowany zbiór publikacji.
Dai, Y. i in.(2022) „Kompleksowe profile ekspresji mRNA, lncRNA i miRNA w chorobie Kashina-Becka zidentyfikowane za pomocą sekwencjonowania RNA”, Molecular Omics, 18(2), s. 154–166.doi: 10.1039/D1MO00370D.
Liu, N. nan i in.(2022) „Analiza pełnej długości transkryptomów odporności na zimno Apis cerana w górach Changbai w okresie zimowania.”, Gene, 830, s. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ i in.(2022) „Oparte na integracji multiomiki ustalanie priorytetów konkurujących endogennych sieci regulacyjnych RNA w drobnokomórkowym raku płuca: charakterystyka molekularna i kandydaci na leki”, Frontiers in Oncology, 12, s. 1.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. i in.(2022) „Zintegrowana analiza profili ekspresji lncRNA/circRNA-miRNA-mRNA ujawnia nowe spojrzenie na potencjalne mechanizmy reakcji na nicienie root-knot w orzeszkach ziemnych”, BMC Genomics, 23(1), s. 1–12.doi: 10.1186/S12864-022-08470-3/RYSUNKI/7.
Yan, Z. i in.(2022) „Sekwencjonowanie RNA całego transkryptomu uwydatnia mechanizmy molekularne związane z utrzymaniem jakości brokułów po zbiorze za pomocą naświetlania czerwoną diodą LED”, Postharvest Biology and Technology, 188, s.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.