-

Sekwencjonowanie pełnej długości mRNA – Nanopore
Sekwencjonowanie RNA jest nieocenionym narzędziem do kompleksowej analizy transkryptomu.Bez wątpienia tradycyjne sekwencjonowanie z krótkim odczytem osiągnęło tutaj wiele ważnych osiągnięć.Niemniej jednak często napotyka ograniczenia w identyfikacji pełnej długości izoform, oznaczaniu ilościowym i błędach PCR.
Sekwencjonowanie nanoporowe różni się od innych platform do sekwencjonowania tym, że nukleotydy są odczytywane bezpośrednio bez syntezy DNA i generują długi odczyt przy dziesiątkach kilozasad.Umożliwia to bezpośrednie odczytywanie transkrypcji pełnej długości i radzenie sobie z wyzwaniami w badaniach na poziomie izoform.
Platforma:Nanopore PromethION
Biblioteka:cDNA-PCR
-

De novo Sekwencjonowanie transkryptomu pełnej długości - PacBio
De novosekwencjonowanie pełnej długości transkryptomu, znane również jakoDe novoIso-Seq wykorzystuje zalety sekwencera PacBio w zakresie długości odczytu, co umożliwia sekwencjonowanie pełnej długości cząsteczek cDNA bez żadnych przerw.Pozwala to całkowicie uniknąć błędów generowanych na etapach składania transkryptu i konstruuje zestawy jednogenowe z rozdzielczością na poziomie izoform.Te jednogenowe zestawy dostarczają potężnych informacji genetycznych jako „genom referencyjny” na poziomie transkryptomu.Ponadto, w połączeniu z danymi sekwencjonowania nowej generacji, usługa ta umożliwia dokładną kwantyfikację ekspresji na poziomie izoformy.
Platforma: PacBio Sequel IIBiblioteka: biblioteka dzwonków SMRT -

Sekwencjonowanie eukariotycznego mRNA – Illumina
Sekwencjonowanie mRNA umożliwia profilowanie wszystkich mRNA transkrypowanych z komórek w określonych warunkach.Jest to potężna technologia ujawniania profilu ekspresji genów, struktur genów i mechanizmów molekularnych niektórych procesów biologicznych.Do tej pory sekwencjonowanie mRNA było szeroko stosowane w badaniach podstawowych, diagnostyce klinicznej, opracowywaniu leków itp.
Platforma: Illumina NovaSeq 6000
-

Sekwencjonowanie mRNA bez odniesienia – Illumina
Sekwencjonowanie mRNA wykorzystuje technikę sekwencjonowania nowej generacji (NGS) w celu wychwycenia informacyjnego RNA (mRNA) z Eukariontu w określonym czasie, w którym aktywują się pewne specjalne funkcje.Najdłuższy splicing transkryptu nazwano „Unigene” i wykorzystano jako sekwencję referencyjną do późniejszej analizy, co jest skutecznym sposobem badania mechanizmu molekularnego i sieci regulacyjnej gatunku bez odniesienia.
Po złożeniu danych transkryptomu i jednogenowej adnotacji funkcjonalnej
(1) Przeprowadzona zostanie analiza SNP, analiza SSR, przewidywanie CDS i struktura genów.
(2) Przeprowadzona zostanie ocena ilościowa ekspresji jednogenowej w każdej próbce.
(3) Wyrażone w różny sposób jednogeny między próbkami (lub grupami) zostaną odkryte w oparciu o ekspresję jednogenową
(4)Zostaną przeprowadzone klastrowanie, funkcjonalna adnotacja i analiza wzbogacania jednogenów o zróżnicowanej ekspresji
-

Długie sekwencjonowanie bez kodowania – Illumina
Długie niekodujące RNA (lncRNA) to rodzaj cząsteczek RNA o długości przekraczającej 200 nt, które charakteryzują się wyjątkowo niskim potencjałem kodowania.LncRNA, jako kluczowy składnik niekodujących RNA, znajduje się głównie w jądrze i osoczu.Rozwój technologii sekwencjonowania i bioinformatyki umożliwia identyfikację wielu nowych lncRNA i powiązanie ich z funkcjami biologicznymi.Zgromadzone dowody sugerują, że lncRNA jest szeroko zaangażowany w regulację epigenetyczną, regulację transkrypcji i regulację potranskrypcyjną.
-
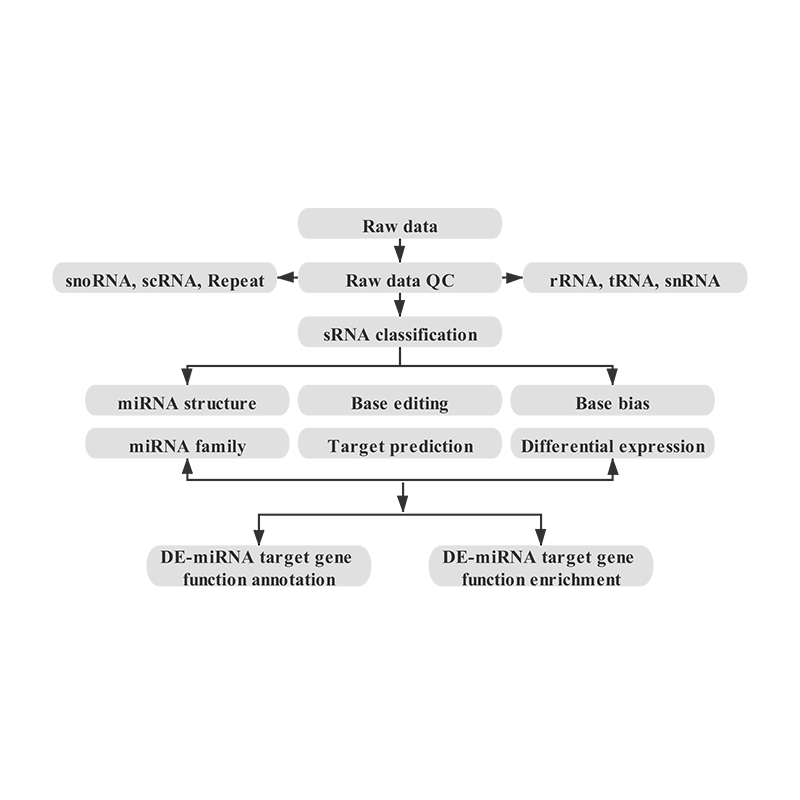
Sekwencjonowanie małych RNA – Illumina
Małe RNA odnosi się do klasy niekodujących cząsteczek RNA, które mają zwykle mniej niż 200 nt długości, w tym mikro RNA (miRNA), małe interferencyjne RNA (siRNA) i RNA oddziałujące z piwi (piRNA).
MikroRNA (miRNA) to klasa endogennych małych RNA o długości około 20-24 nt, które odgrywają różne ważne role regulacyjne w komórkach.miRNA bierze udział w wielu procesach życiowych, które ujawniają specyficzną ekspresję tkankową – specyficzną i fazową – i są wysoce konserwatywne u różnych gatunków.
-
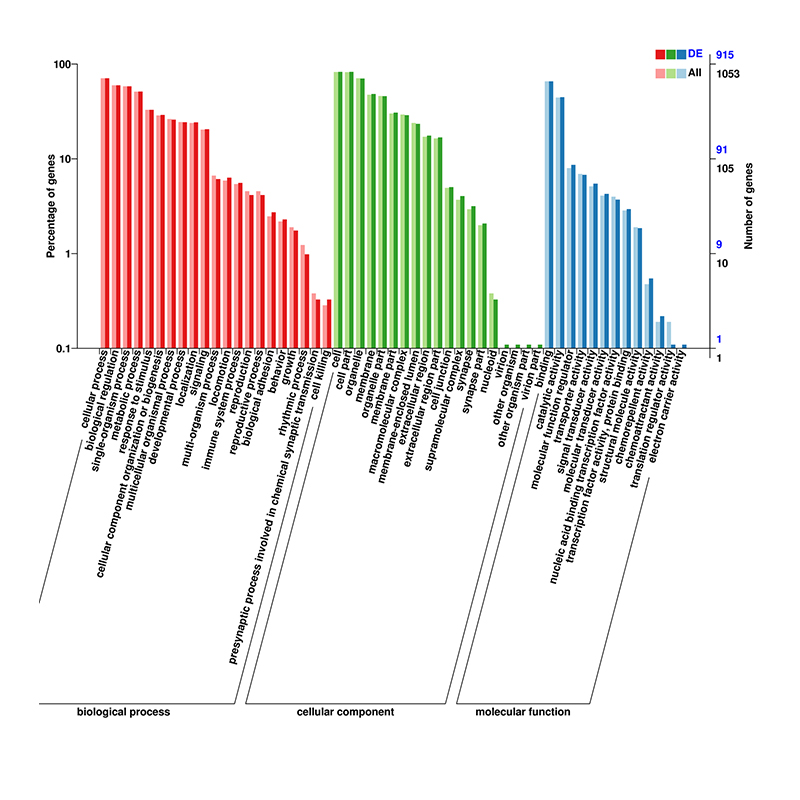
sekwencjonowanie circRNA – Illumina
Sekwencjonowanie całego transkryptomu ma na celu profilowanie wszystkich rodzajów cząsteczek RNA, w tym kodujących (mRNA) i niekodujących RNA (w tym lncRNA, circRNA i miRNA), które są transkrybowane przez określone komórki w określonym czasie.Sekwencjonowanie całego transkryptomu, znane również jako „sekwencjonowanie całkowitego RNA”, ma na celu ujawnienie kompleksowych sieci regulacyjnych na poziomie transkryptomu.Wykorzystując technologię NGS, sekwencje całych produktów transkryptomu są dostępne do analizy ceRNA i analizy RNA wspólnego, co stanowi pierwszy krok w kierunku charakterystyki funkcjonalnej.Ujawnienie sieci regulacyjnej ceRNA opartego na circRNA-miRNA-mRNA.
-
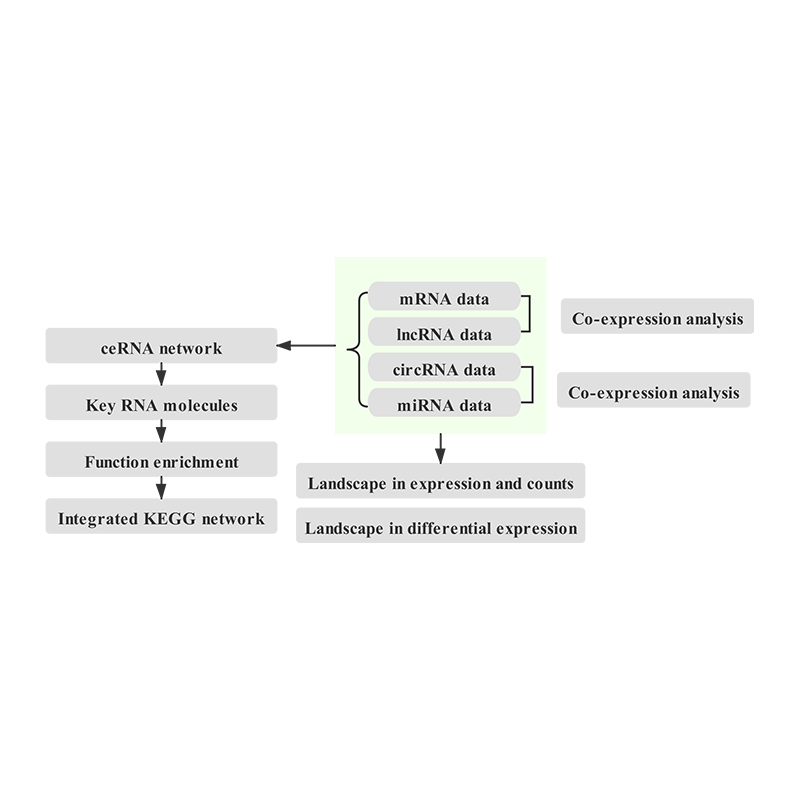
Sekwencjonowanie całego transkryptomu – Illumina
Sekwencjonowanie całego transkryptomu ma na celu profilowanie wszystkich rodzajów cząsteczek RNA, w tym kodujących (mRNA) i niekodujących RNA (w tym lncRNA, circRNA i miRNA), które są transkrybowane przez określone komórki w określonym czasie.Sekwencjonowanie całego transkryptomu, znane również jako „sekwencjonowanie całkowitego RNA”, ma na celu ujawnienie kompleksowych sieci regulacyjnych na poziomie transkryptomu.Wykorzystując technologię NGS, sekwencje całych produktów transkryptomu są dostępne do analizy ceRNA i analizy RNA wspólnego, co stanowi pierwszy krok w kierunku charakterystyki funkcjonalnej.Ujawnienie sieci regulacyjnej ceRNA opartego na circRNA-miRNA-mRNA.
-

Sekwencjonowanie prokariotycznego RNA
Sekwencjonowanie prokariotycznego RNA wykorzystuje sekwencjonowanie nowej generacji (NGS) w celu ujawnienia obecności i ilości RNA w danym momencie poprzez analizę zmieniającego się transkryptomu komórkowego.Prokariotyczne sekwencjonowanie RNA naszej firmy ma na celu w szczególności prokariota z genomami referencyjnymi, zapewniając profilowanie transkryptomu, analizę struktury genów itp. Jest szeroko stosowane w podstawowych badaniach naukowych, badaniach i rozwoju leków i nie tylko.
Platforma: Illumina NovaSeq 6000
-

Sekwencjonowanie metatranskryptomu
Sekwencjonowanie metatranskryptomu identyfikuje ekspresję genów drobnoustrojów (zarówno eukariontów, jak i prokariotów) w środowisku naturalnym (tj. gleba, woda, morze, kał i jelita). gatunków, analiza funkcjonalnego wzbogacenia genów o różnej ekspresji i nie tylko.
Platforma: Illumina NovaSeq 6000











