Sekwencjonowanie jednojądrowego RNA
Zasada techniczna
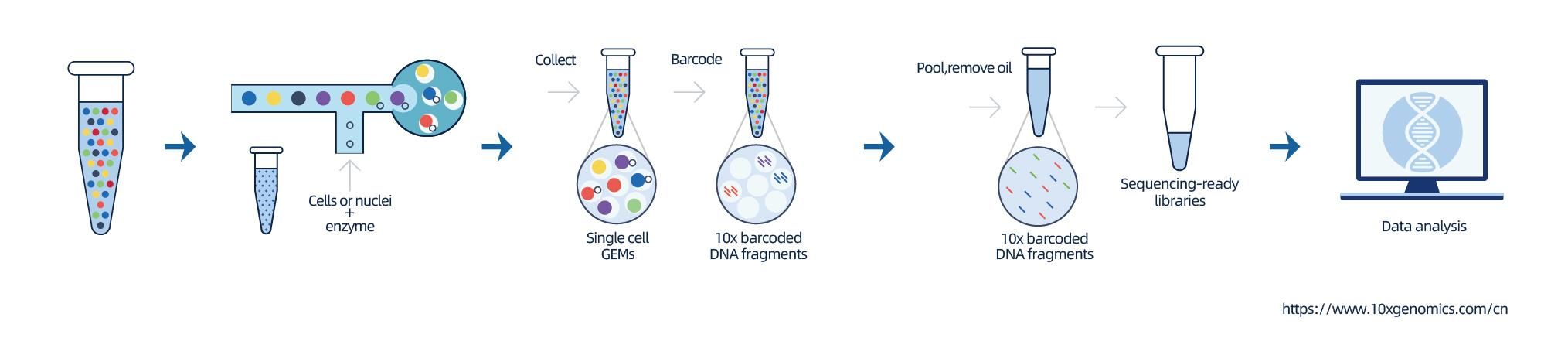
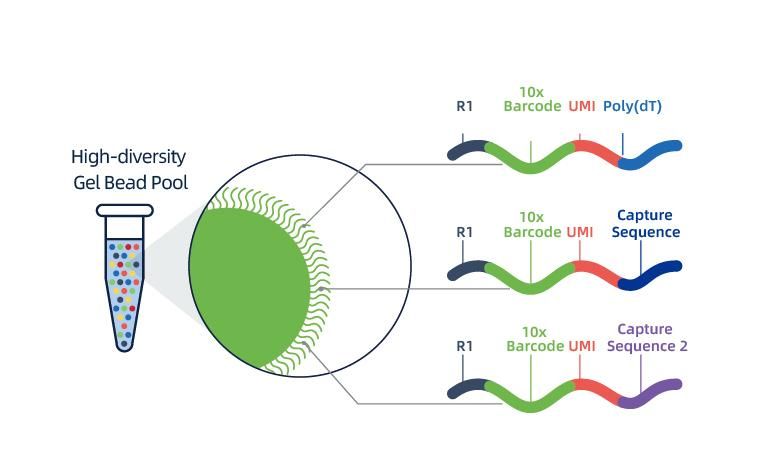
Izolację jąder uzyskuje się za pomocą 10× Genomics ChromiumTM, który składa się z ośmiokanałowego układu mikroprzepływowego z podwójnym przejściem.W tym systemie kulki żelowe z kodami kreskowymi i podkładem, enzymami i pojedynczym jądrem są zamknięte w kropli oleju o wielkości nanolitra, tworząc żelową kulkę w emulsji (GEM).Po utworzeniu GEM w każdym GEM przeprowadzana jest liza komórek i uwalnianie kodów kreskowych.mRNA poddaje się odwrotnej transkrypcji na cząsteczki cDNA z 10-krotnymi kodami kreskowymi i UMI, które następnie podlegają standardowej konstrukcji biblioteki sekwencjonowania.
Tkanka nieodpowiednia do przygotowania zawiesiny pojedynczych komórek
| Komórka/Tkanka | Powód |
| Nieświeża zamrożona tkanka | Nie można uzyskać nowych lub długo zapisanych organizacji |
| Komórka mięśniowa, megakariocyt, tłuszcz… | Średnica kuwety jest zbyt duża, aby wejść do instrumentu |
| Wątroba… | Zbyt kruche, aby je rozbić, niezdolne do rozróżnienia pojedynczych komórek |
| Komórka neuronowa, mózg… | Bardziej wrażliwy, łatwy do zestresowania, zmieni wyniki sekwencjonowania |
| Trzustka, tarczyca… | Bogaty w enzymy endogenne, wpływające na wytwarzanie zawiesiny pojedynczych komórek |
Pojedyncze jądro vs pojedyncza komórka
| Jednojądrowe | Pojedyncza komórka |
| Nieograniczona średnica ogniwa | Średnica komórki: 10-40 µm |
| Materiałem może być zamrożona tkanka | Materiał musi być świeżą chusteczką |
| Niski stres zamrożonych komórek | Traktowanie enzymami może powodować reakcję stresową komórek |
| Nie ma potrzeby usuwania czerwonych krwinek | Należy usunąć czerwone krwinki |
| Jądrowy wyraża bioinformację | Cała komórka wyraża bioinformację |
Specyfikacje usług
| Biblioteka | Strategia sekwencjonowania | Ilość danych | Przykładowe wymagania | Tkanka |
| 10 × biblioteka pojedynczych jąder Genomics | 10x Genomika -Illumina PE150 | 100 000 odczytów/komórkę ok.100-200 Gb | Numer komórki: >2×105 Stężenie komórekprzy 700-1200 komórek/µl | ≥ 200 mg |
Aby uzyskać więcej informacji na temat wskazówek dotyczących przygotowywania próbek i przebiegu usług, skontaktuj się z: aEkspert BMKGENE
Przebieg prac serwisowych

Projekt eksperymentu

Dostawa próbek
Izolacja jąder

Budowa biblioteki

Sekwencjonowanie

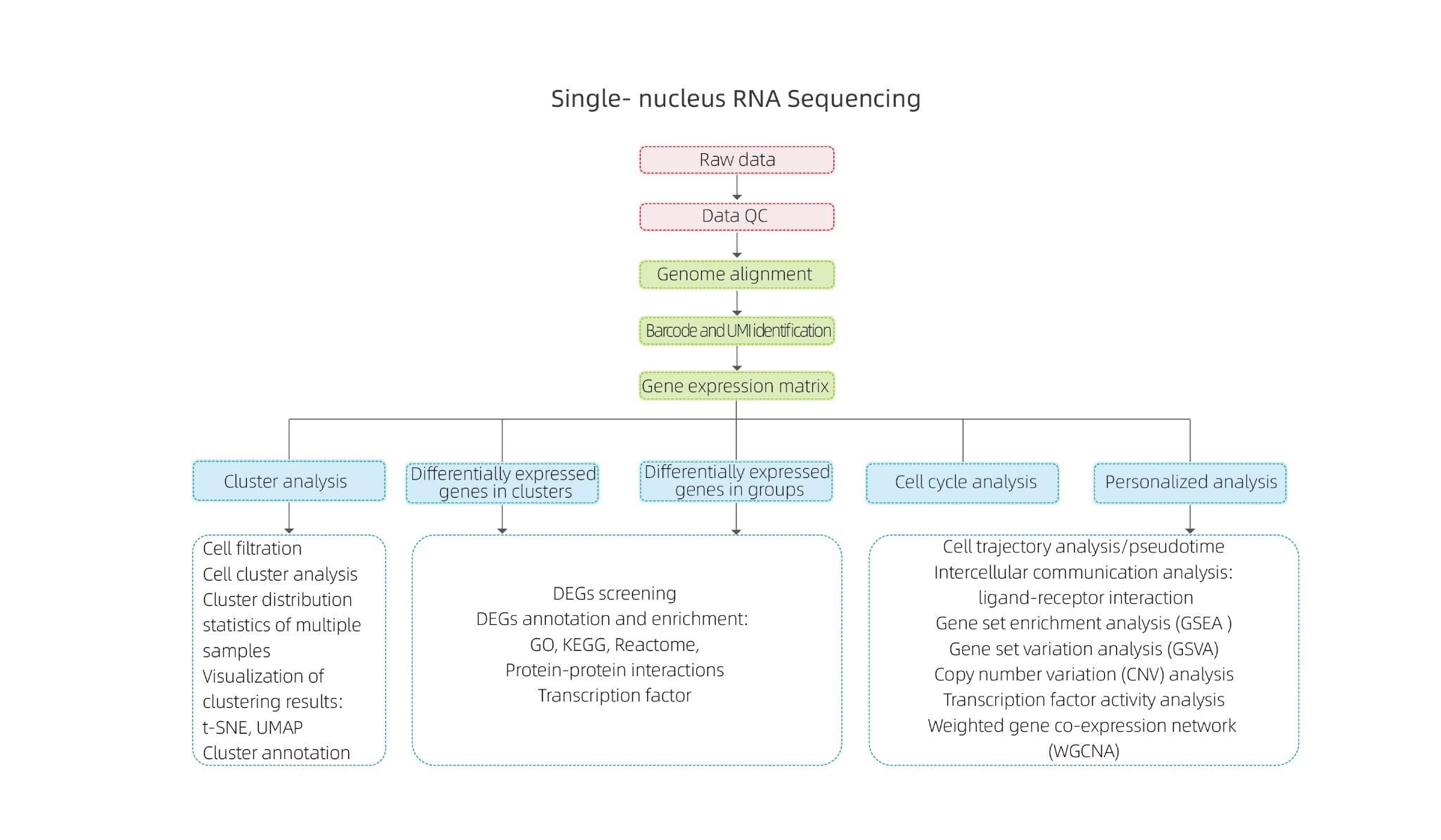
Analiza danych

Usługi posprzedażowe
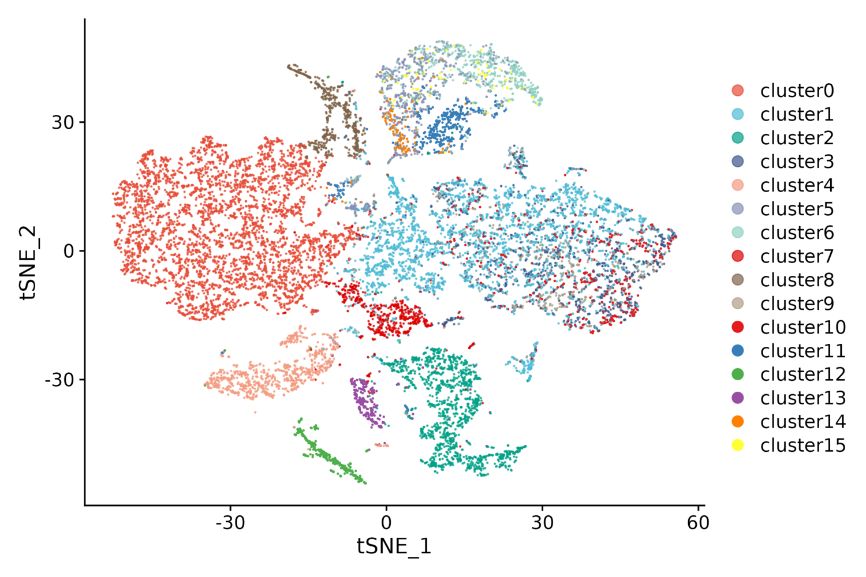
1. Grupowanie punktowe
2. Mapa cieplna grupowania wyrażeń markerów
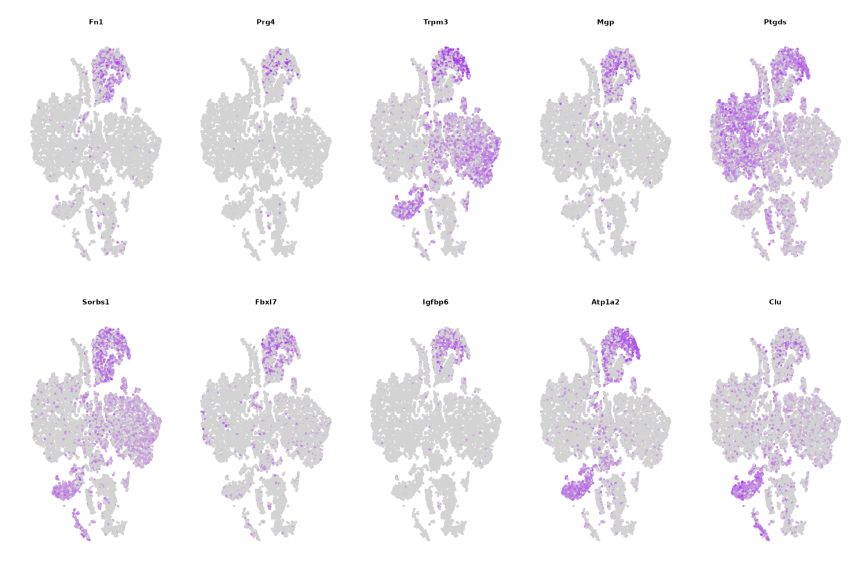
 3.Rozmieszczenie genu Makera w różnych klastrach
3.Rozmieszczenie genu Makera w różnych klastrach
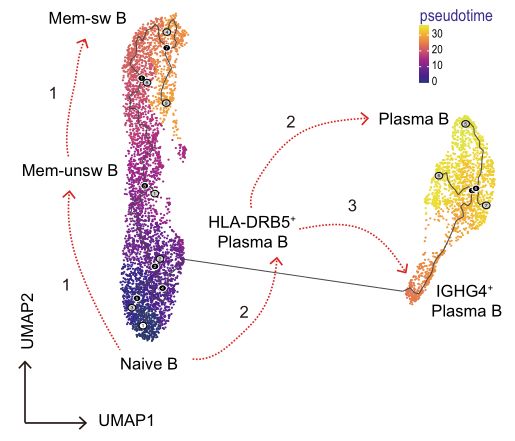
4.Analiza trajektorii komórki/pseudoczas