-

Porównawcze analizy genomu podkreślają ekspansję genomu za pośrednictwem transpozonów i ewolucyjną architekturę trójwymiarowego fałdowania genomu bawełny
EWOLUCJA GENOMEMU Porównawcze analizy genomu podkreślają ekspansję genomu za pośrednictwem transpozonów i ewolucyjną architekturę trójwymiarowego fałdowania genomu w bawełnie Sekwencjonowanie nanoporów |Hi-C |PacBio...Czytaj więcej -
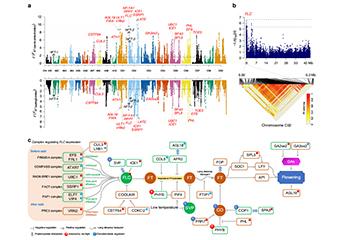
W nasionach rzepaku metodą GWAS zidentyfikowano geny związane z tolerancją na stres, zawartością oleju, jakością nasion i poprawą ekotypu
GWAS Tytuł: Ponowne sekwencjonowanie całego genomu ujawnia pochodzenie Brassica napus i loci genetyczne zaangażowane w jego doskonalenie Dziennik: Nature Communications NGS |WGS |Ponowna sekwencja |GWAS |Transkrypt |Sekwencja RNA |Brassica napus |Ewolucja |Domowy...Czytaj więcej -
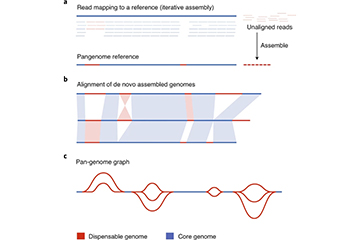
Badania pangenomu dostarczają głębokiego i pełnego obrazu genetycznego gatunku
EWOLUCJA GENOMEMU, PANGENOME Co to jest pangenom?Zgromadzone dowody pokazują, że różnice między różnymi szczepami gatunku mogą być ogromne.Jeden genom to zdecydowanie za mało, aby uzyskać pełny obraz informacji genetycznej pojedynczego gatunku.T...Czytaj więcej




