
Sekwencjonowanie metagenomiczne -NGS
Zalety serwisu
● Brak izolacji i hodowli w celu profilowania społeczności drobnoustrojów
● Wysoka rozdzielczość w wykrywaniu gatunków o niskiej liczebności w próbkach środowiskowych
● Idea „meta-” integruje wszystkie cechy biologiczne na poziomie funkcjonalnym, poziomie gatunku i poziomie genu, co odzwierciedla dynamiczne spojrzenie bliższe rzeczywistości.
● BMK gromadzi ogromne doświadczenie w zakresie różnorodnych typów próbek, przetwarzając ponad 10 000 próbek.
Specyfikacje usług
| Platforma | Sekwencjonowanie | Zalecane dane | Czas realizacji |
| Platforma Illumina NovaSeq | PE150 | 6G/10G/20G | 45 dni roboczych |
Analizy bioinformatyczne
● Kontrola jakości surowych danych
● Składanie metagenomu
● Niezbędny zestaw genów i adnotacja
● Analiza różnorodności gatunkowej
● Analiza różnorodności funkcji genetycznych
● Analiza międzygrupowa
● Analiza asocjacyjna względem czynników eksperymentalnych
Przykładowe wymagania i dostawa
Przykładowe wymagania:
DlaEkstrakty DNA:
| Typ próbki | Kwota | Stężenie | Czystość |
| Ekstrakty DNA | > 30 ng | > 1 ng/µl | OD260/280= 1,6-2,5 |
Dla próbek środowiskowych:
| Typ próbki | Zalecana procedura pobierania próbek |
| Gleba | Ilość próbki: ok.5 g;Pozostałą zwiędłą substancję należy usunąć z powierzchni;Zmiel duże kawałki i przepuść przez filtr 2 mm;Próbki w sterylnej probówce EP lub cyrotubie do rezerwacji. |
| Kał | Ilość próbki: ok.5 g;Zbieraj i rozdzielaj próbki do sterylnej probówki EP lub krioprobówki w celu rezerwacji. |
| Treść jelitowa | Próbki należy przetwarzać w warunkach aseptycznych.Przemyć pobraną tkankę PBS;Odwirować PBS i zebrać osad do probówek EP. |
| Osad | Ilość próbki: ok.5 g;Zbierz i podziel na porcje próbkę osadu do sterylnej probówki EP lub krioprobówki w celu rezerwacji |
| Zbiornik wodny | W przypadku próbki zawierającej ograniczoną ilość drobnoustrojów, takiej jak woda z kranu, woda ze studni itp., zebrać co najmniej 1 l wody i przepuścić przez filtr 0,22 μm, aby wzbogacić mikroorganizmy na membranie.Przechowywać membranę w sterylnej probówce. |
| Skóra | Ostrożnie zeskrob powierzchnię skóry sterylnym wacikiem lub ostrzem chirurgicznym i umieść go w sterylnej probówce. |
Zalecana dostawa próbek
Zamrozić próbki w ciekłym azocie na 3-4 godziny i przechowywać w ciekłym azocie lub w -80 stopniach z rezerwacją długoterminową.Wymagana jest wysyłka próbek z suchym lodem.
Przebieg prac serwisowych

Dostawa próbek

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
1.Histogram: rozmieszczenie gatunków
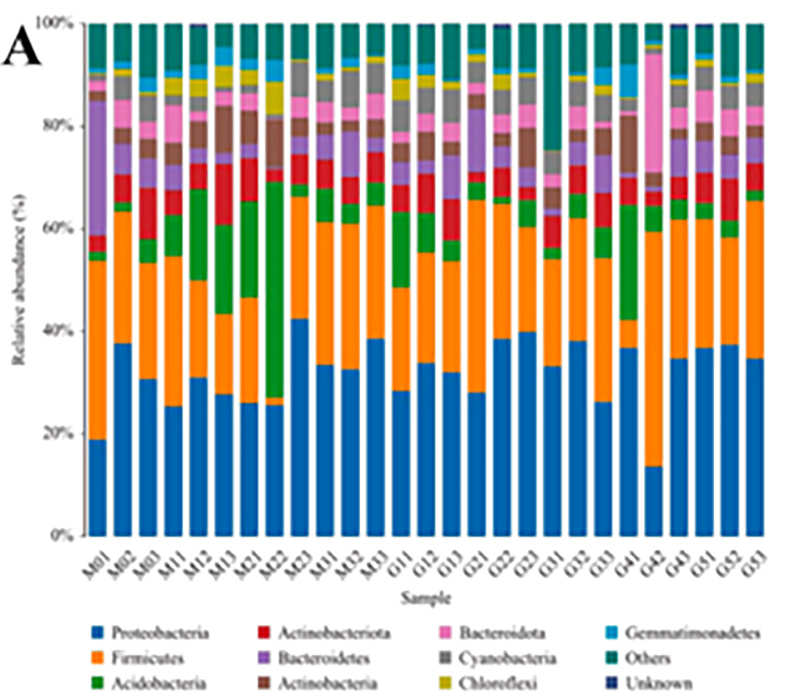
2. Geny funkcjonalne przypisane do szlaków metabolicznych KEGG
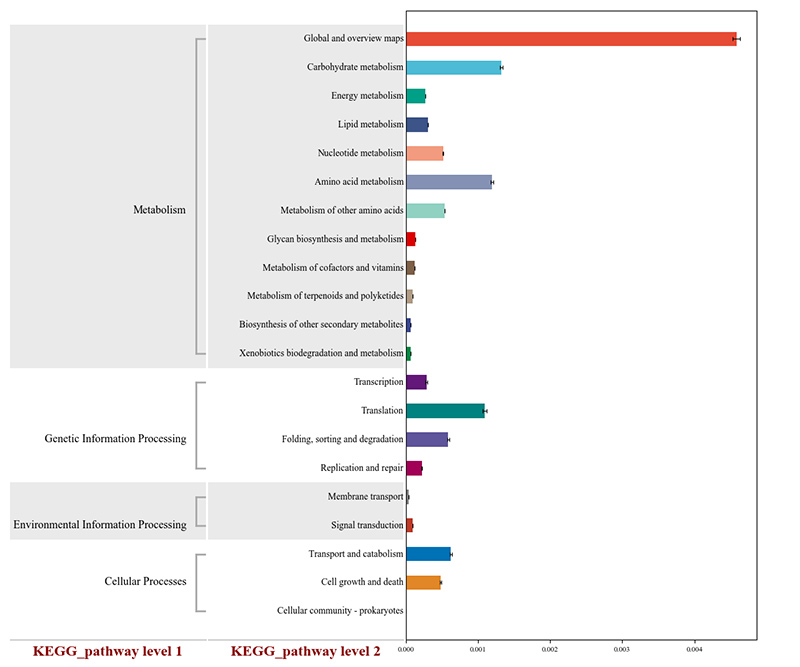
3. Mapa cieplna: Funkcje różnicowe oparte na względnej obfitości genów 4.Circos genów oporności na antybiotyki CARD
4.Circos genów oporności na antybiotyki CARD
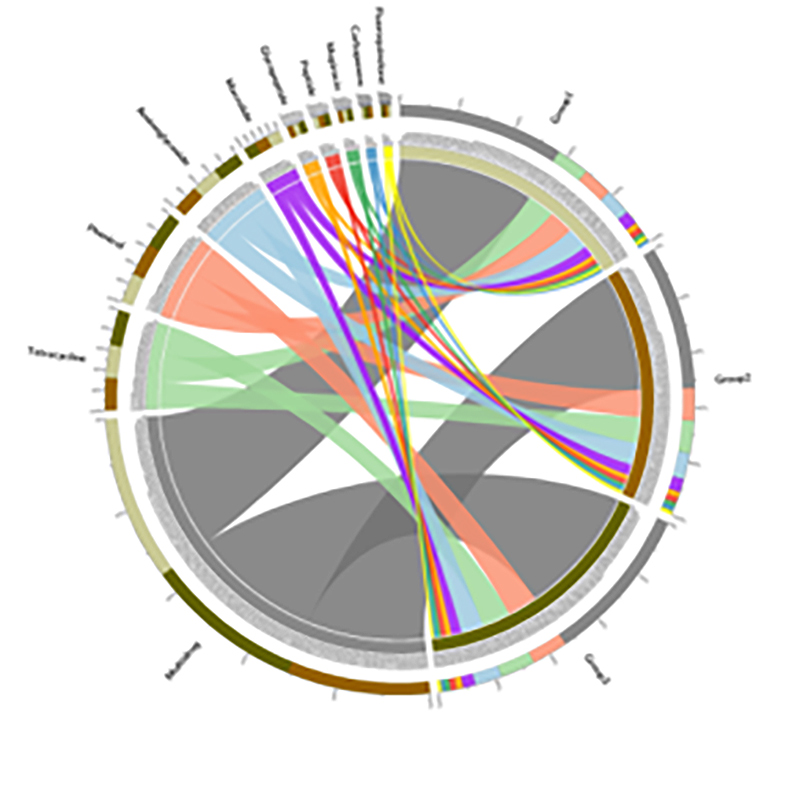
Sprawa BMK
Występowanie genów oporności na antybiotyki i patogenów bakteryjnych wzdłuż kontinuum korzeni gleby i namorzynów
Opublikowany:Dziennik materiałów niebezpiecznych, 2021
Strategia sekwencjonowania:
Materiały: Ekstrakty DNA czterech fragmentów próbek związanych z korzeniami namorzynów: gleby nieobsadzonej, ryzosfery, episfery i przedziałów endosfery
Platforma: Illumina HiSeq 2500
Cele: Metagenom
Region V3-V4 genu 16S rRNA
Kluczowe wyniki
Aby zbadać rozprzestrzenianie się genów oporności na antybiotyki (ARG) z gleby do roślin, przeprowadzono sekwencjonowanie metagenomiczne i profilowanie oparte na kodach kreskowych kontinuum gleba-korzeń sadzonek namorzynów.Dane metagenomiczne wykazały, że 91,4% genów oporności na antybiotyki powszechnie identyfikowano we wszystkich czterech wspomnianych powyżej przedziałach gleby, co wykazuje tendencję ciągłą.Sekwencjonowanie amplikonu 16S rRNA wygenerowało 29 285 sekwencji reprezentujących 346 gatunków.W połączeniu z profilowaniem gatunkowym poprzez sekwencjonowanie amplikonu stwierdzono, że rozprzestrzenianie się jest niezależne od mikroflory związanej z korzeniami, jednakże można je ułatwić dzięki mobilności elementów genetycznych.W badaniu tym zidentyfikowano przepływ ARG i patogenów z gleby do roślin poprzez wzajemnie połączone kontinuum gleba-korzeń.
Odniesienie
Wang, C., Hu, R., Strong, PJ, Zhuang, W. i Shu, L. .(2020).Występowanie genów oporności na antybiotyki i patogenów bakteryjnych wzdłuż kontinuum korzeni gleby i namorzynów.Dziennik materiałów niebezpiecznych, 408, 124985.











