Sekwencjonowanie całego egzomu człowieka
Zalety serwisu
● Ukierunkowany region kodujący białko: wychwytując i sekwencjonując region kodujący białko, hWES wykorzystuje się do ujawnienia wariantów związanych ze strukturą białka;
● Wysoka dokładność: dzięki dużej głębokości sekwencjonowania hWES ułatwia wykrywanie powszechnych wariantów i rzadkich wariantów z częstotliwościami niższymi niż 1%;
• Opłacalność: hWES pozwala uzyskać około 85% mutacji chorób ludzkich z 1% ludzkiego genomu;
● Pięć rygorystycznych procedur kontroli jakości obejmujących cały proces z gwarancją Q30>85%.
Przykładowe specyfikacje
| Platforma
| Biblioteka
| Strategia przechwytywania egzonów
| Zalecana strategia sekwencjonowania
|
|
Platforma Illumina NovaSeq
| PE150 | Agilent SureSelect Human All Exon V6 Panel hybrydowy IDT xGen Exome V2 | 5 GB 10 Gb |
Przykładowe wymagania
| Typ próbki
| Kwota(Qubit®)
| Tom
| Stężenie
| Czystość (NanoDrop™) |
|
Genomowe DNA
| ≥ 300 ng | ≥ 15 µl | ≥ 20 ng/µl | OD260/280=1,8-2,0 bez degradacji i bez zanieczyszczeń
|
Zalecana głębokość sekwencjonowania
W przypadku zaburzeń mendlowskich/rzadkich chorób: efektywna głębokość sekwencjonowania powyżej 50×
W przypadku próbek nowotworu: efektywna głębokość sekwencjonowania powyżej 100×
Bioinformatyka


Przebieg prac serwisowych

Dostawa próbek

Ekstrakcja DNA

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Dostarczanie danych

Usługi posprzedażowe
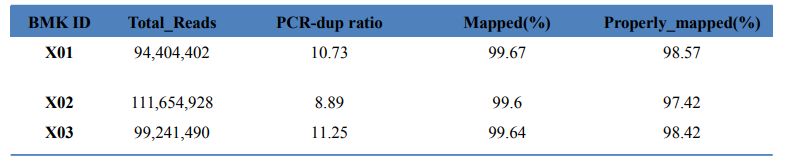
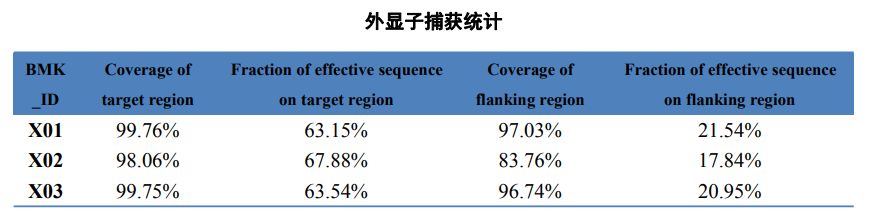
1. Statystyka wyrównania
Tabela 1 Statystyka wyniku mapy
Tabela 2 Statystyki wychwytywania egzomu
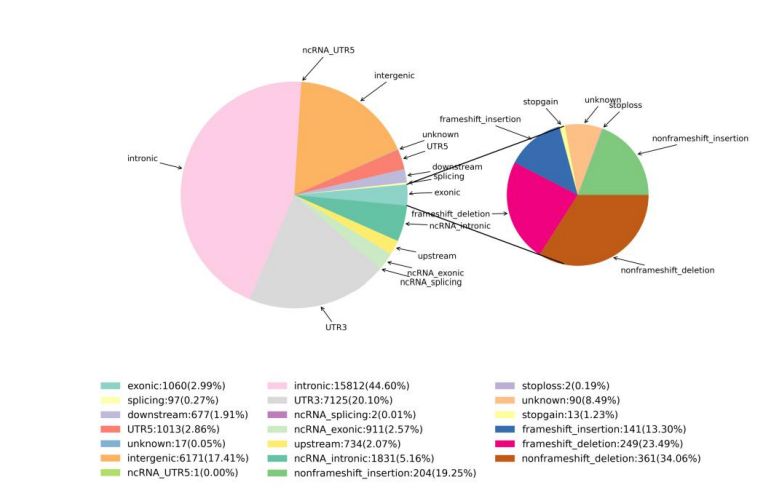
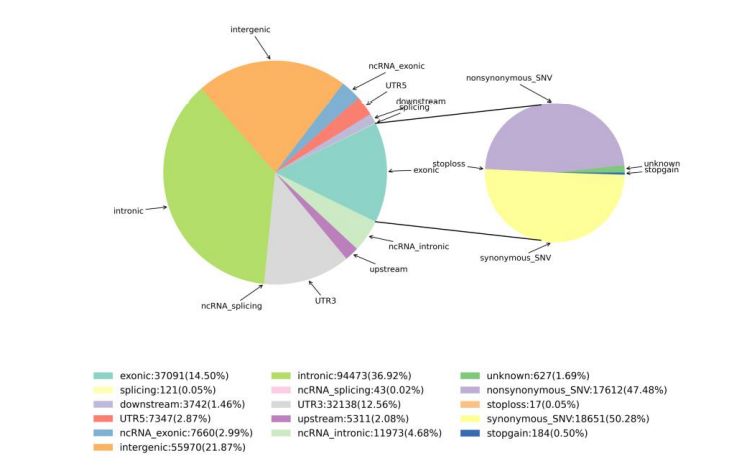
2. Wykrywanie zmian
Rysunek 1 Statystyki SNV i InDel
3.Zaawansowana analiza
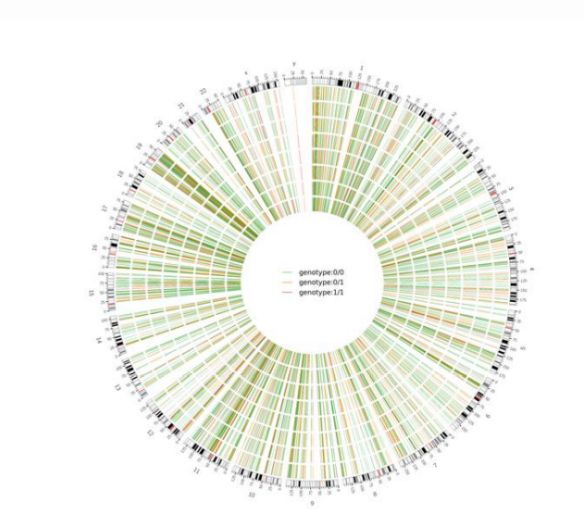
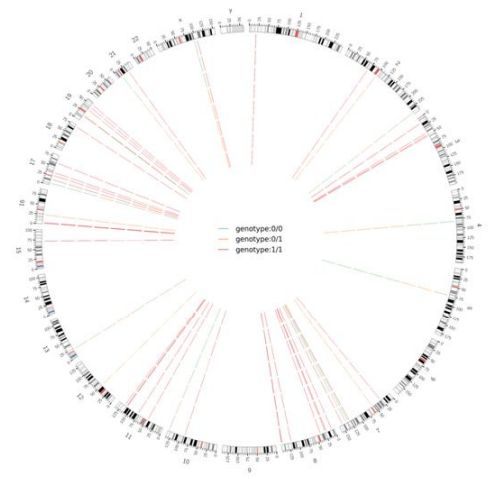
Rysunek 2 Wykres Circos przedstawiający szkodliwe SNV i InDel dla całego genomu
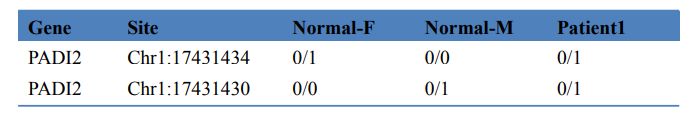
Tabela 3. Badania przesiewowe genów chorobotwórczych