Liten RNA-sekvensering-Illumina
Egenskaper
● Størrelsesvalg av RNA før bibliotekklargjøring
● Bioinformatisk analyse sentrert rundt miRNA-prediksjon og deres mål
Tjenestefordeler
●Omfattende bioinformatikkanalyse:muliggjør identifisering av både kjente og nye miRNA-er, identifisering av miRNA-mål og tilsvarende funksjonell merknad og berikelse med flere databaser (KEGG, GO)
●Streng kvalitetskontroll: Vi implementerer kjernekontrollpunkter på tvers av alle stadier, fra prøve- og bibliotekforberedelse til sekvensering og bioinformatikk.Denne grundige overvåkingen sikrer levering av konsekvent høykvalitetsresultater.
●Support etter salg: Vårt engasjement strekker seg utover prosjektgjennomføring med en 3-måneders ettersalgsserviceperiode.I løpet av denne tiden tilbyr vi prosjektoppfølging, feilsøkingshjelp og spørsmål og svar for å løse eventuelle spørsmål knyttet til resultatene.
●Omfattende kompetanse: Med en merittliste for vellykket lukking av flere sRNA-prosjekter som dekker over 100 arter i ulike forskningsdomener, bringer teamet vårt et vell av erfaring til hvert prosjekt.
Prøvekrav og levering
| Bibliotek | Plattform | Anbefalte data | Data QC |
| Størrelse valgt | Illumina SE50 | 10M-20M avlesninger | Q30≥85 % |
Eksempelkrav:
Nukleotider:
| Kons.(ng/μl) | Mengde (μg) | Renhet | Integritet |
| ≥ 80 | ≥ 0,5 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Begrenset eller ingen protein- eller DNA-kontaminering vist på gel. | RIN≥6,5; 5,0≥28S/18S≥1,0; begrenset eller ingen grunnlinjehøyde |
● Planter:
Rot, stilk eller kronblad: 450 mg
Blad eller frø: 300 mg
Frukt: 1,2 g
● Dyr:
Hjerte eller tarm: 450 mg
Innvoller eller hjerne: 240 mg
Muskel: 600 mg
Bein, hår eller hud: 1,5 g
● Leddyr:
Insekter: 9g
Krepsdyr: 450 mg
● Fullblod: 2 rør
● Celler: 106 celler
● Serum og plasma:6 ml
Anbefalt prøvelevering
Container:
2 ml sentrifugerør (tinnfolie anbefales ikke)
Prøvemerking: Gruppe+replikat f.eks. A1, A2, A3;B1, B2, B3... ...
Forsendelse:
1.Tørris: Prøver må pakkes i poser og begraves i tørris.
2.RNAstabile rør: RNA-prøver kan tørkes i RNA-stabiliseringsrør (f.eks. RNAstable®) og sendes i romtemperatur.
Servicearbeidsflyt

Eksperimentdesign

Prøvelevering

RNA-ekstraksjon

Bygging av bibliotek

Sekvensering

Dataanalyse

Ettersalgstjenester
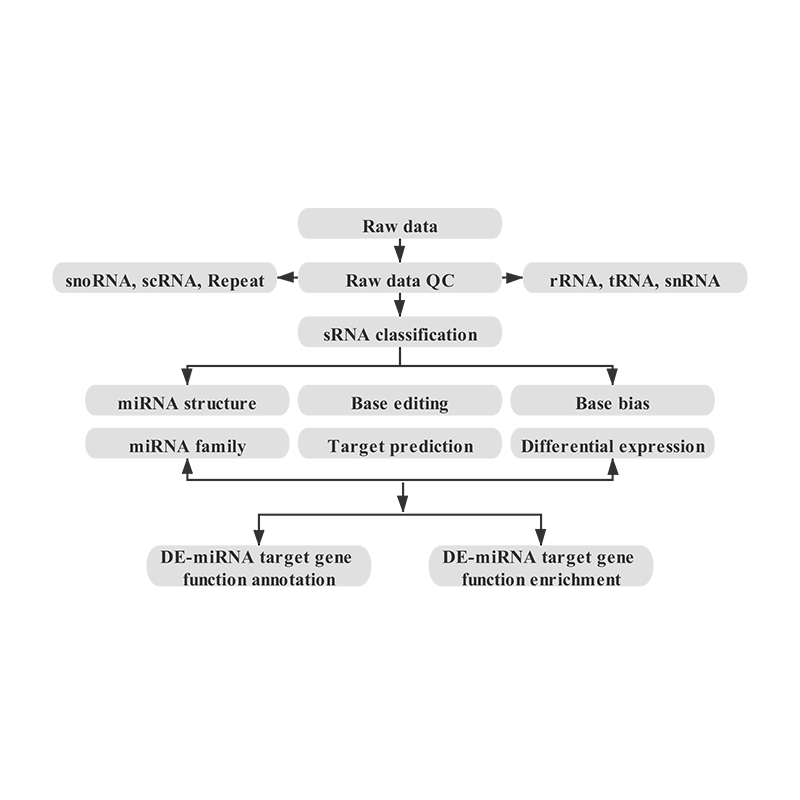
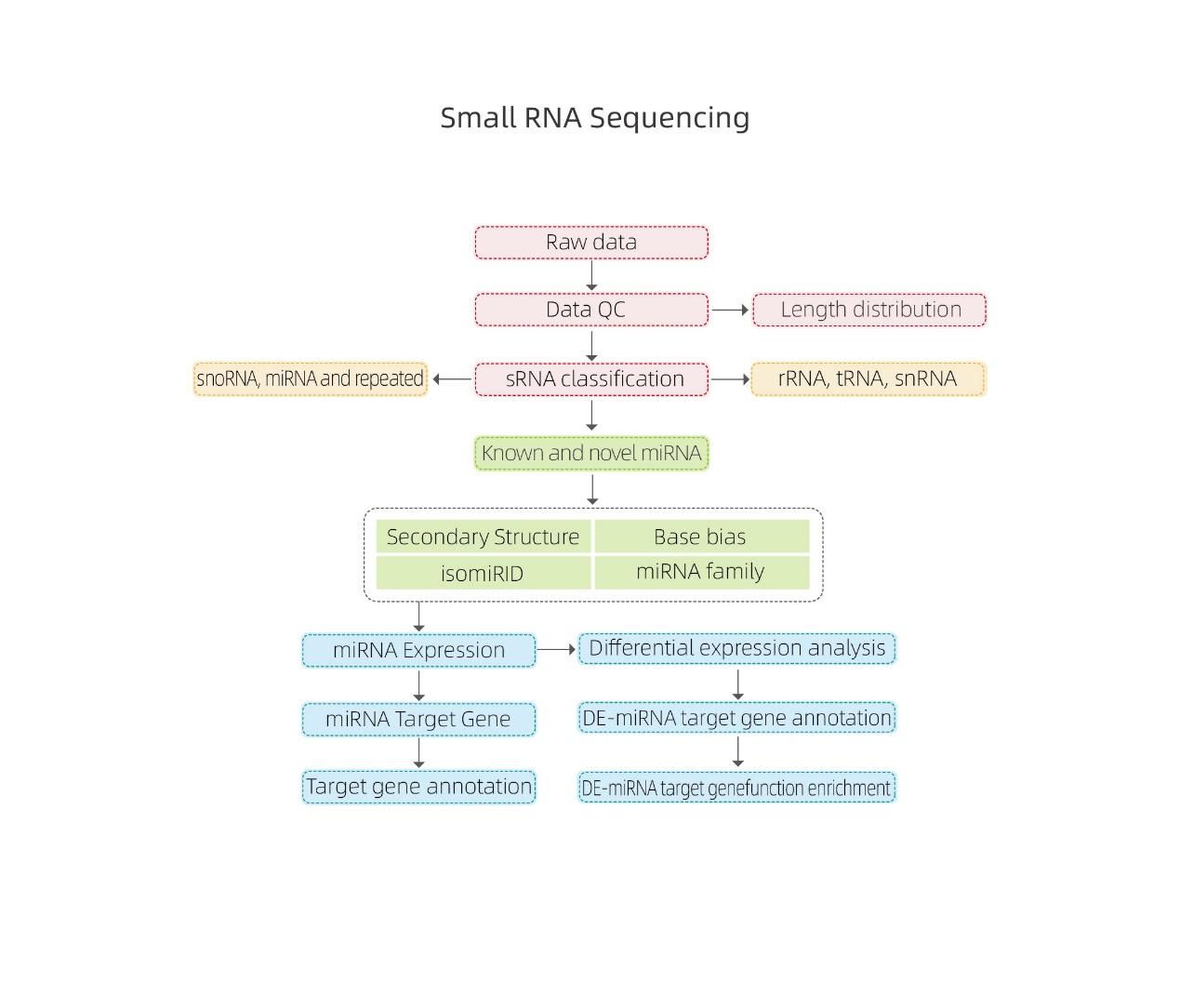
Bioinformatikk
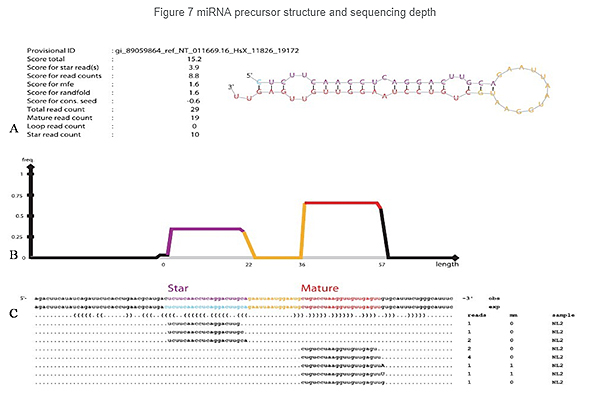
Identifikasjon av miRNA: struktur og dybde
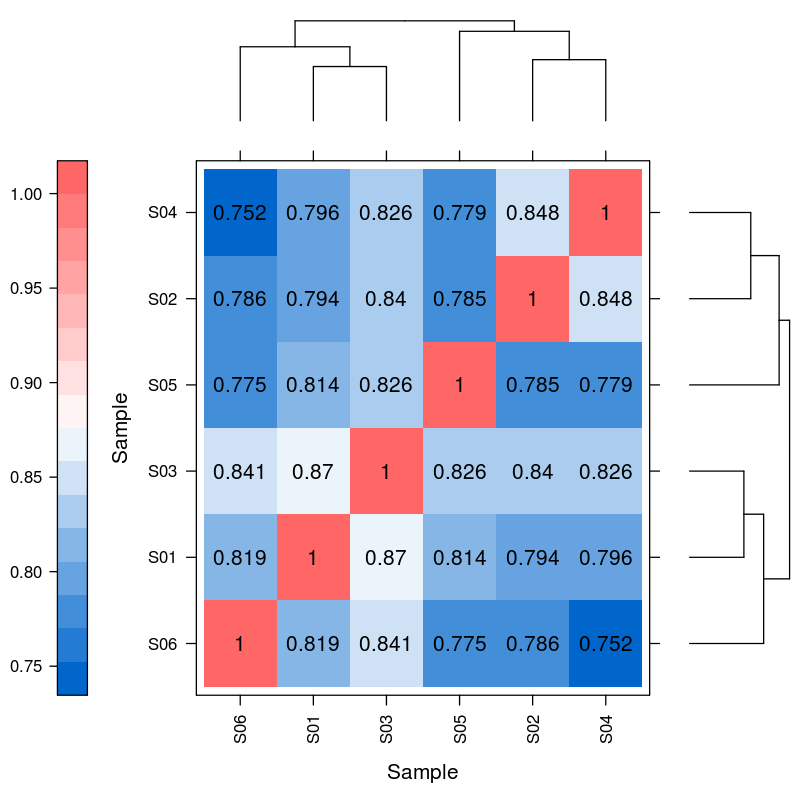
Differensielt uttrykk for miRNA - hierarkisk clustering
Funksjonell annotering av mål for differensielt uttrykte miRNAer
Utforsk forskningsfremskritt tilrettelagt av BMKGene sine sRNA-sekvenseringstjenester gjennom en kuratert samling av publikasjoner.
Chen, H. et al.(2023) 'Virale infeksjoner hemmer saponinbiosyntese og fotosyntese i Panax notoginseng', Plant Physiology and Biochemistry, 203, s.108038. doi: 10.1016/J.PLAPHY.2023.108038.
Li, H. et al.(2023) 'Anlegget FYVE-domeneholdig protein FREE1 assosieres med mikroprosessorkomponenter for å undertrykke miRNA-biogenese', rapporterer EMBO, 24(1).doi: 10.15252/EMBR.202255037/SUPPL_FILE/EMBR202255037-SUP-0004-SDATAFIG4.TIF.
Yu, J. et al.(2023) 'MicroRNA Ame-Bantam-3p kontrollerer utviklingen av larvepuppe ved å målrette mot flere epidermale vekstfaktorlignende domener 8-genet (megf8) i honningbien, Apis mellifera', International Journal of Molecular Sciences, 24(6), s. .5726. doi: 10.3390/IJMS24065726/S1.
Zhang, M. et al.(2018) 'Integrert analyse av MiRNA og gener assosiert med kjøttkvalitet avslører at Gga-MiR-140-5p påvirker intramuskulær fettavsetning hos kyllinger', Cellular Physiology and Biochemistry, 46(6), s. 2421–2433.doi: 10.1159/000489649.