Hele transcriptoomsequencing – Illumina
Functies
● Dubbele bibliotheek om het volledige transcriptoom te sequencen: rRNA-uitputting gevolgd door voorbereiding van de PE150-bibliotheek en grootteselectie gevolgd door voorbereiding van de SE50-bibliotheek
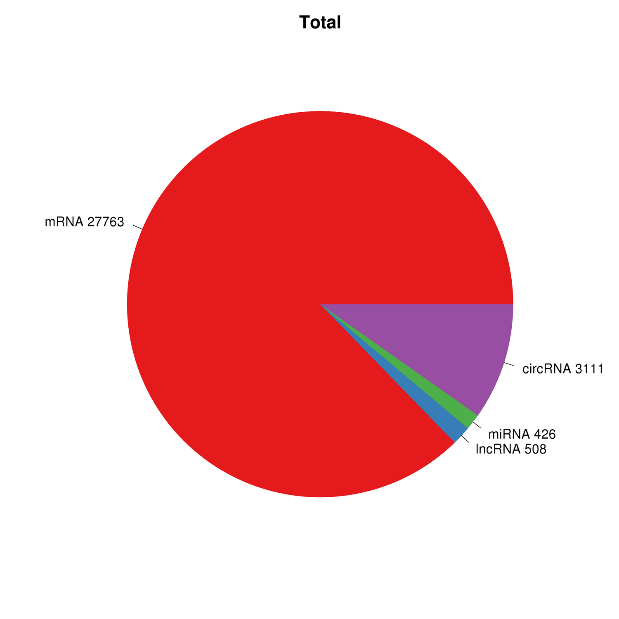
● Volledige bio-informatica-analyse van mRNA, lncRNA, circRNA en miRNA in afzonderlijke bio-informaticarapporten
● Gezamenlijke analyse van alle RNA-expressie in gecombineerd rapport, inclusief analyse van ceRNA-netwerken.
Servicevoordelen
●Diepgaande analyse van regelgevende netwerken: ceRNA-netwerkanalyse wordt mogelijk gemaakt door de gezamenlijke sequencing van mRNA, lncRNA, circRNA en miRNA en door een uitgebreide bio-informatische workflow.
●Uitgebreide annotatie: we gebruiken meerdere databases om de differentieel tot expressie gebrachte genen (DEG's) functioneel te annoteren en de overeenkomstige verrijkingsanalyse uit te voeren, waardoor inzicht wordt verkregen in de cellulaire en moleculaire processen die ten grondslag liggen aan de transcriptoomrespons.
●Uitgebreide expertise: met een track record van het succesvol afsluiten van meer dan 2000 volledige transcriptoomprojecten in verschillende onderzoeksdomeinen, brengt ons team een schat aan ervaring mee naar elk project.
●Strenge kwaliteitscontrole: we implementeren kerncontrolepunten in alle stadia, van monster- en bibliotheekvoorbereiding tot sequencing en bio-informatica.Deze nauwgezette monitoring garandeert de levering van resultaten van consistent hoge kwaliteit.
● Uitgebreide annotatie: we gebruiken meerdere databases om de differentieel tot expressie gebrachte genen (DEG's) functioneel te annoteren en de overeenkomstige verrijkingsanalyse uit te voeren, waardoor inzicht wordt verkregen in de cellulaire en moleculaire processen die ten grondslag liggen aan de transcriptoomrespons.
●Ondersteuning na verkoop: Onze inzet gaat verder dan de voltooiing van het project met een after-sales serviceperiode van 3 maanden.Gedurende deze tijd bieden we projectopvolging, hulp bij het oplossen van problemen en vraag- en antwoordsessies om eventuele vragen met betrekking tot de resultaten te beantwoorden
Monstervereisten en levering
| Bibliotheek | Sequentiestrategie | Gegevens aanbevolen | Kwaliteitscontrole |
| rRNA uitgeput | Illumina PE150 | 16 GB | Q30≥85% |
| Maat geselecteerd | Illumina SE50 | 10-20 miljoen keer gelezen |
Voorbeeldvereisten:
Nucleotiden:
| Conc. (ng/μl) | Hoeveelheid (μg) | Puurheid | Integriteit |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Beperkte of geen eiwit- of DNA-verontreiniging weergegeven op de gel. | Planten: RIN≥6,5 Dier: RIN≥7,0 5,0≥28S/18S≥1,0; beperkte of geen basislijnhoogte |
Aanbevolen monsterlevering
Houder:
Centrifugeerbuisje van 2 ml (aluminiumfolie wordt niet aanbevolen)
Monsterlabeling: groeperen+repliceren, bijvoorbeeld A1, A2, A3;B1, B2, B3... ...
Verzending:
1. Droogijs: monsters moeten in zakken worden verpakt en in droogijs worden begraven.
2.RNAstable buizen: RNA-monsters kunnen worden gedroogd in een RNA-stabilisatiebuis (bijv. RNAstable®) en bij kamertemperatuur worden verzonden.
Servicewerkstroom

Experimentontwerp

Levering van monsters

RNA-extractie

Bouw van bibliotheek

Volgorde aanbrengen in

Gegevensanalyse

After-sales diensten
Bio-informatica
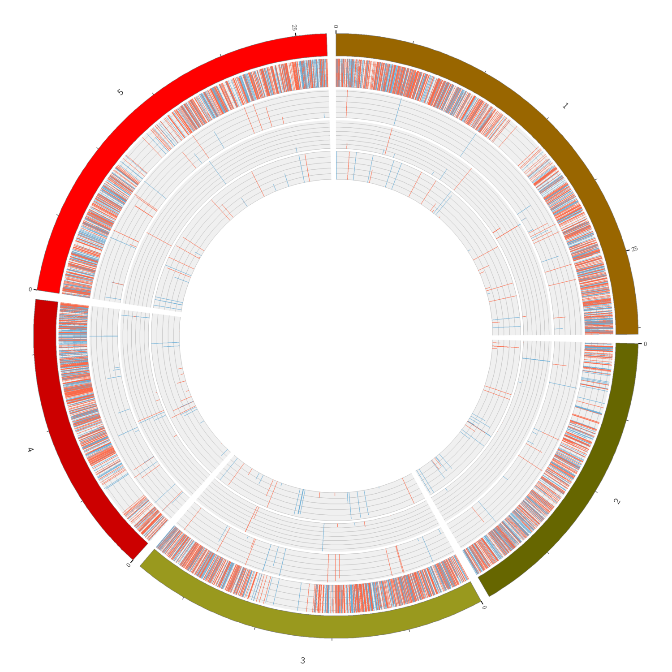
Overzicht van RNA-expressie
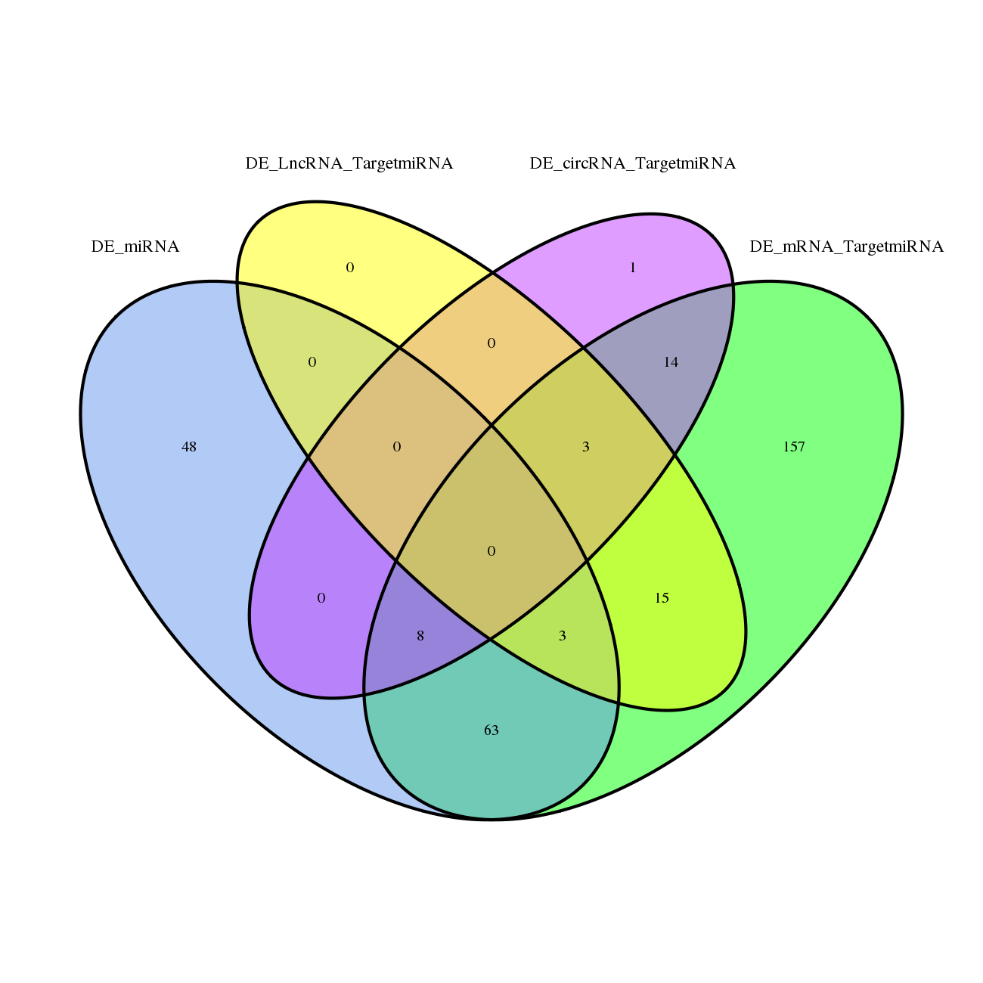
Differentieel tot expressie gebrachte genen
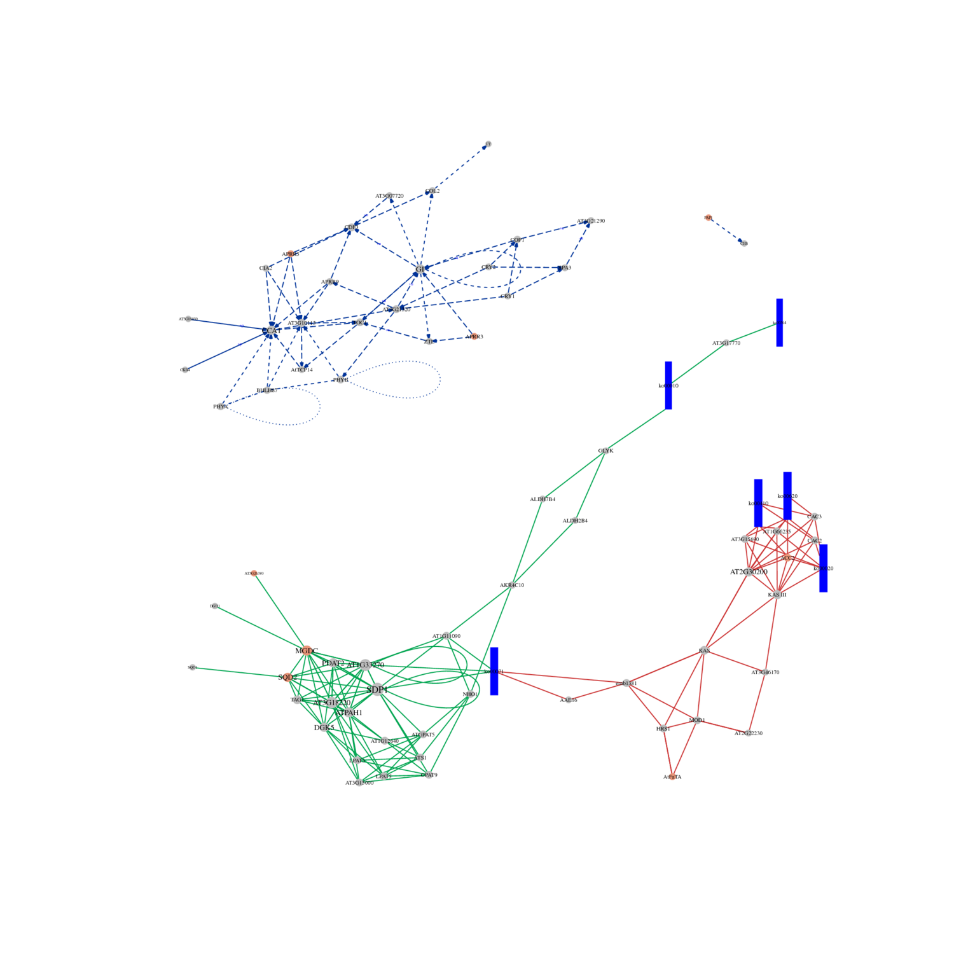
ceRNA-analyse
 Differentieel tot expressie gebrachte miRNA's en gerelateerde RNA's
Differentieel tot expressie gebrachte miRNA's en gerelateerde RNA's
Ontdek de onderzoeksvooruitgang die wordt mogelijk gemaakt door de volledige transcriptoomsequencingdiensten van BMKGene via een samengestelde verzameling publicaties.
Dai, Y. et al.(2022) 'Uitgebreide expressieprofielen van mRNA's, lncRNA's en miRNA's bij de ziekte van Kashin-Beck geïdentificeerd door RNA-sequencing', Molecular Omics, 18(2), pp. 154–166.doi: 10.1039/D1MO00370D.
Liu, N. nan et al.(2022) 'Volledige transcriptoomanalyse van de kouderesistentie van Apis cerana in de Changbai-berg tijdens de overwinteringsperiode.', Gene, 830, pp. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ et al.(2022) 'Multi-Omics op integratie gebaseerde prioritering van concurrerende endogene RNA-regulatienetwerken bij kleincellige longkanker: moleculaire kenmerken en kandidaat-geneesmiddelen', Frontiers in Oncology, 12, p.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. et al.(2022) 'Geïntegreerde analyse van de lncRNA/circRNA-miRNA-mRNA-expressieprofielen onthult nieuwe inzichten in potentiële mechanismen als reactie op wortelknobbelnematoden in pinda's', BMC Genomics, 23(1), pp. 1–12.doi: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. et al.(2022) 'Volledige transcriptoom-RNA-sequencing benadrukt de moleculaire mechanismen die geassocieerd zijn met het behoud van de kwaliteit na de oogst in broccoli door rode LED-bestraling', Postharvest Biology and Technology, 188, p.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.