TRANSCRIPTOMICA
natuur
COMMUNICATIE
Volledige transcriptiekarakterisering van SF3B1-mutatie bij chronische lymfatische leukemie onthult downregulatie van vastgehouden introns
Volledige transcripties|Nanoporiënsequencing|Alternatieve isovormanalyse
Achtergrond
SEr is algemeen gerapporteerd dat omatische mutaties in splicingfactor SF3B1 geassocieerd zijn met verschillende vormen van kanker, waaronder chronische lymfatische leukemie (CLL), oogmelanoom, borstkanker, enz. Bovendien hebben kort gelezen transcriptomische onderzoeken afwijkende splitsingspatronen aan het licht gebracht die worden geïnduceerd door SF3B1-mutaties.Onderzoek naar deze alternatieve splitsingspatronen is echter lange tijd beperkt gebleven tot het niveau van gebeurtenissen en het gebrek aan kennis op isovormniveau vanwege de beperking van kort gelezen samengestelde transcripties.Hier werd een nanopore-sequencingplatform geïntroduceerd om transcripties van volledige lengte te genereren, wat onderzoek naar AS-isovormen mogelijk maakte.
Experimenteel ontwerp
Experimenten
Groepering:1. CLL-SF3B1(WT) 2. CLL-SF3B1(K700E-mutatie);3. Normale B-cellen
Sequentiestrategie:MinION 2D-bibliotheeksequencing, PromethION 1D-bibliotheeksequencing;Short-read-gegevens van dezelfde monsters
Sequencing-platform:ONT MinION;ONT PromethION;
Bioinformatische analyse
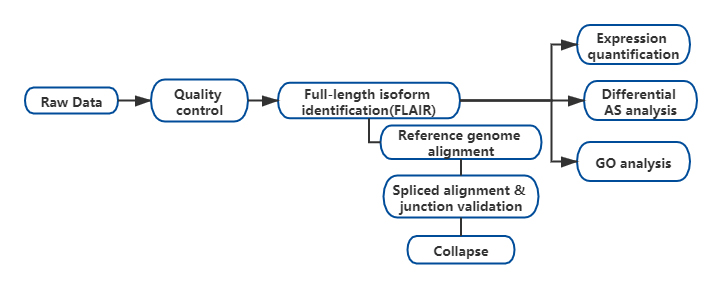
Resultaten
AEr werden in totaal 257 miljoen metingen gegenereerd uit 6 CLL-monsters en 3 B-cellen.Gemiddeld werd 30,5% van deze lezingen geïdentificeerd als volledige transcripties.
FEr werd een alternatieve isovormanalyse van RNA (FLAIR) met een volledige lengte ontwikkeld om een reeks betrouwbare isovormen te genereren.FLAIR kan worden samengevat als:
Nanopore leest uitlijning: identificeer de algemene transcriptstructuur op basis van referentiegenoom;
Scorrectie van plice-junctie: correcte sequentiefouten (rood) met splitsingsplaats van geannoteerde introns, introns van kort gelezen gegevens of beide;
Collapse: vat representatieve isovormen samen op basis van splitsingsketens (first-pass set).Selecteer isofrom met veel vertrouwen op basis van het aantal ondersteunende leesbewerkingen (drempel: 3).
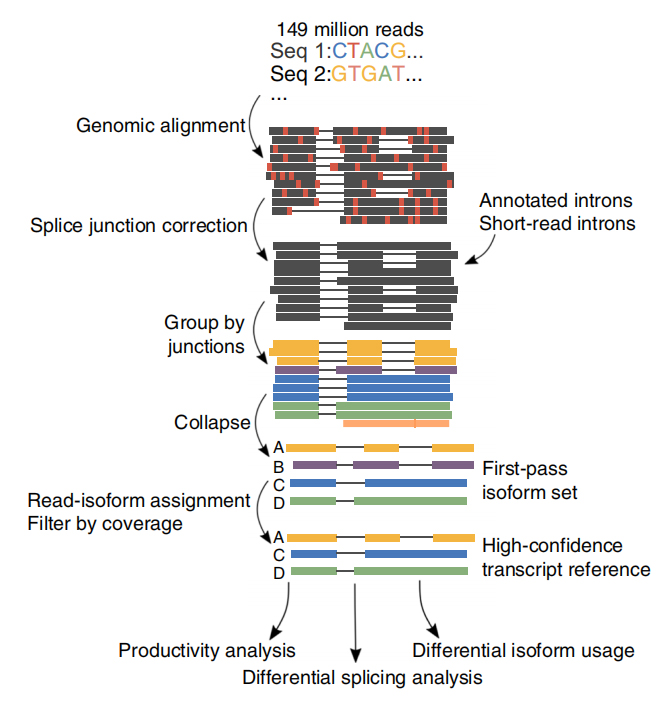
Figuur 1. FLAIR-analyse om isovormen met de volledige lengte te identificeren geassocieerd met SF3B1-mutatie in CLL
FLAIR heeft 326.699 gesplitste isovormen met hoge betrouwbaarheid geïdentificeerd, waarvan 90% nieuwe isovormen zijn.De meeste van deze niet-geannoteerde isovormen bleken nieuwe combinaties te zijn van bekende splitsingsverbindingen (142.971), terwijl de overige nieuwe isovormen ofwel behouden intron (21.700) of nieuw exon (3594) bevatten.
LOngelezen sequenties maken de identificatie mogelijk van mutante SF3B1-K700E-gewijzigde splitsingsplaatsen op isovormniveau.Er werd gevonden dat 35 alternatieve 3'SS's en 10 alternatieve 5'SS's significant verschillend gesplitst waren tussen SF3B1-K700E en SF3B1-WT.33 van de 35 wijzigingen werden nieuw ontdekt door lang gelezen sequenties.In Nanopore-gegevens is de verdeling van de afstand tussen SF3B1-K700E-gewijzigde 3'SS's en pieken van canonieke locaties ongeveer -20 bp, wat aanzienlijk verschilt van een controleverdeling, vergelijkbaar met wat is gerapporteerd in CLL-kortgelezen sequenties.Isovormen van het ERGIC3-gen werden geanalyseerd, waarbij een nieuwe isovorm die de proximale splitsingsplaats bevatte overvloediger werd aangetroffen in SF3B1-K700E.Zowel proximale als distale 3'SS waren geassocieerd met onderscheidende AS-patronen die meerdere isovormen genereerden.
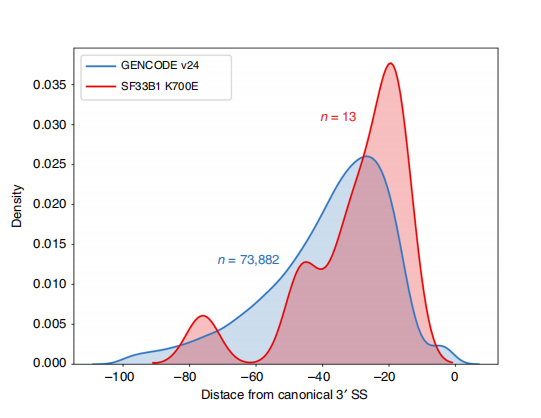
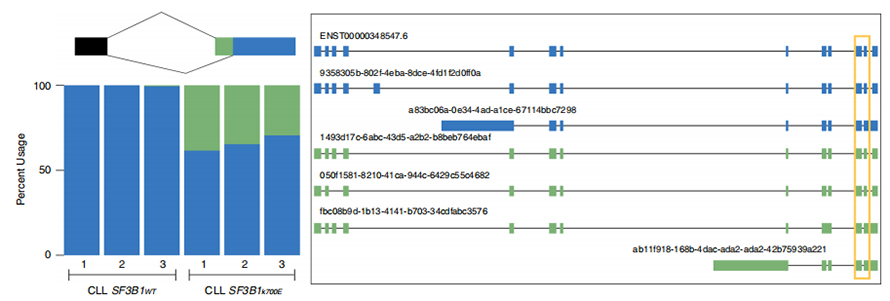
Figuur 2. Alternatieve 3'-splitsingspatronen geïdentificeerd met sequentiegegevens van nanoporiën
Analyse van het gebruik van IR-gebeurtenissen is lange tijd beperkt gebleven in op korte metingen gebaseerde analyses vanwege het vertrouwen in IR-identificatie en kwantificering.De expressie van IR-isovormen in SF3B1-K700E en SF3B1-WT werd gekwantificeerd op basis van nanoporiesequenties, wat een globale neerwaartse regulatie van IR-isovormen in SF3B1-K700E onthulde.
Figuur 4. Landbouwintensiteit en netwerkconnectiviteit tussen drie landbouwsystemen (A en B);Willekeurige bosanalyse (C) en relatie tussen landbouwintensiteit en AMF-kolonisatie (D)
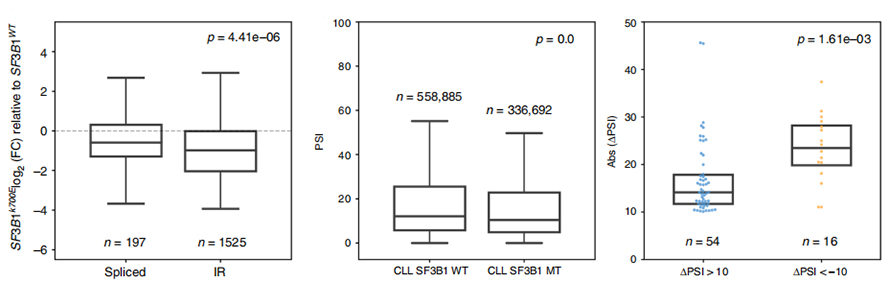
Figuur 3. Intron-retentiegebeurtenissen worden sterker gedownreguleerd in CLL SF3B1-K700E
Technologie
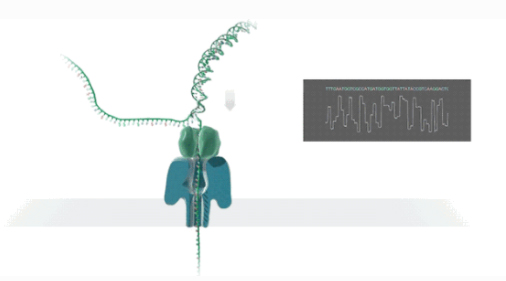
Nanopore Long-read-sequencing
NAnopore-sequencing is een real-time elektrische signaalsequencing-technologie met één molecuul.
Ddubbelstrengig DNA of RNA zal binden aan nanoporeus eiwit ingebed in de biofilm en zich afwikkelen onder leiding van motoreiwit.
DNA/RNA-strengen passeren met een bepaalde snelheid het nanoporiekanaaleiwit onder invloed van spanningsverschil.
Molecules genereren verschillende elektrische signalen volgens de chemische structuur.
Rrealtime detectie van sequenties wordt bereikt door basisoproepen.
Prestaties van transcriptoomsequencing over de volledige lengte
√ Gegevensverzadiging
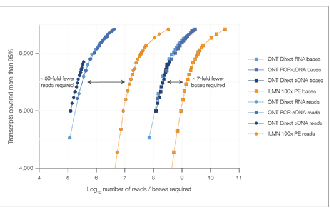
Er zijn zeven keer minder metingen nodig om een vergelijkbare gegevensverzadiging te bereiken.
√ Identificatie van transcriptstructuur
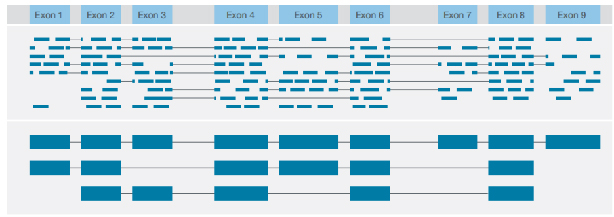
Identificatie van diverse structurele varianten met consensus-uitlezing van de volledige lengte van elk transcript
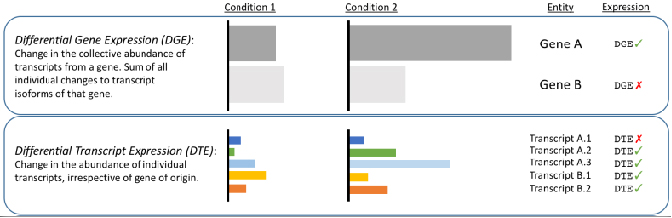
√ Differentiële analyse op transcriptieniveau - Onthul veranderingen die verborgen zijn door korte lezingen
Referentie
Tang AD, Soulette CM, Baren MJV, et al.Volledige transcriptkarakterisering van de SF3B1-mutatie bij chronische lymfatische leukemie onthult downregulatie van vastgehouden introns [J].Natuurcommunicatie.
Technologie en hoogtepunten heeft tot doel de meest recente succesvolle toepassing van verschillende high-throughput sequencing-technologieën in verschillende onderzoeksarena's te delen, evenals briljante ideeën op het gebied van experimenteel ontwerp en datamining.
Posttijd: 08-jan-2022

