
အပင်/တိရစ္ဆာန် De Novo ဂျီနိုမ် စီစစ်ခြင်း။
ဝန်ဆောင်မှု အားသာချက်များ

sequencing platforms နှင့် bioinformatics inဒီနိုဗိုဂျီနိုအာ စည်းဝေးပွဲ
(Amarasinghe SL et al.၊Genome ဇီဝဗေဒ2020)
● ဆန်းသစ်သော ဂျီနိုမ်များကို တည်ဆောက်ခြင်းနှင့် အကျိုးစီးပွားမျိုးစိတ်များအတွက် လက်ရှိရည်ညွှန်းထားသော ဂျီနိုမ်များကို ပိုမိုကောင်းမွန်အောင် ပြုလုပ်ခြင်း။
● တပ်ဆင်မှုတွင် ပိုမိုတိကျမှု၊ အဆက်ပြတ်မှုနှင့် ပြီးပြည့်စုံမှု
● မျိုးရိုးဗီဇ၊ QTLs၊ မျိုးရိုးဗီဇတည်းဖြတ်ခြင်း၊ မွေးမြူခြင်းစသည်ဖြင့် သုတေသနအတွက် အခြေခံအရင်းအမြစ်ကို တည်ဆောက်ခြင်း။
● တတိယမျိုးဆက် sequencing ပလပ်ဖောင်းများ၏ spectrum အပြည့်ဖြင့် တပ်ဆင်ထားသည်- one-stop genome assembly solution
● ကွဲပြားသောအင်္ဂါရပ်များဖြင့် ကွဲပြားသောမျိုးရိုးဗီဇများကို ဖြည့်ဆည်းပေးသည့် လိုက်လျောညီထွေရှိသော စီတန်းစီခြင်းနှင့် ပေါင်းစပ်နည်းဗျူဟာများ
● polyploids၊ ဧရာမဂျီနိုမ်များ စသည်တို့အပါအဝင် ရှုပ်ထွေးသော ဂျီနိုမ်စည်းဝေးပွဲများတွင် အတွေ့အကြုံကောင်းရှိသော ကျွမ်းကျင်သော ဇီဝနည်းပညာပညာရှင်အဖွဲ့။
● 900 ကျော် စုစည်းထုတ်ဝေထားသော သက်ရောက်မှုအချက်ပါရှိသော အောင်မြင်သော အမှုပေါင်း 100 ကျော်
● ခရိုမိုဆုန်းအဆင့် ဂျီနိုမ်စုစည်းမှုအတွက် 3 လအထိ အချိန်အလှည့်ကျ မြန်သည်။
● စမ်းသပ်ဘက်ခြမ်းနှင့် ဇီဝနည်းပညာနှစ်မျိုးစလုံးတွင် မူပိုင်ခွင့်များနှင့် ဆော့ဖ်ဝဲမူပိုင်ခွင့်များ ဆက်တိုက်ပါဝင်သည့် ခိုင်မာသောနည်းပညာပံ့ပိုးမှု။
ဝန်ဆောင်မှုသတ်မှတ်ချက်များ
|
အကြောင်းအရာ
|
ပလပ်ဖောင်း
|
အရှည်ဖတ်ပါ။
|
လွှမ်းခြုံ
|
| Genome စစ်တမ်း
| Illumina NovaSeq
| PE150
| ≥ 50X
|
| Genome Sequencing
| PacBio Revio
| 15 kb HiFi Reads
| ≥ 30X
|
| Hi-C
| Illumina NovaSeq
| PE150
| ≥100X
|
လုပ်ငန်းအသွားအလာ

နမူနာလိုအပ်ချက်များနှင့် ပေးပို့ခြင်း။
နမူနာလိုအပ်ချက်များ-
| မျိုးစိတ် | တစ်သျှူး | PacBio အတွက် | Nanopore အတွက် |
| တိရစ္ဆာန်များ | Visceral အင်္ဂါများ (အသည်း၊ သရက်ရွက် စသည်)၊ | ≥ 1.0 ဂရမ် | ≥ 3.5 ဂရမ် |
| ကြွက်သား | ≥ 1.5 ဂရမ် | ≥ 5.0 ဂရမ် | |
| နို့တိုက်သတ္တဝါများ၏သွေး | ≥ 1.5 mL | ≥ 5.0 mL | |
| ငါး သို့မဟုတ် ငှက်တို့၏သွေး | ≥ 0.2 mL | ≥ 0.5 mL | |
| အပင်များ | လတ်ဆတ်သောအရွက် | ≥ 1.5 ဂရမ် | ≥ 5.0 ဂရမ် |
| ပန်းပွင့် သို့မဟုတ် ပင်စည် | ≥ 3.5 ဂရမ် | ≥ 10.0 ဂရမ် | |
| အမြစ် သို့မဟုတ် အစေ့များ | ≥ 7.0 ဂရမ် | ≥ 20.0 ဂရမ် | |
| ဆဲလ်များ | ဆဲလ်ယဉ်ကျေးမှု | ≥ 3×107 | ≥ 1×108 |
နမူနာပေးပို့မှု အကြံပြုထားသည်။
ကွန်တိန်နာ- 2 ml centrifuge tube (Tin foil ကို မထောက်ခံပါ)
နမူနာအများစုအတွက်၊ အီသနောကို မထိန်းသိမ်းဖို့ အကြံပြုထားပါတယ်။
နမူနာတံဆိပ်ကပ်ခြင်း- နမူနာများကို တင်ပြသည့်နမူနာအချက်အလက်ဖောင်အတွက် ရှင်းရှင်းလင်းလင်း တံဆိပ်တပ်ထားရန် လိုအပ်ပါသည်။
ပို့ဆောင်မှု- ရေခဲခြောက်- နမူနာများကို အိတ်များတွင် ဦးစွာထုပ်ပိုးပြီး ရေခဲခြောက်၌ မြှုပ်နှံရန် လိုအပ်သည်။
Service Work Flow

စမ်းသပ်ထားတာ

နမူနာပေးပို့ခြင်း။

DNA ထုတ်ယူခြင်း။

စာကြည့်တိုက်တည်ဆောက်ရေး

Sequencing

ဒေတာခွဲခြမ်းစိတ်ဖြာ

ရောင်းချပြီးနောက်ဝန်ဆောင်မှုများ
*ဤတွင်ပြသထားသည့် သရုပ်ပြရလဒ်များသည် Biomarker Technologies ဖြင့်ထုတ်ဝေထားသော ဂျီနိုမ်များမှဖြစ်သည်။
1.Circos on chromosome-level genome assembly ofG. rotundifoliumNanopore sequencing platform မှ
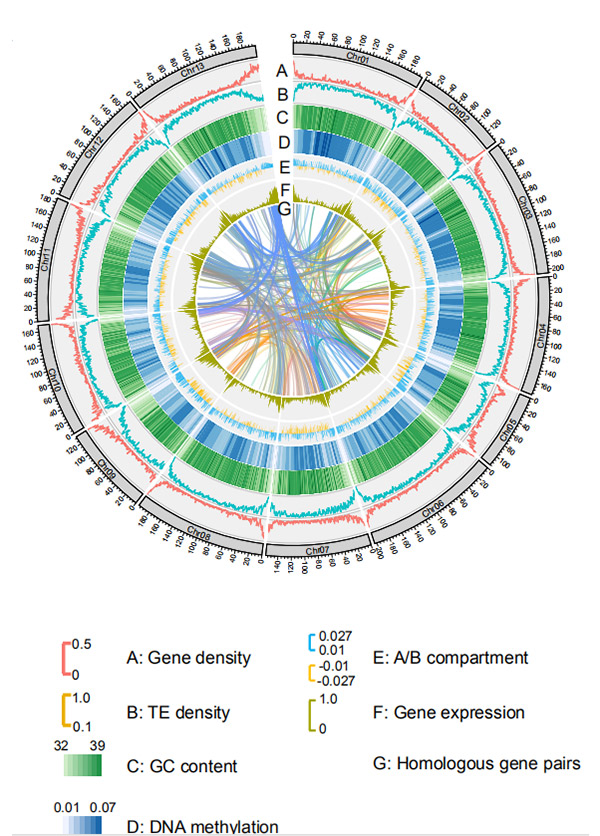
Wang M et al.၊မော်လီကျူး ဇီဝဗေဒနှင့် ဆင့်ကဲဖြစ်စဉ်၊ 2021
2.Statistics of Weining rye ဂျီနိုမ် စည်းဝေးပွဲနှင့် မှတ်ချက်
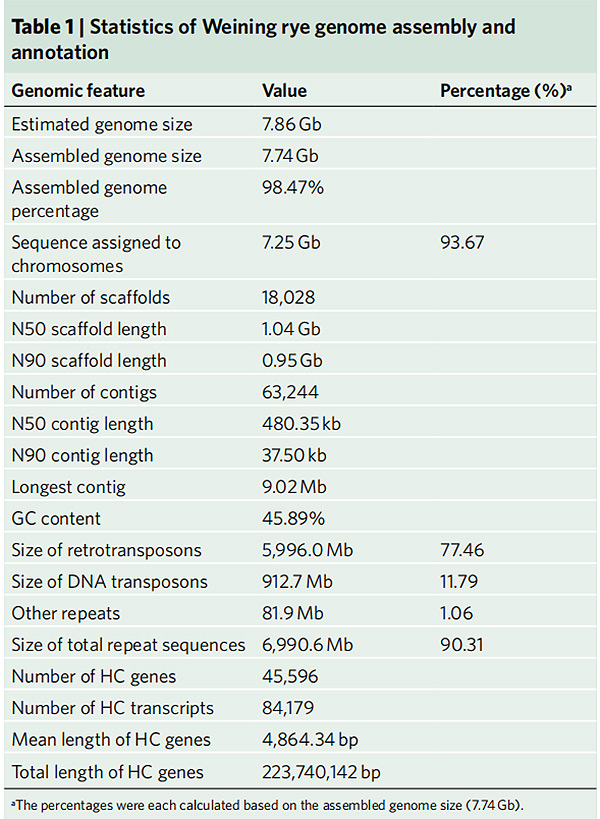
Li G et al.၊သဘာဝမျိုးရိုးဗီဇ၊ 2021
3.Gene ၏ခန့်မှန်းချက်Sechium eduleခန့်မှန်းနည်းသုံးမျိုးမှ ဆင်းသက်လာသော ဂျီနိုမီ၊ဒီနိုဗိုခန့်မှန်းချက်၊ Homology-based ဟောကိန်းနှင့် RNA-Seq ဒေတာအခြေခံ ခန့်မှန်းချက်
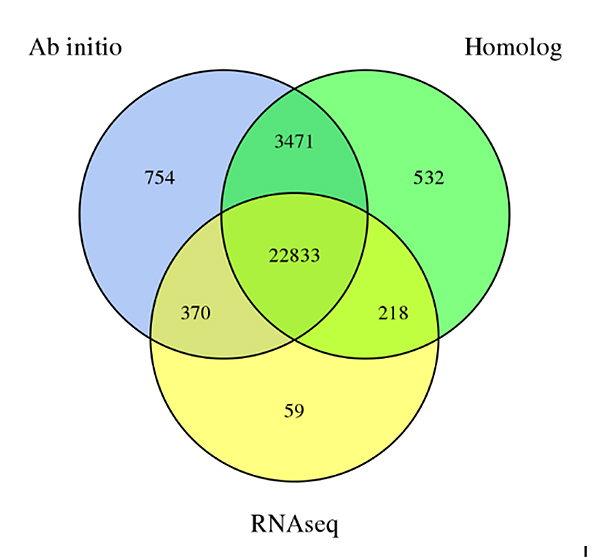
Fu A et al.၊ပန်းမာန်သုတေသန၊ 2021
4. ဂွမ်းဂျီနိုမ်သုံးမျိုးတွင် နဂိုအတိုင်း ရှည်လျားသော terminal ထပ်ခါတလဲလဲများကို ဖော်ထုတ်ခြင်း။
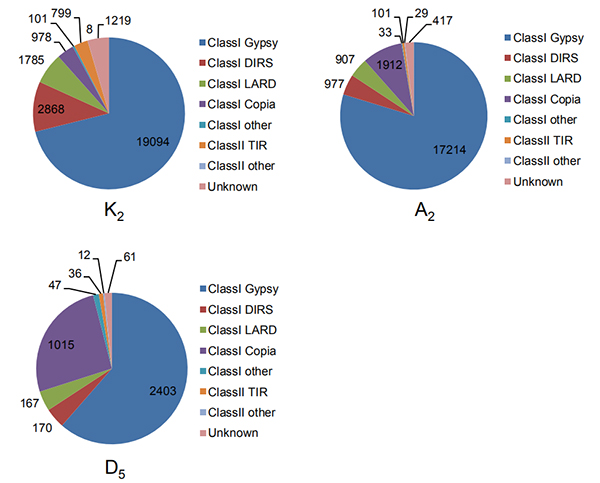
Wang M et al.၊မော်လီကျူး ဇီဝဗေဒနှင့် ဆင့်ကဲဖြစ်စဉ်၊ 2021
5.Hi-C အပူမြေပုံC. acuminataဂျီနိုမ်သည် ဂျီနိုမ်ကျယ်ပြန့်သော အလုံးစုံသော အပြန်အလှန်တုံ့ပြန်မှုများကို ပြသသည်။Hi-C အပြန်အလှန်တုံ့ပြန်မှုများ၏ပြင်းထန်မှုသည် contigs များကြားရှိ အကွာအဝေးနှင့် အချိုးကျပါသည်။ဤအပူမြေပုံပေါ်ရှိ သန့်ရှင်းသောမျဉ်းကြောင်းသည် ခရိုမိုဇုန်းများပေါ်ရှိ အဆက်အစပ်များကို အလွန်တိကျစွာ တွယ်ကပ်နေခြင်းကို ညွှန်ပြသည်။(Contig anchoring ratio: 96.03%)
kang M et al.၊သဘာဝဆက်သွယ်ရေး၊၂၀၂၁
BMK ဖြစ်ရပ်မှန်
အရည်အသွေးမြင့် ဂျီနိုမ်စည်းဝေးပွဲသည် ကောက်မျိုးရိုးဗီဇလက္ခဏာများနှင့် စိုက်ပျိုးရေးဆိုင်ရာ အရေးကြီးသော ဗီဇများကို မီးမောင်းထိုးပြသည်
ထုတ်ဝေသည်- သဘာဝမျိုးရိုးဗီဇ၊ 2021
ဆင့်ကဲနည်းဗျူဟာ-
Genome စည်းဝေးပွဲ- 20 kb စာကြည့်တိုက်ပါရှိသော PacBio CLR မုဒ် (497 Gb၊ ခန့်မှန်းခြေ 63×)
ဆက်တိုက်ပြင်ဆင်ခြင်း- Illumina ပလပ်ဖောင်းပေါ်တွင် 270 bp DNA စာကြည့်တိုက် (430 Gb၊ ခန့်မှန်းခြေ 54×) ပါရှိသော NGS
Contigs ကျောက်ချခြင်း- Illumina ပလပ်ဖောင်းပေါ်တွင် Hi-C စာကြည့်တိုက် (560 Gb၊ ခန့်မှန်းခြေ 71×)
အလင်းပြမြေပုံ- Bionano Irys ရှိ (779.55 Gb၊ ခန့်မှန်းခြေ 99×)
အဓိကရလဒ်များ
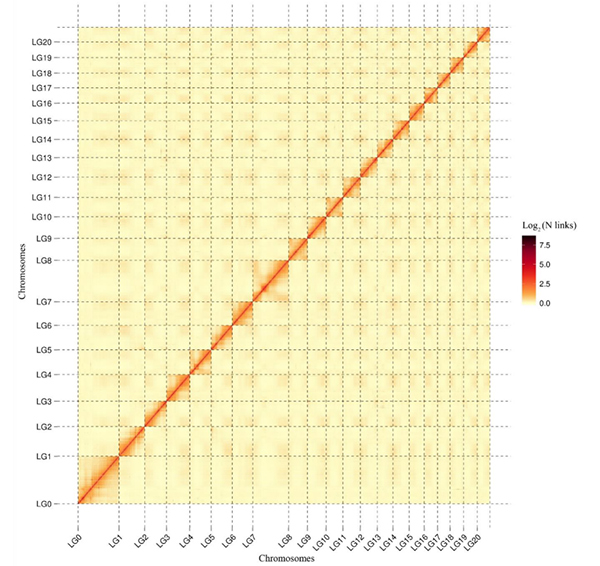
1. Weining rye ဂျီနိုမ်စည်းဝေးပွဲကို စုစုပေါင်း ဂျီနိုမ်အရွယ်အစား 7.74 Gb (စီးဆင်းမှု cytometry ဖြင့် ခန့်မှန်းထားသော ဂျီနိုမ်အရွယ်အစား၏ 98.74%) ဖြင့် ထုတ်ဝေခဲ့သည်။ဤတပ်ဆင်မှု၏ Scaffold N50 သည် 1.04 Gb ရရှိသည်။ပိုးမွှားများ၏ 93.67% ကို pseudo-chromosomes 7 ခုတွင် အောင်မြင်စွာ ကျောက်ချခဲ့သည်။ဤစုဝေးပွဲကို ချိတ်ဆက်မြေပုံ၊ LAI နှင့် BUSCO တို့က အကဲဖြတ်ခဲ့ပြီး အကဲဖြတ်မှုအားလုံးတွင် ရမှတ်များ မြင့်မားခဲ့သည်။
2. မျိုးရိုးဗီဇဆိုင်ရာ နှိုင်းယှဉ်လေ့လာမှုများ၊ မျိုးရိုးဗီဇချိတ်ဆက်မှုမြေပုံ၊ transcriptomics လေ့လာမှုများကို ဤဂျီနိုမ်အခြေခံပေါ်တွင် ပြုလုပ်ခဲ့သည်။ဂျီနိုမ်ကျယ်ပြန့်သော မျိုးရိုးဗီဇ ထပ်တူပွားမှုနှင့် ဓာတ်ဇီဝပေါင်းစပ်မှု ဗီဇအပေါ် သက်ရောက်မှုများ အပါအဝင် မျိုးဗီဇဆိုင်ရာ အင်္ဂါရပ်များနှင့် ဆက်စပ်သော စရိုက်လက္ခဏာများ စီးရီးများကို ဖော်ထုတ်ခဲ့သည်။ရှုပ်ထွေးသော prolamin loci ၏ ရုပ်ပိုင်းဆိုင်ရာအဖွဲ့အစည်း၊ မျိုးရိုးဗီဇဖော်ပြမှုတွင် အစောပိုင်းဦးခေါင်းစရိုက်လက္ခဏာများနှင့် putative domestication-sociated chromosomal regions နှင့် rye ရှိ loci တို့ပါဝင်သည်။
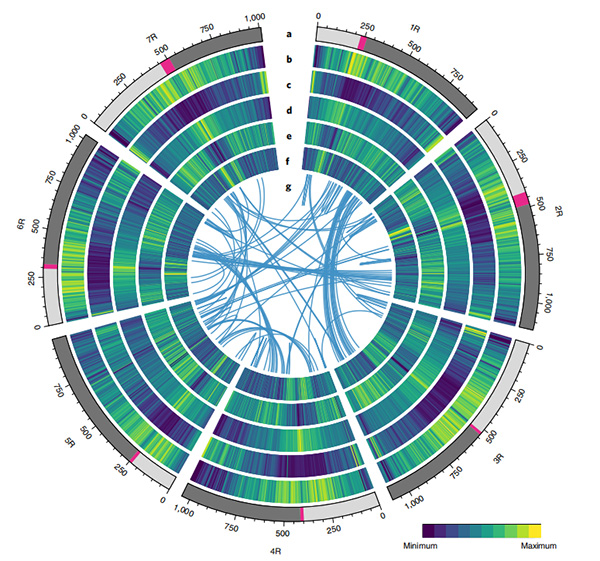 Weining rye genome ၏ မျိုးဗီဇဆိုင်ရာ အင်္ဂါရပ်များဆိုင်ရာ Circos ပုံကြမ်း | 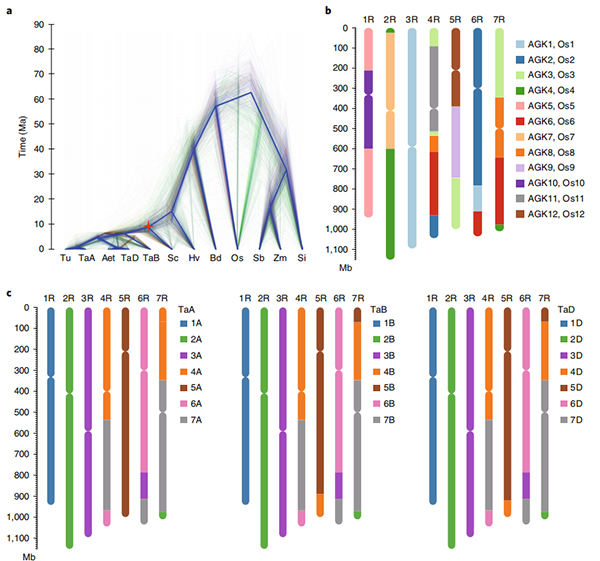 ကောက်ဂျီနိုမ်၏ ဆင့်ကဲဖြစ်စဉ်နှင့် ခရိုမိုဆုန်းပေါင်းစပ်ခွဲခြမ်းစိတ်ဖြာမှု |
Li, G., Wang, L., Yang, J.et al ။အရည်အသွေးမြင့် ဂျီနိုမ်စည်းဝေးပွဲသည် ကောက်မျိုးရိုးဗီဇလက္ခဏာများနှင့် စိုက်ပျိုးရေးဆိုင်ရာ အရေးကြီးသောမျိုးဗီဇများကို မီးမောင်းထိုးပြသည်။နတ် Genet ၅၃၊၅၇၄–၅၈၄ (၂၀၂၁)။
https://doi.org/10.1038/s41588-021-00808-z










