GENOMA EVOLŪCIJA

Salīdzinošās genoma analīzes izceļ transposonu izraisītu genoma paplašināšanos un 3D genoma locīšanas evolucionāro arhitektūru kokvilnā
Nanoporu sekvencēšana |Hi-C |PacBio sekvencēšana |Illumina |RNS sekvencēšana |3D genoma arhitektūra |Transposons |Salīdzinošā genomika
Šajā pētījumā Biomarker Technologies sniedza tehnisko atbalstu Nanopore sekvencēšanai, Hi-C un atbilstošai bioinformātikai.
Abstrakts
Transponējamo elementu (TE) amplifikācija ir atzīta par dzinējspēku, kas veicina genoma lieluma paplašināšanos un evolūciju, bet sekas 3D genoma arhitektūras veidošanā augos joprojām nav zināmas.Šeit mēs ziņojam par atsauces līmeņa genoma komplektiem trim kokvilnas sugām, kuru genoma izmērs ir trīskāršs, proti,Gossypium rotundifolium(K2),G. arboreum(A2) unG. raimondii(D5), izmantojot Oxford Nanopore Technologies.Salīdzinošās genoma analīzes dokumentē informāciju par līnijai raksturīgo TE amplifikāciju, kas veicina lielās genoma lieluma atšķirības (K2, 2, 44 Gb; A2, 1, 62 Gb; D5, 750, 19 Mb), un norāda uz salīdzinoši konservētu gēnu saturu un sintēzes attiecībām starp genomiem.Mēs noskaidrojām, ka aptuveni 17% sintētisko gēnu uzrāda hromatīna statusa izmaiņas starp aktīvajiem (“A”) un neaktīvajiem (“B”) nodalījumiem, un TE amplifikācija bija saistīta ar A nodalījuma īpatsvara palielināšanos gēnu reģionos (~ 7000 gēnu). ) K2 un A2 attiecībā pret D5.Trīs genomu vidū tika saglabāti tikai 42% no topoloģiski asociējošā domēna (TAD) robežām.Mūsu dati liecina par neseno TE pastiprināšanos pēc ciltsrakstam raksturīgu TAD robežu veidošanās.Šis pētījums atklāj transpozonu izraisītas genoma paplašināšanās lomu augstākas kārtas hromatīna struktūras attīstībā augos.
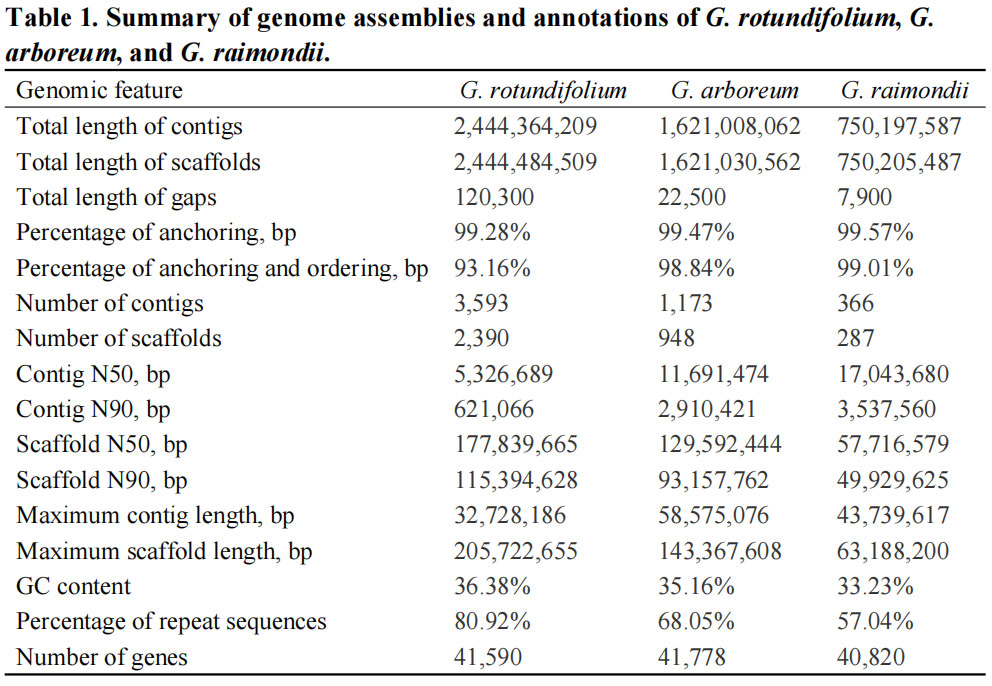
Galvenā genoma montāžas statistika
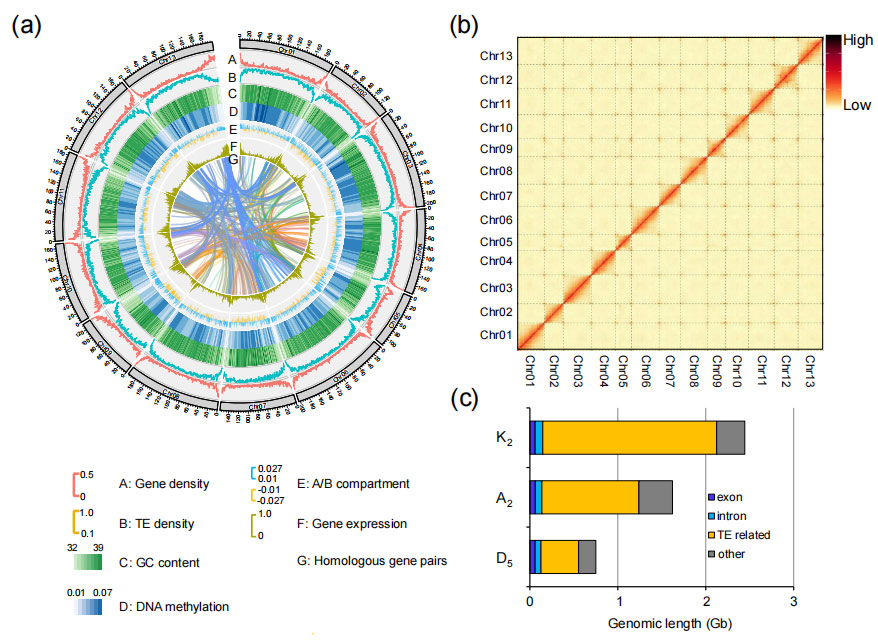
attēls.G. rotundifolium (K2) genoma komplektācija un pazīmju apraksts
Jaunumi un svarīgākie notikumi mērķis ir dalīties ar jaunākajiem veiksmīgajiem gadījumiem ar Biomarker Technologies, tverot jaunus zinātnes sasniegumus, kā arī ievērojamas metodes, kas izmantotas pētījuma laikā.
Izlikšanas laiks: Jan-05-2022

