Visa transkripto seka – Illumina
funkcijos
● Dvi biblioteka visam transkriptui sekti: rRNR išeikvojimas, po to PE150 bibliotekos paruošimas ir dydžio parinkimas, o po to SE50 bibliotekos paruošimas
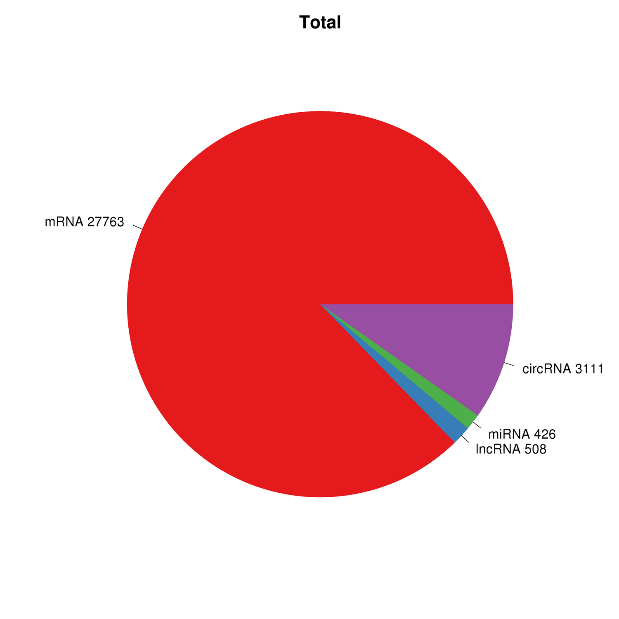
● Išsami mRNR, lncRNR, cirRNR ir miRNR bioinformatinė analizė atskirose bioinformatikos ataskaitose
● Bendra visos RNR ekspresijos analizė kombinuotoje ataskaitoje, įskaitant ceRNR tinklų analizę.
Paslaugos privalumai
●Nuodugni reguliavimo tinklų analizė: ceRNR tinklo analizę įgalina bendra mRNR, lncRNR, cirRNR ir miRNR seka ir išsami bioinformatinė darbo eiga.
●Išsami anotacija: naudojame kelias duomenų bazes, kad galėtume funkciškai komentuoti skirtingai išreikštus genus (DEG) ir atlikti atitinkamą sodrinimo analizę, suteikdami įžvalgų apie ląstelių ir molekulinius procesus, kuriais grindžiamas transkripto atsakas.
●Plati ekspertizė: sėkmingai užbaigusi daugiau nei 2000 transkripto projektų įvairiose tyrimų srityse, mūsų komanda kiekvienam projektui suteikia daug patirties.
●Griežta kokybės kontrolė: diegiame pagrindinius valdymo taškus visuose etapuose, nuo mėginių ir bibliotekos paruošimo iki sekos nustatymo ir bioinformatikos.Šis kruopštus stebėjimas užtikrina nuolatinių aukštos kokybės rezultatų teikimą.
● Išsami anotacija: naudojame kelias duomenų bazes, kad galėtume funkciškai komentuoti skirtingai išreikštus genus (DEG) ir atlikti atitinkamą sodrinimo analizę, suteikdami įžvalgų apie ląstelių ir molekulinius procesus, kuriais grindžiamas transkripto atsakas.
●Pagalba po pardavimo: Mūsų įsipareigojimas apima ne tik projekto užbaigimą, bet ir 3 mėnesių aptarnavimo po pardavimo laikotarpį.Per šį laiką siūlome tolesnius projekto veiksmus, trikčių šalinimo pagalbą ir klausimų ir atsakymų sesijas, kad galėtume atsakyti į bet kokias su rezultatais susijusias užklausas.
Pavyzdžių reikalavimai ir pristatymas
| biblioteka | Sekos nustatymo strategija | Rekomenduojami duomenys | Kokybės kontrolė |
| rRNR išeikvota | Illumina PE150 | 16 Gb | Q30≥85 % |
| Pasirinktas dydis | Illumina SE50 | 10-20 mln |
Pavyzdiniai reikalavimai:
Nukleotidai:
| Konc. (ng/μl) | Kiekis (μg) | Grynumas | Sąžiningumas |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Ant gelio rodomas ribotas baltymų ar DNR užterštumas arba jo nėra. | Augalai: RIN≥6,5 Gyvūnas: RIN≥7,0 5,0≥28S/18S≥1,0; ribotas arba jo nėra |
Rekomenduojamas pavyzdžių pristatymas
Sudėtis:
2 ml centrifugos mėgintuvėlis (Alavo folija nerekomenduojama)
Mėginio ženklinimas: Grupė+pakartojimas pvz. A1, A2, A3;B1, B2, B3.....
Siuntimas:
1. Sausas ledas: Mėginiai turi būti supakuoti į maišus ir palaidoti sausame lede.
2.RNR stabilūs mėgintuvėliai: RNR mėginius galima džiovinti RNR stabilizavimo mėgintuvėlyje (pvz., RNAstable®) ir gabenti kambario temperatūroje.
Aptarnavimo darbų eiga

Eksperimento dizainas

Mėginio pristatymas

RNR ekstrahavimas

Bibliotekos statyba

Sekos nustatymas

Duomenų analizė

Paslaugos po pardavimo
Bioinformatika
RNR ekspresijos apžvalga
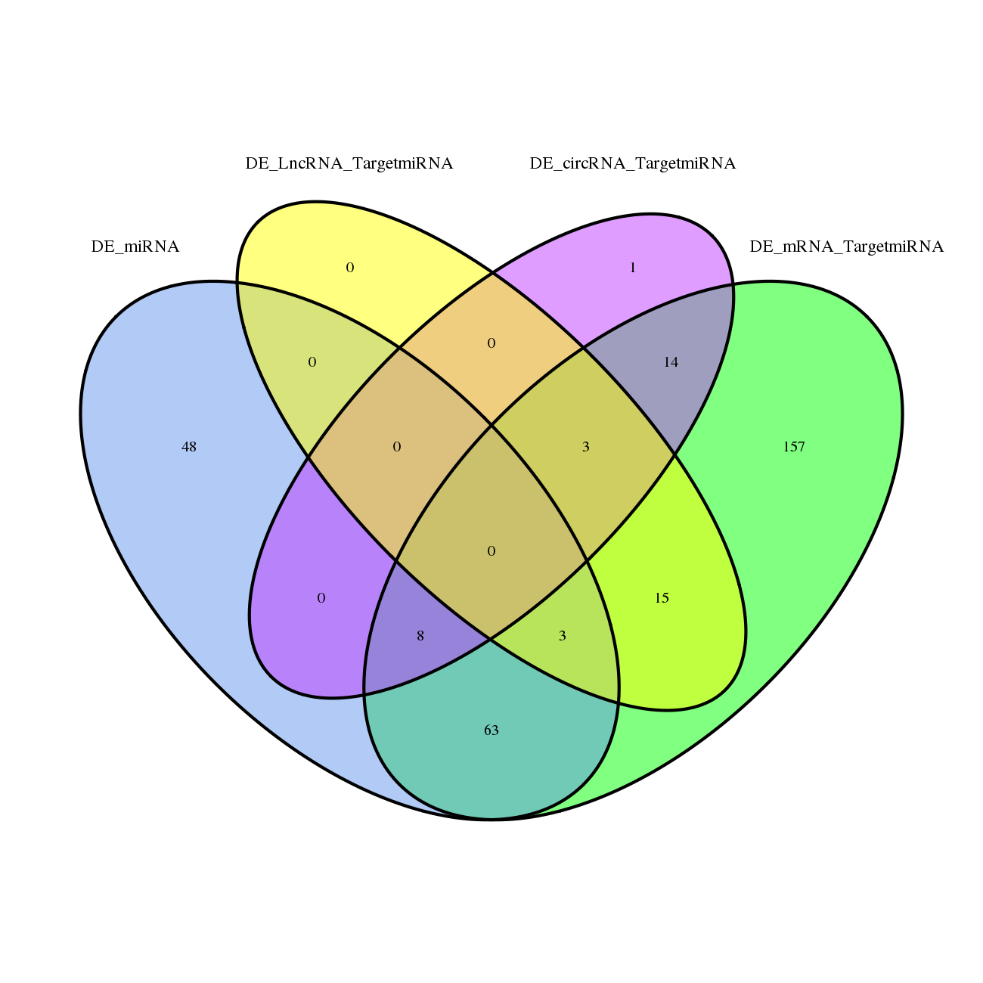
Skirtingai išreikšti genai
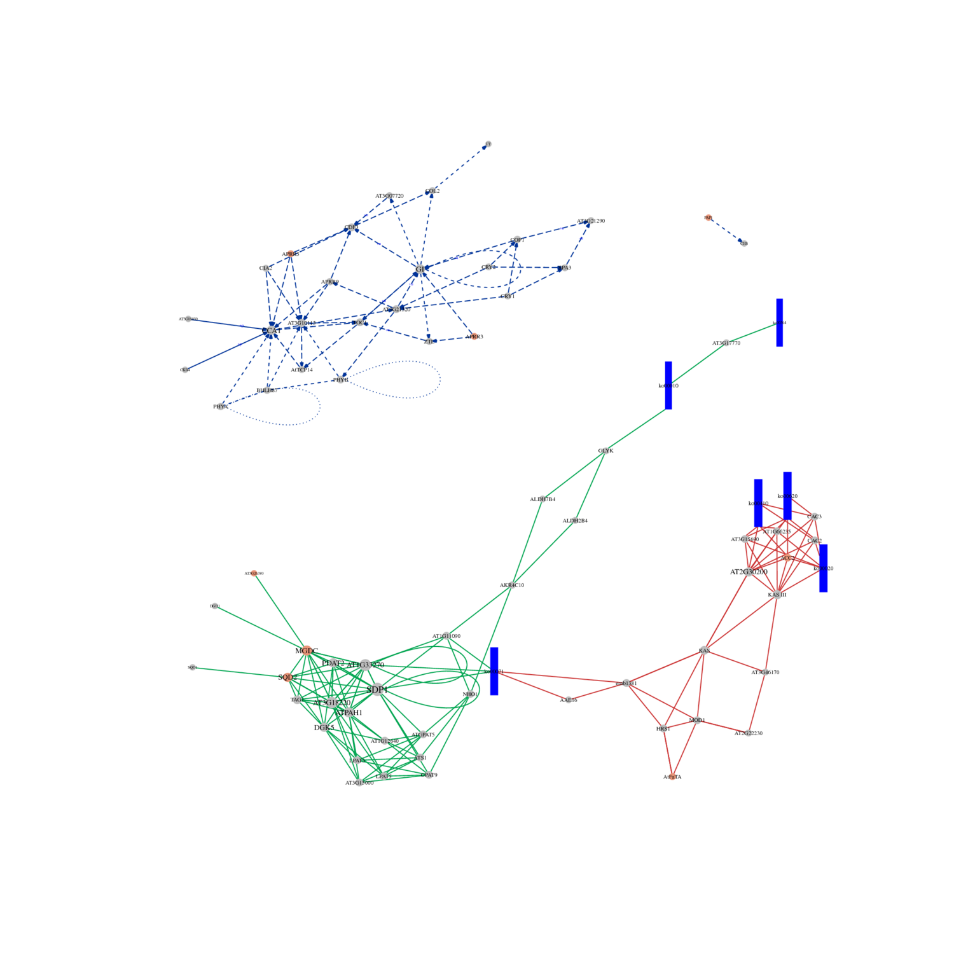
ceRNR analizė
Naršykite mokslinių tyrimų pažangą, kurią palengvino visos BMKGene transkriptų sekos nustatymo paslaugos, per kuruojamą publikacijų rinkinį.
Dai, Y. ir kt.(2022) „Išsamūs mRNR, lncRNR ir miRNR ekspresijos profiliai sergant Kashin-Beck liga, nustatyta RNR sekos nustatymu“, Molecular Omics, 18(2), p. 154–166.doi: 10.1039 / D1MO00370D.
Liu, N. nan ir kt.(2022) „Apis cerana atsparumo šalčiui viso ilgio transkriptų analizė Changbai kalne žiemojimo laikotarpiu.“, Gene, 830, p. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ ir kt.(2022) „Kelių omikų integracija pagrįstas konkuruojančių endogeninių RNR reguliavimo tinklų prioritetų nustatymas mažų ląstelių plaučių vėžyje: molekulinės charakteristikos ir vaistų kandidatai“, Onkologijos sienos, 12, p.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. ir kt.(2022) „Integruota lncRNR/cirRNR-miRNR-mRNR ekspresijos profilių analizė atskleidžia naujų įžvalgų apie galimus mechanizmus, reaguojant į žemės riešutų šaknų mazgų nematodus“, BMC Genomics, 23(1), p. 1–12.doi: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. ir kt.(2022) „Visos transkriptinės RNR sekos nustatymas išryškina molekulinius mechanizmus, susijusius su brokolių kokybės palaikymu po derliaus nuėmimo, naudojant raudoną šviesos diodą“, „Posharvest Biology and Technology“, 188, p.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.