
ການຈັດລໍາດັບການຖອດຂໍ້ຄວາມທັງໝົດ – Illumina
ຂໍ້ໄດ້ປຽບການບໍລິການ
● ການຄາດຄະເນກ່ຽວກັບ RNAs ທຸກປະເພດໃນແງ່ຂອງການນັບ, ການສະແດງອອກແລະການສະແດງອອກພີ່ນ້ອງທີ່ອີງໃສ່ chromosome
● ການກໍານົດ RNAs ທີ່ສະແດງອອກທີ່ແຕກຕ່າງກັນແລະການສະແດງອອກທີ່ສອດຄ້ອງກັນ
● ການວິເຄາະການສະແດງອອກຂອງເຊື້ອພັນທຸກໍາ
● ການວິເຄາະເຄືອຂ່າຍ ceRNA
● genes ທີ່ສໍາຄັນທີ່ກ່ຽວຂ້ອງກັບການວິເຄາະເສັ້ນທາງ
● ການຈັດສົ່ງຜົນໄດ້ຮັບໂດຍອີງໃສ່ BMKCloud: ການຂຸດຄົ້ນຂໍ້ມູນແບບກຳນົດເອງທີ່ມີຢູ່ໃນເວທີ
● ການບໍລິການຫຼັງການຂາຍມີເວລາ 3 ເດືອນຫຼັງຈາກໂຄງການສໍາເລັດ
ຄວາມຕ້ອງການຕົວຢ່າງແລະການຈັດສົ່ງ
| ຫໍສະໝຸດ | ເວທີ | ຂໍ້ມູນທີ່ແນະນໍາ | ຂໍ້ມູນ QC |
| RNA ທັງໝົດ | Illumina PE150&SE50 | Circ/lnc/mRNA: 16G;miRNA: 10M ອ່ານ | Q30≥85% |
ຄວາມຕ້ອງການຕົວຢ່າງ:
Nucleotides:
| ຄວາມບໍລິສຸດ | ຄວາມຊື່ສັດ | ການປົນເປື້ອນ | ຈໍານວນ |
| OD260/280≥1.7-2.5; OD260/230≥0.5-2.5; | ສໍາລັບພືດ: RIN≥6.5;ສໍາລັບສັດ: RIN≥7;28S/18S≥1.0;ຂອບເຂດຈໍາກັດ ຫຼືບໍ່ມີລະດັບຄວາມສູງ | ຈໍາກັດຫຼືບໍ່ມີການປົນເປື້ອນທາດໂປຼຕີນຫຼື DNA ທີ່ສະແດງຢູ່ໃນເຈນ. | Conc.≥100 ng/μl;ປະລິມານ ≥ 10 μl;ທັງໝົດ ≥ 2 μg |
ເນື້ອເຍື່ອ: ນ້ຳໜັກ (ແຫ້ງ): ≥1 g
* ສໍາລັບເນື້ອເຍື່ອຂະຫນາດນ້ອຍກວ່າ 5 ມລກ, ພວກເຮົາແນະນໍາໃຫ້ສົ່ງຕົວຢ່າງເນື້ອເຍື່ອ frozen (ໃນໄນໂຕຣເຈນຂອງແຫຼວ) flash.
ການລະງັບເຊວ: ຈຳນວນເຊວ = 3×107
*ພວກເຮົາແນະນໍາໃຫ້ສົ່ງ lysate ຫ້ອງ frozen.ໃນກໍລະນີທີ່ຕາລາງນັ້ນມີຂະໜາດນ້ອຍກວ່າ 5×105, flash frozen ໃນໄນໂຕຣເຈນຂອງແຫຼວແມ່ນແນະນໍາ.
ຕົວຢ່າງເລືອດ:
PA×geneBloodRNATube;
6 mLTRIzol ແລະ 2mL ເລືອດ (TRIzol: ເລືອດ = 3: 1)
ການຈັດສົ່ງຕົວຢ່າງທີ່ແນະນໍາ
ຕູ້ຄອນເທນເນີ:
ທໍ່ centrifuge 2 ມລ (ບໍ່ແນະນໍາໃຫ້ໃຊ້ຟອຍກົ່ວ)
ການຕິດສະຫຼາກຕົວຢ່າງ: Group+replicate ເຊັ່ນ: A1, A2, A3;B1, B2, B3......
ການຂົນສົ່ງ:
1.Dry-ice: ຕົວຢ່າງຕ້ອງຖືກບັນຈຸໃນຖົງແລະຝັງຢູ່ໃນແຫ້ງ-ice.
2.RNAstable tubes: ຕົວຢ່າງ RNA ສາມາດຕາກແຫ້ງໃນທໍ່ສະຖຽນລະພາບ RNA (ເຊັ່ນ: RNAstable®) ແລະສົ່ງໃນອຸນຫະພູມຫ້ອງ.
ກະແສວຽກບໍລິການ

ການອອກແບບທົດລອງ

ການຈັດສົ່ງຕົວຢ່າງ

ການສະກັດເອົາ RNA

ການກໍ່ສ້າງຫໍສະຫມຸດ

ການຈັດລໍາດັບ

ການວິເຄາະຂໍ້ມູນ

ບໍລິການຫຼັງການຂາຍ
ຊີວະວິທະຍາ
1.ການຄາດຄະເນກ່ຽວກັບທຸກປະເພດຂອງ RNAs ການສະແດງອອກພີ່ນ້ອງ
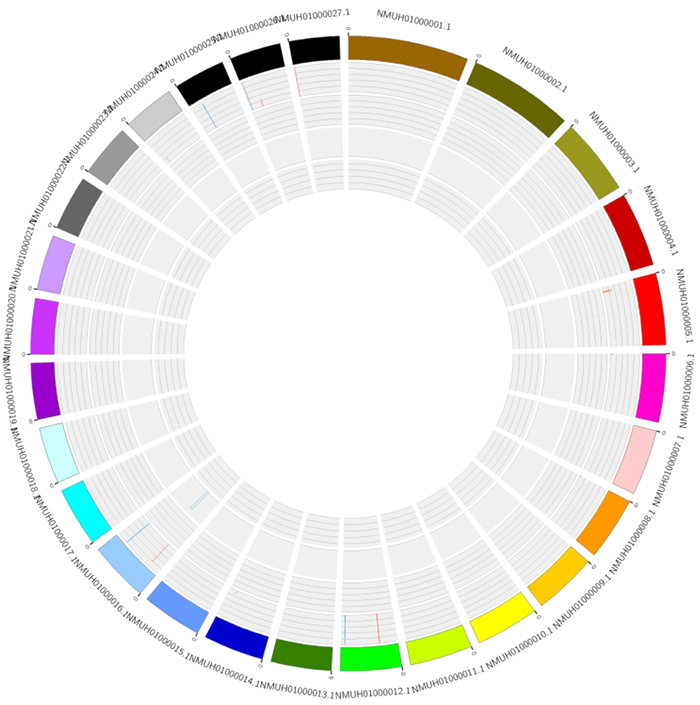
Circos ກ່ຽວກັບຄວາມສໍາຄັນຂອງຄວາມແຕກຕ່າງໃນການສະແດງອອກ RNA
2.Integrated ເຄືອຂ່າຍເສັ້ນທາງ KEGG
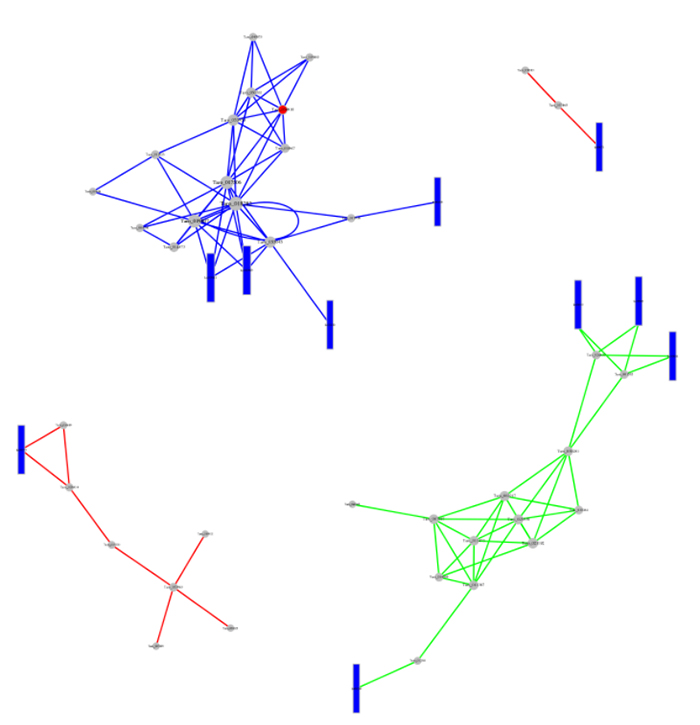
ເຄືອຂ່າຍເສັ້ນທາງ KEGG ປະສົມປະສານ
3.ceRNA ການວິເຄາະເຄືອຂ່າຍ
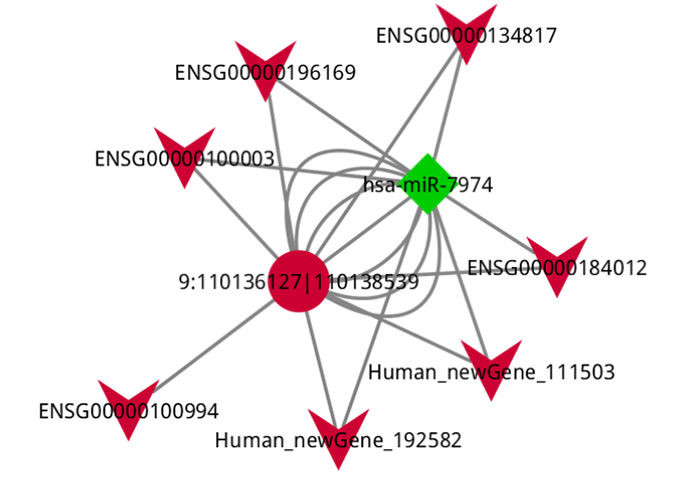
ceRNA ການໂຕ້ຕອບ DE-circRNA-miRNA-mRNA ທີ່ອີງໃສ່ເຄືອຂ່າຍ
ກໍລະນີ BMK
ຮູບແບບການສະແດງອອກ CircRNA ແລະເຄືອຂ່າຍ ceRNA ແລະ miRNA-mRNA ມີສ່ວນຮ່ວມໃນການພັດທະນາ Anther ໃນສາຍ CMS ຂອງ campestris Brassica
ຈັດພີມມາ:ວາລະສານສາກົນຂອງວິທະຍາສາດໂມເລກຸນ,2019
ຈຸລັງ NONMMUT015812-knockdown KP (shRNA-2) ແລະຈຸລັງຄວບຄຸມທາງລົບ (sh-Scr) ໄດ້ຮັບໃນວັນທີ 6 ຂອງການຕິດເຊື້ອໄວຣັດສະເພາະ.
ຜົນໄດ້ຮັບທີ່ສໍາຄັນ
ການສຶກສານີ້ໄດ້ສ້າງຕັ້ງສາຍພັນ Polima cytoplasm male sterility (CMS) “Bcpol97-05A”, ແລະສາຍທີ່ອຸດົມສົມບູນ, “Bcajh97-01B”, ໃນ Brassica campestris L. ssp.chinensis Makino, syn.B. rapa ssp.chinensis, ແລະປະຕິບັດການປຽບທຽບການສະແດງອອກຂອງ RNA profile ລະຫວ່າງຕາດອກຂອງສາຍເປັນຫມັນແລະສາຍອຸດົມສົມບູນໂດຍການລໍາດັບການຖອດຂໍ້ຄວາມທັງຫມົດ.
1.ຈຳນວນທັງໝົດ 31 ອັນທີ່ສະແດງອອກດ້ວຍຄວາມແຕກຕ່າງ (DE) circRNAs, 47 DE miRNAs, ແລະ 4779 DE mRNAs ຖືກລະບຸ.
2.ການວິເຄາະ gene ontology (GO) ສະແດງໃຫ້ເຫັນວ່າ genes DE ສ່ວນໃຫຍ່ມີສ່ວນຮ່ວມໃນການພັດທະນາກໍາແພງ pollen.
3. ການວິເຄາະການເສີມສ້າງທາງ KEGG ຂອງ DE mRNAs (ລວມທັງ mRNAs ທີ່ມີການຄວບຄຸມຂຶ້ນ ແລະລົງ-ຄວບຄຸມຢູ່ໃນເສັ້ນທີ່ເປັນໝັນເມື່ອທຽບກັບສາຍທີ່ອຸດົມສົມບູນ) ສະແດງໃຫ້ເຫັນວ່າ "ທາດແປ້ງ ແລະ sucrosemetabolism", "phenylpropanoid biosynthesis", ແລະ "pentose ແລະ glucuronate interconversions. ” ແມ່ນເສັ້ນທາງການເຜົາຜະຫລານອາຫານທີ່ອຸດົມສົມບູນທີ່ສຸດ.
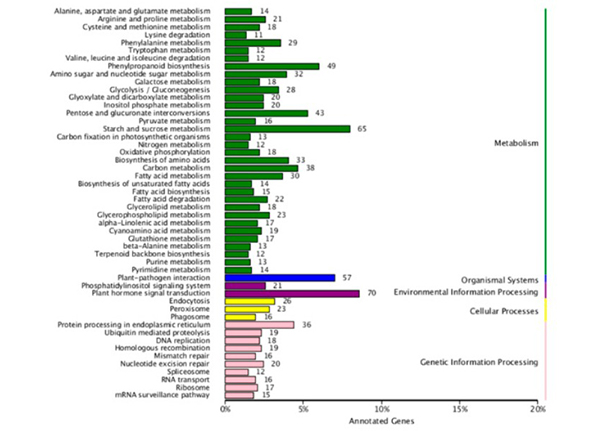 ການວິເຄາະການຂະຫຍາຍເສັ້ນທາງ KEGG ຂອງ mRNAs ທີ່ສະແດງອອກແຕກຕ່າງກັນ | 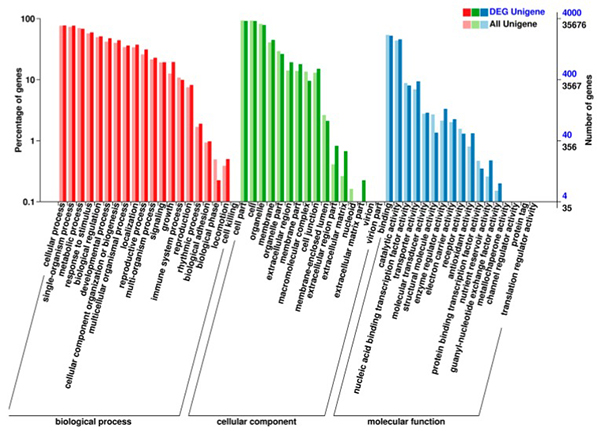 ການຈັດປະເພດພັນທຸກໍາ ontology (GO) ຂອງ mRNAs ທີ່ສະແດງອອກແຕກຕ່າງກັນ | 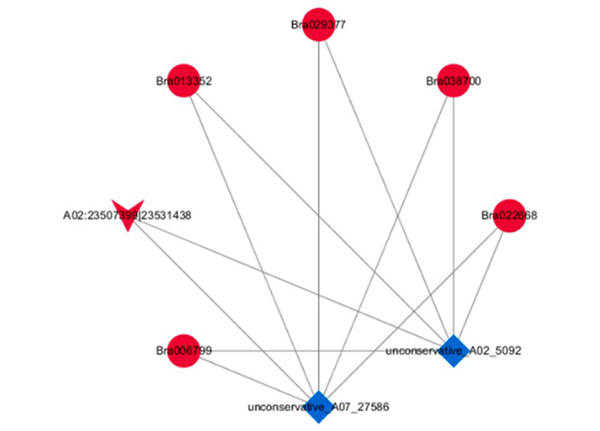 ມຸມມອງຂອງເຄືອຂ່າຍສາມເທົ່າ DEcircRNA–DEmiRNA–DEmRNA |
ອ້າງອິງ
Liang Y, Zhang Y, Xu L, et al.ຮູບແບບການສະແດງອອກ CircRNA ແລະເຄືອຂ່າຍ ceRNA ແລະ miRNA–mRNA ມີສ່ວນຮ່ວມໃນການພັດທະນາ Anther ໃນສາຍ CMS ຂອງ campestris Brassica[J].International Journal of Molecular Sciences, 2019, 20(19):4808-.DoI: 10.3390/ijms20194808














