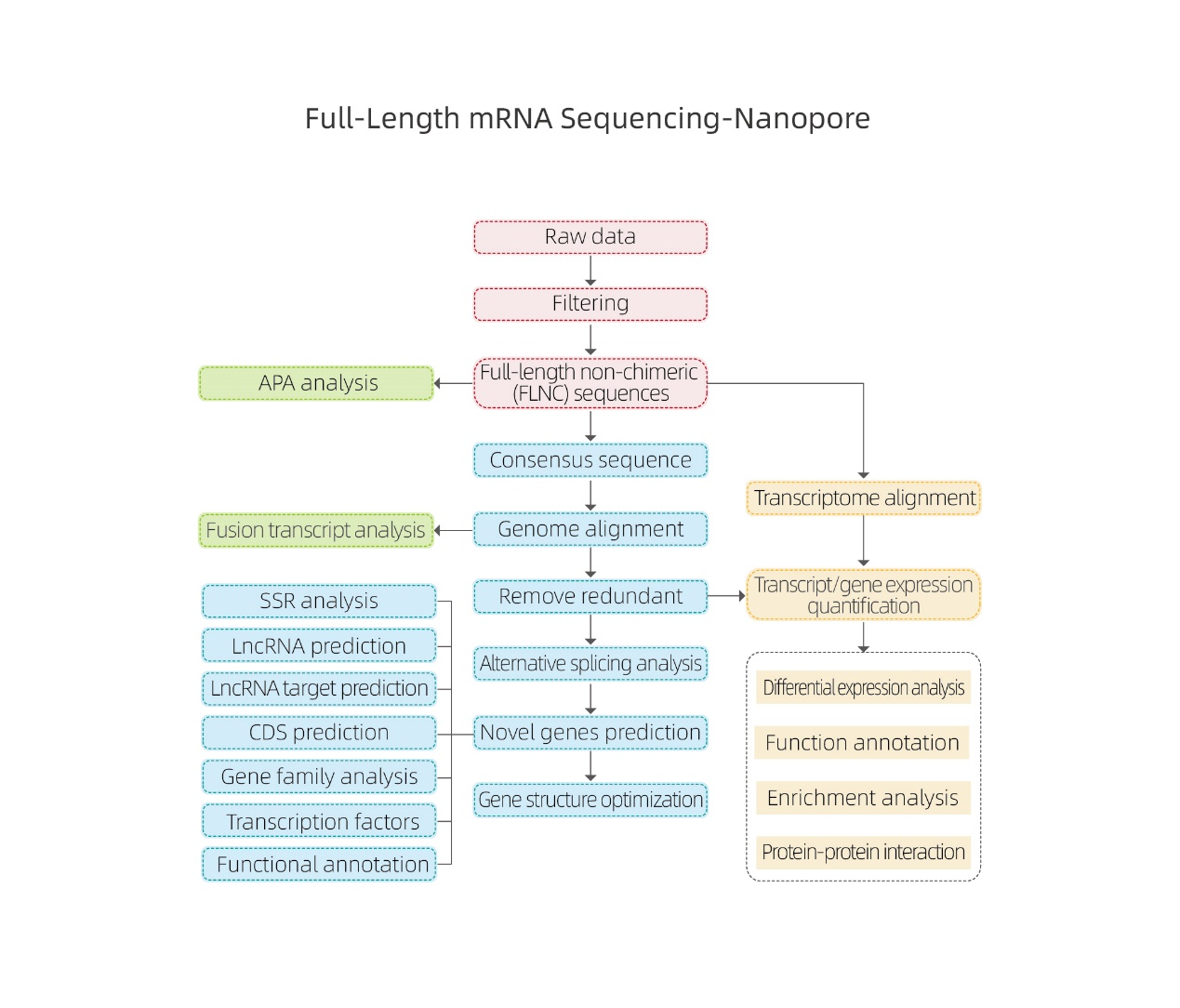
ຄວາມຍາວເຕັມ mRNA Sequencing-Nanopore
ຂໍ້ໄດ້ປຽບການບໍລິການ
● ຄວາມລຳອຽງຂອງລຳດັບຕໍ່າ
● ເປີດເຜີຍໂມເລກຸນ cDNA ທີ່ມີຄວາມຍາວເຕັມ
● ຕ້ອງການຂໍ້ມູນໜ້ອຍລົງເພື່ອໃຫ້ກວມເອົາຈໍານວນການຖອດຂໍ້ຄວາມດຽວກັນ
● ການກໍານົດຫຼາຍ isoforms ຕໍ່ gene
● ປະລິມານການສະແດງອອກໃນລະດັບ isoform
ຂໍ້ມູນຈໍາເພາະການບໍລິການ
| ຫໍສະໝຸດ | ເວທີ | ຂໍ້ມູນທີ່ແນະນຳ (Gb) | ການຄວບຄຸມຄຸນນະພາບ |
| cDNA-PCR(Poly-A ເສີມສ້າງ) | Nanopore PromethION P48 | 6 Gb/ຕົວຢ່າງ (ຂຶ້ນກັບຊະນິດ) | ອັດຕາສ່ວນຄວາມຍາວເຕັມ 70% ຄະແນນຄຸນນະພາບສະເລ່ຍ: Q10
|
ການວິເຄາະ bioinformatics
●ການປະມວນຜົນຂໍ້ມູນດິບ
● ການລະບຸການຖອດຂໍ້ຄວາມ
● ການເຊື່ອມຕົວສຳຮອງ
● ປະລິມານການສະແດງອອກໃນລະດັບ gene ແລະລະດັບ isoform
● ການວິເຄາະການສະແດງອອກທີ່ແຕກຕ່າງ
● ການອະທິບາຍຟັງຊັນ ແລະການປັບປຸງ (DEGs ແລະ DETs)
ຄວາມຕ້ອງການຕົວຢ່າງແລະການຈັດສົ່ງ
ຄວາມຕ້ອງການຕົວຢ່າງ:
Nucleotides:
| Conc.(ng/μl) | ປະລິມານ (μg) | ຄວາມບໍລິສຸດ | ຄວາມຊື່ສັດ |
| ≥ 100 | ≥ 0.6 | OD260/280=1.7-2.5 OD260/230=0.5-2.5 ຈໍາກັດຫຼືບໍ່ມີການປົນເປື້ອນທາດໂປຼຕີນຫຼື DNA ທີ່ສະແດງຢູ່ໃນເຈນ. | ສໍາລັບພືດ: RIN≥7.0; ສໍາລັບສັດ: RIN≥7.5; 5.0≥28S/18S≥1.0; ຂອບເຂດຈໍາກັດ ຫຼືບໍ່ມີລະດັບຄວາມສູງ |
ເນື້ອເຍື່ອ: ນ້ຳໜັກ (ແຫ້ງ): ≥1 g
* ສໍາລັບເນື້ອເຍື່ອຂະຫນາດນ້ອຍກວ່າ 5 ມລກ, ພວກເຮົາແນະນໍາໃຫ້ສົ່ງຕົວຢ່າງເນື້ອເຍື່ອ frozen (ໃນໄນໂຕຣເຈນຂອງແຫຼວ) flash.
ການລະງັບເຊວ: ຈຳນວນເຊວ = 3×106- 1×107
*ພວກເຮົາແນະນໍາໃຫ້ສົ່ງ lysate ຫ້ອງ frozen.ໃນກໍລະນີທີ່ຕາລາງນັ້ນນັບໜ້ອຍກວ່າ 5×105, ກະພິບ frozen ໃນໄນໂຕຣເຈນຂອງແຫຼວແມ່ນແນະນໍາໃຫ້, ເຊິ່ງດີກວ່າສໍາລັບການສະກັດຈຸນລະພາກ.
ຕົວຢ່າງເລືອດ: ປະລິມານ≥1 ml
ການຈັດສົ່ງຕົວຢ່າງທີ່ແນະນໍາ
ບັນຈຸ: ທໍ່ centrifuge 2 ມລ (ບໍ່ແນະນໍາໃຫ້ໃຊ້ຟອຍກົ່ວ)
ການຕິດສະຫຼາກຕົວຢ່າງ: Group+replicate ເຊັ່ນ: A1, A2, A3;B1, B2, B3......
ການຂົນສົ່ງ: 2, ກ້ອນແຫ້ງ: ຕົວຢ່າງຈໍາເປັນຕ້ອງໄດ້ບັນຈຸຢູ່ໃນຖົງແລະຝັງຢູ່ໃນກ້ອນແຫ້ງ.
- ທໍ່ RNAstable: ຕົວຢ່າງ RNA ສາມາດຕາກແຫ້ງໃນທໍ່ສະຖຽນລະພາບ RNA (ເຊັ່ນ: RNAstable®) ແລະສົ່ງໃນອຸນຫະພູມຫ້ອງ.
ກະແສວຽກບໍລິການ
Nucleotides:

ການຈັດສົ່ງຕົວຢ່າງ

ການກໍ່ສ້າງຫໍສະຫມຸດ

ການຈັດລໍາດັບ

ການວິເຄາະຂໍ້ມູນ

ບໍລິການຫຼັງການຂາຍ
ກະແສວຽກບໍລິການ
ເນື້ອເຍື່ອ:

ການອອກແບບທົດລອງ

ການຈັດສົ່ງຕົວຢ່າງ

ການສະກັດເອົາ RNA

ການກໍ່ສ້າງຫໍສະຫມຸດ

ການຈັດລໍາດັບ

ການວິເຄາະຂໍ້ມູນ

ບໍລິການຫຼັງການຂາຍ
1.ການວິເຄາະການສະແດງອອກທີ່ແຕກຕ່າງກັນ - ແຜນການຂອງພູເຂົາໄຟ
ການວິເຄາະການສະແດງອອກທີ່ແຕກຕ່າງກັນສາມາດຖືກປຸງແຕ່ງໃນລະດັບ gene ເພື່ອກໍານົດ genes ທີ່ສະແດງອອກແຕກຕ່າງກັນ (DEGs) ແລະໃນລະດັບ isoform ເພື່ອກໍານົດຄວາມແຕກຕ່າງ.
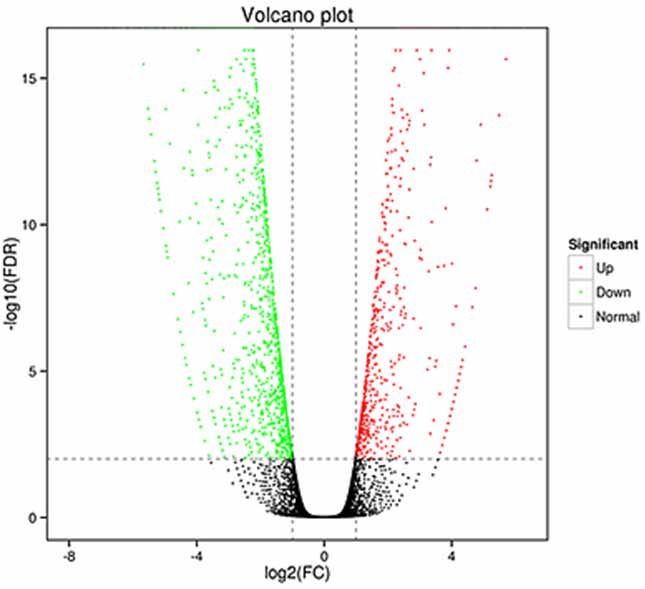
ການຖອດຂໍ້ຄວາມທີ່ສະແດງອອກ (DETs)
2.ແຜນຜັງຄວາມຮ້ອນຂອງກຸ່ມຕາມລຳດັບ
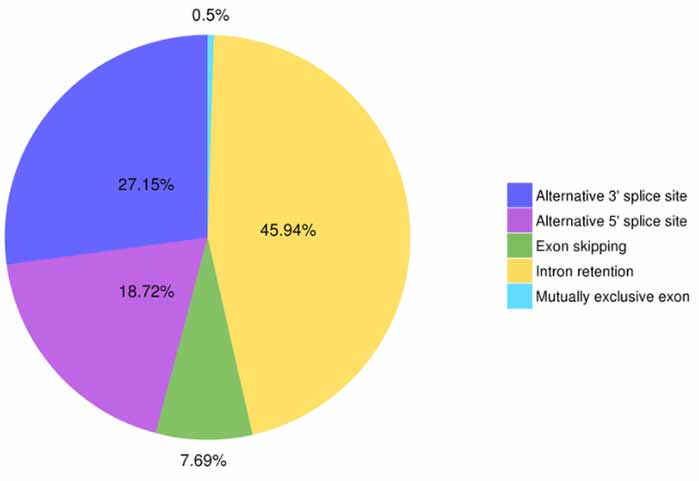
3.ການກໍານົດແລະການຈັດປະເພດ splicing ທາງເລືອກ
ຫ້າປະເພດຂອງເຫດການ splicing ທາງເລືອກສາມາດໄດ້ຮັບການຄາດຄະເນໂດຍ Astalavista.
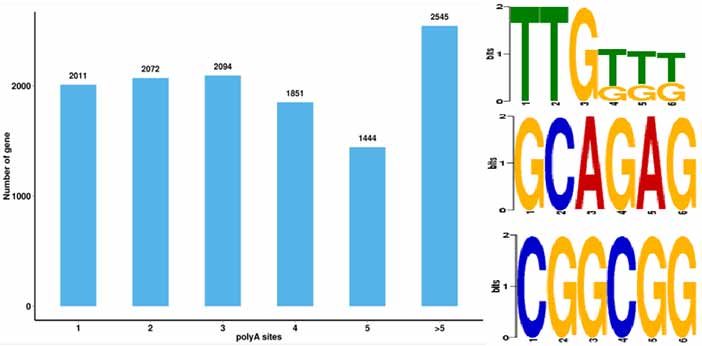
4.ການກໍານົດເຫດການ poly-adenylation ທາງເລືອກ (APA) ແລະ Motif ຢູ່ 50 bp ຂ້າງເທິງຂອງ poly-A
ກໍລະນີ BMK
ການກໍານົດຕົວແຍກທາງເລືອກແລະການວັດແທກລະດັບ isoform ໂດຍການລໍາດັບການຖອດຂໍ້ຄວາມແບບເຕັມຄວາມຍາວຂອງ nanopore
ຈັດພີມມາ:ການສື່ສານທໍາມະຊາດ, 2020
ຍຸດທະສາດການຈັດລໍາດັບ:
ການຈັດກຸ່ມ: 1. CLL-SF3B1(WT);2. ການກາຍພັນ CLL-SF3B1(K700E);3. ຈຸລັງ B ປົກກະຕິ
ຍຸດທະສາດການຈັດລໍາດັບ: ການຈັດລໍາດັບຫ້ອງສະຫມຸດ MinION 2D, ການຈັດລໍາດັບຫ້ອງສະຫມຸດ PromethION 1D;ຂໍ້ມູນທີ່ອ່ານສັ້ນຈາກຕົວຢ່າງດຽວກັນ
ເວທີການຈັດລໍາດັບ: Nanopore MinION;Nanopore PromethION;
ຜົນໄດ້ຮັບທີ່ສໍາຄັນ
1.Isoform-level Alternative Splicing Identification
ລໍາດັບທີ່ອ່ານດົນນານເຮັດໃຫ້ການກໍານົດຕົວຂອງ SF3B1 mutantK700E- ປ່ຽນແປງສະຖານທີ່ splice ໃນລະດັບ isoform.35 ທາງເລືອກ 3′SSs ແລະ 10 ທາງເລືອກ 5′SSs ຖືກພົບເຫັນວ່າມີຄວາມແຕກຕ່າງຢ່າງຫຼວງຫຼາຍລະຫວ່າງ SF3B1.K700Eແລະ SF3B1WT.33 ໃນ 35 ການປ່ຽນແປງໄດ້ຖືກຄົ້ນພົບໃຫມ່ໂດຍລໍາດັບທີ່ອ່ານຍາວ.
2.Isoform-level Alternative Splicing quantification
ການສະແດງອອກຂອງ isoforms ການເກັບຮັກສາ intron (IR) ໃນ SF3B1K700Eແລະ SF3B1WTໄດ້ຮັບການຄິດໄລ່ໂດຍອີງໃສ່ລໍາດັບ nanopore, ເປີດເຜີຍໃຫ້ເຫັນລະບຽບການຫຼຸດລົງຂອງໂລກຂອງ IR isoforms ໃນ SF3B1K700E.
ອ້າງອິງ
Tang AD , Soulette CM , Baren MJV , et al.ລັກສະນະການຖອດຂໍ້ຄວາມແບບເຕັມຄວາມຍາວຂອງການກາຍພັນຂອງ SF3B1 ໃນ leukemia lymphocytic ຊໍາເຮື້ອສະແດງໃຫ້ເຫັນການຫຼຸດລົງຂອງ introns ເກັບຮັກສາໄວ້ [J].ການສື່ສານທໍາມະຊາດ.