GENOME Evolutionis
geneticae naturae
Summus qualitas genome conventus elucidat indolem genomicam siliginis et genesis agronomically magni momenti
PacBio |Illumina |Bionano Optical Map |Hi-C Genome Conventus |Genetic Map |Selective Sweeps |RNA-Seq |ISO-seq |SLAF-seq
Biomarker Technologies subsidium technicum in Pacbio sequendo, Hi-C sequendo et analysin in hoc studio dedit.
Quisque ornare
1. Prima chromosomatis qualitas Siliginis genome alta est, quae una magnitudine chromosomatum ampliorem habet quam 1 Gb.
2. Cum Tu, Aet et Hv genome comparata, singularis eventus recentis LTR-RT in Sili genome observatus est, quod author erat pro extensione magnitudinis genome siliginis.
3. Distinctio siliginis et diploidis inter triticum post separationem hordei a tritico fiebat, disiunctio temporum pro duobus eventis circiter 9.6 et 15 MYA.
FT genes phosphorylation may control the early heading trait in siligo.
4. Analysis selectivae scopas indicant possibilis implicationem ScID1 in moderatione capitis date et probabilis lectio per domesticationem in siligine.
Background
Background
Siligo est magni pretii cibus et seges pabuli, magna subsidia genetica pro tritico et triticali melioratione, et necessaria materia ad efficaces studiorum genomicorum comparativorum in herbis.Siligine Weinging, florentis varietas antiqua in Sinis culta, praestantissima est ob latum spectrum eius resistentia utrique rubigine pulverulenta et livore rubigo.Ad intelligendum geneticum et hypotheticum fundamentum siligo elite lineamenta et ad studia genomica et admissura promovenda in segetibus siligine et affinibus, hic secuntur et enucleantur genome Weining siliginis.
Res gestae
Siliginis Genome
Siliginis genome componendo PacBio SMRT constructum est, legit, brevem Illumina sequentem, tum quae ex chromatin conformatione ceperunt (Hi-C), tabularum geneticarum, et Analysis BioNano.Conglobata contigs (7.74 Gb) pro 98.47% magnitudine genome aestimatae (7.86 Gb), cum 93.67% contigorum (7.25 Gb) septem chromosomatum assignata.Repetita elementa 90,31% conglobatae genomae constituuntur.
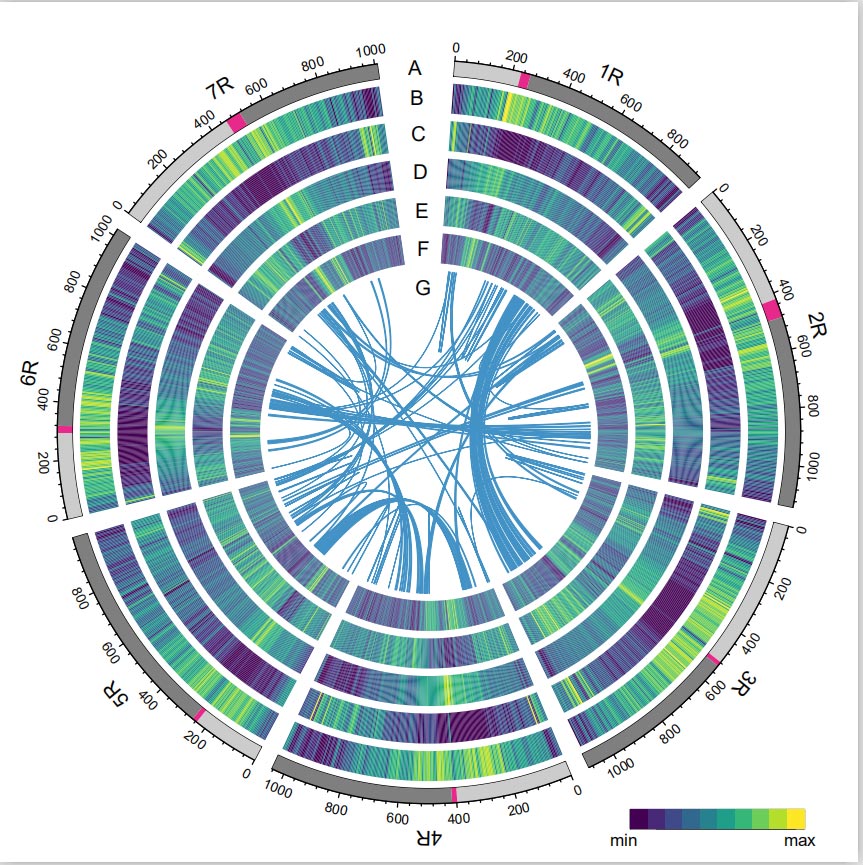
Siliginis Genome
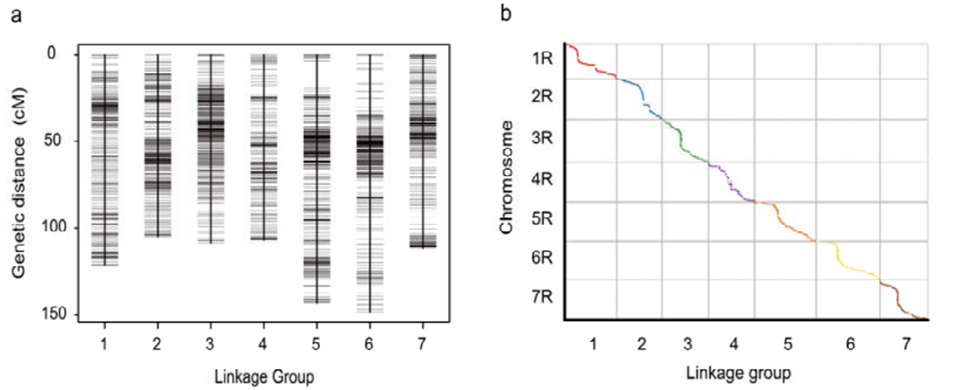
Tabula geographica genetica (WJ) elaborata utens 295 F2 plantarum ex transitu duarum landraceorum siliginis (Weining × Jingzhou)
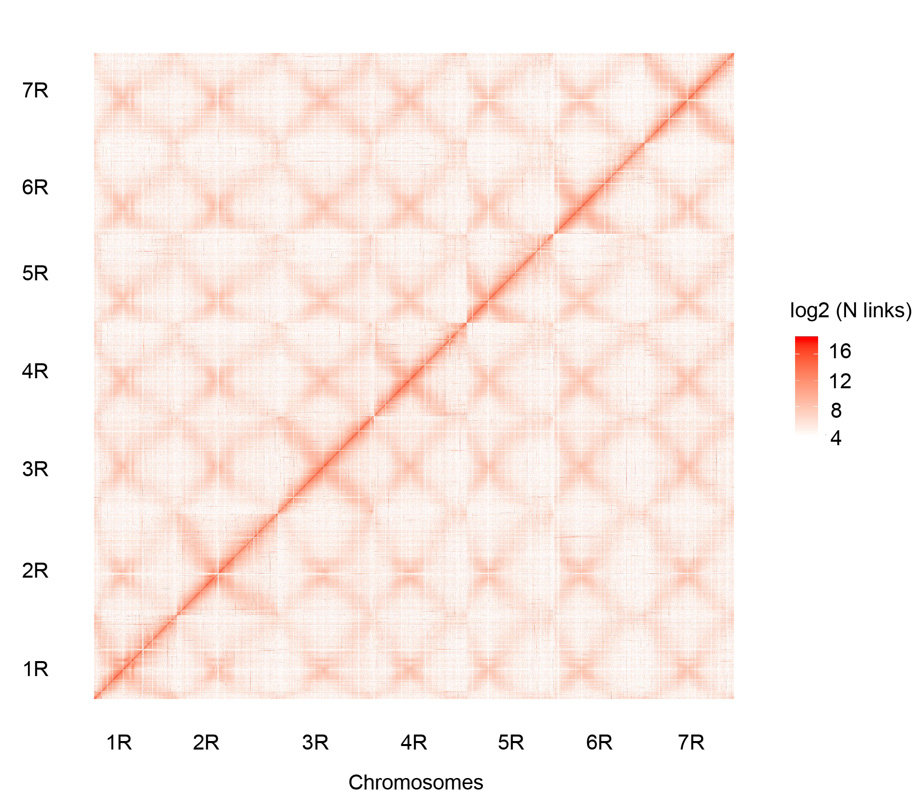
Hi-C map septem contactuum convenerunt Weining chromosomatum siliginis (1R – 7R)
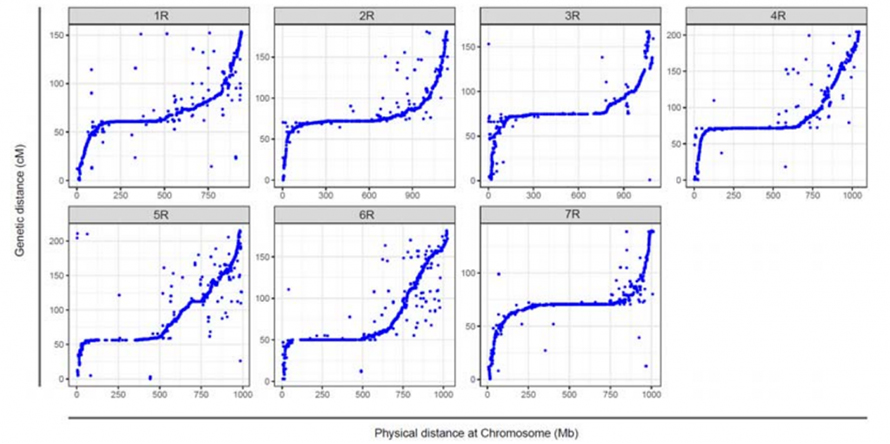
Noctis inter septem chromosomata conglobata de Weining siligine et septem coetibus siliginis iunctis elaboratis utens Lo7 x Lo255 RIL population
Index Conventus LTR (LAI) valor genome Rye repertus est 18.42 et 1,393 (96.74%) ex 1,440 generum BUSCO valde conservatorum notus est. Hi eventus suggerunt seriem genome siligo Weining optimum esse in utroque intergenico. et regionum genics.Summa 86,991 genes interdum coding, inter 45,596 genes alta fiducia (HC) et 41,395 genes humilis fiduciae praedictae sunt.
2. Analysis of TEs
Analysis of TEs.Multitudo 6.99 Gb, 90,31% conventus Weiningorum repraesentans, TEs annotatum est, quod 2,671,941 elementa ad 537 familias pertinentia comprehenderunt.Hoc TE contentum evidenter eminebat quam antea nuntiaverat Ta (84.70%), Tu (81.42%), Aet (84.40%), WEW (82.20%), vel Hv (80.80%).Longae terminales repetitae retrotransposones (LTR-RTs), inter quas Gypsy, Copia et elementa RT non classificata, dominabantur TEs, et 84.49% contentorum TE contentorum annotatorum et 76,29% conglobatorum genome Weiningensis erant occupatae;CACTA DNA transposones erant secundi TEs copiosissimi, constituentes 11.68% contentum TE annotatum et 10.55% conglobatum genome Weining.
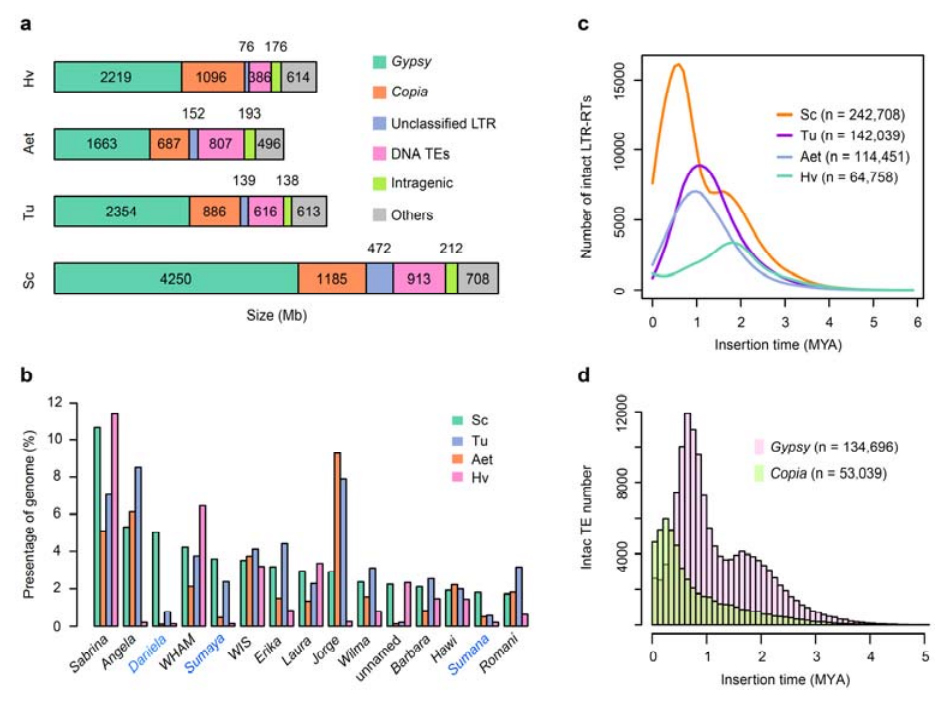
Analysis transposon elementis siliginis
Siliginis Weining comparative altam proportionem habuit recentium insertionum LTR-RTs cum cacumine amplificationis apparuit circiter 0,5 miliones abhinc annos (MYA), quae recentissima inter quattuor species erat;alterum cacumen, circiter 1.7 MYA occurrit, antiquiorem et etiam in hordeo vidi.In plano superfamiliae recentissimae copiae copiae in Weining siligine ad 0.3 MYA inventa sunt, dum amplificationes Gypsy RTs dominanter formaverunt distributionem bimodalem dynamicorum LTR-RT ruptorum.
3. Inquisitio evolutionis genome siliginis et synteniae chromosomatum
Distinctio siliginis et diploidis inter tritica facta est post separationem hordei a tritico, cum disiunctio temporum pro duobus eventis circiter 9.6 et 15 MYA, respective.1R, 2R, 3R omnino collineantur cum coetibus 1, 2 et 3 chromosomatum tritici, respective.4R, 5R, 6R, 7R inventae sunt fusions magna-scalarum et segmenta.
4. Analysis generum duplicationes et earum impulsum in biosynthesi genes amyli
Egregie, numeri genesis tandem duplicati (TDGs) et genesis (PDGs) Weining siliginis proxime duplicati tam superiores fuerunt quam illi qui pro Tu, Aet, Hv, Bd et Os.Genera duplicata (TrDGs) transposita fuerunt etiam plures quam illae quae nominatim Tu et Aet inventae sunt.Dilatatio siliginis genome comitatur numeros gena duplicationes superiores.Auctus TE siligine inruptus ad numerum elevatum TrDGs perduxisset.
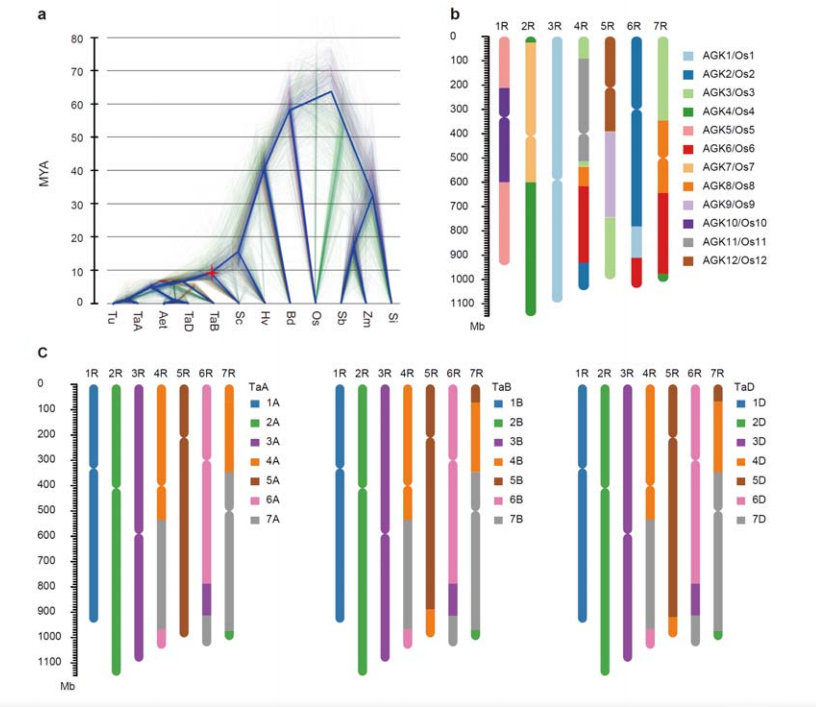
Syntenia evolutionis et chromosomatum analyses genome siliginis
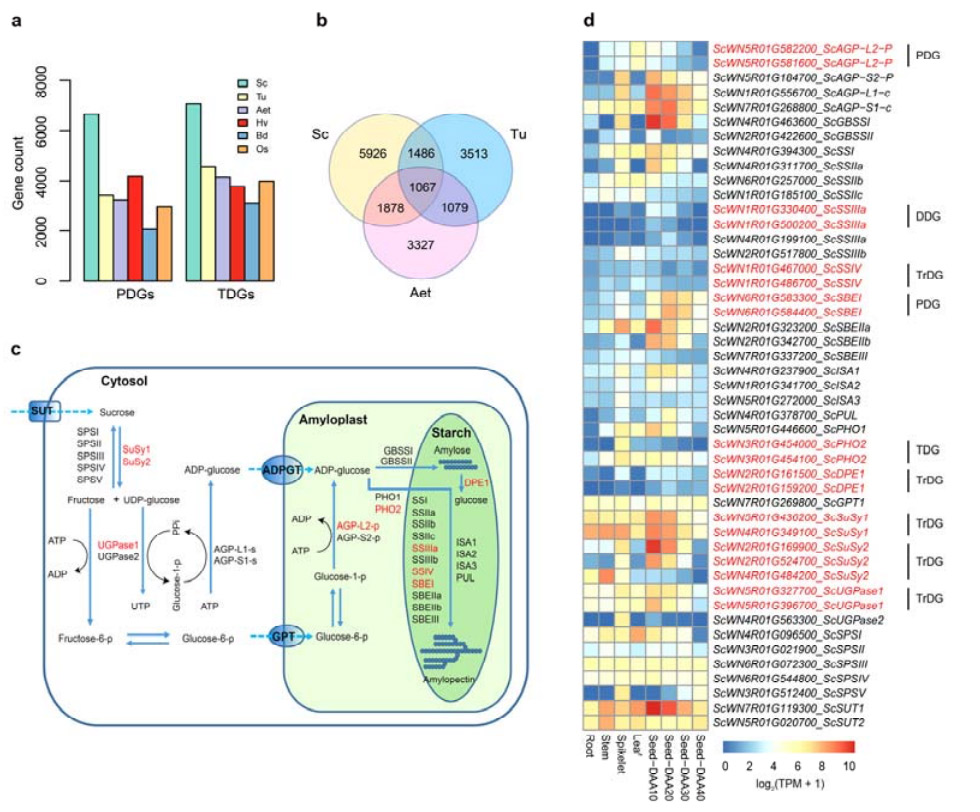
Analysis siliginis gene duplicationum earumque impulsum in diversitatibus genesis amyli biosynthesis cognatae (SBRGs)
5. Dissectio seminis siliginis seminis repositionis dapibus (SSP) gene loci
Quattuor loci chromosomales (sec-1 ad Sec-4) rationem siliginis SSPs notati sunt in 1R vel 2R.α-gliadin gena nuper in tritico solum evoluta sunt, et species propinqua post divergentiam tritici ex siligine.
6. Examen transcriptionis factoris et morbi resistentiae genes
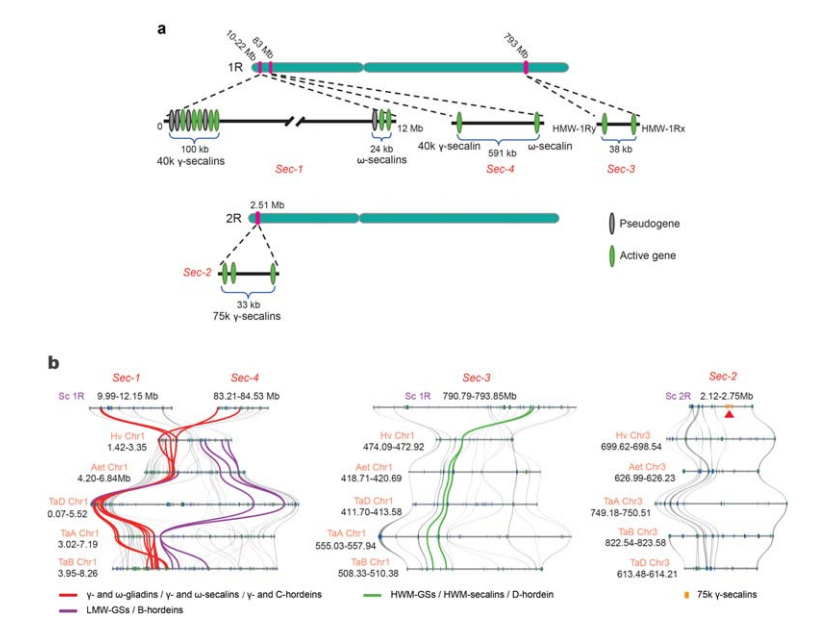
Analysis siliginis secalin loci
Weining siligo plures morbos genesis (DRA) resistentia habuit (1,989, Accessiones Data 3) quam Tu (1,621), Aet (1,758), Hv (1,508), Bd (1,178), Os (1,575), et A (1,836). ) B (1,728) et D (1,888) subgenomes tritici communis.
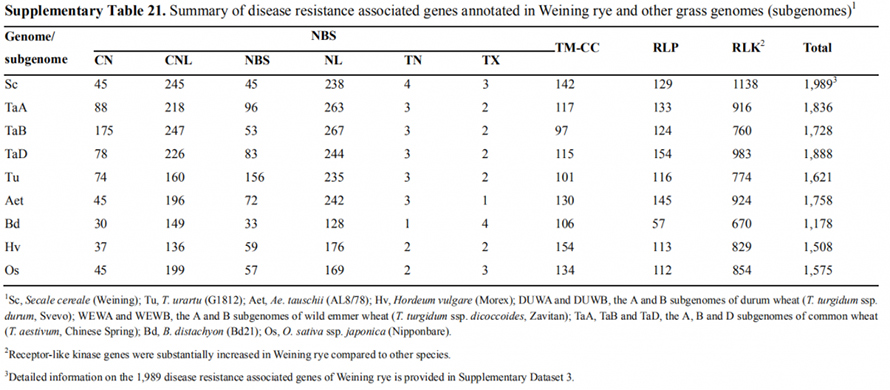
7. Investigatio gene expressionis lineamenta cum primo capite quivis
Duo FT genes relative alta expressione sub longis conditionibus, ScFT1 et ScFT2, in Weining genome conventu annotatae sunt.Duae residua amino acidorum ScFT2 (S76 et T132) inventae sunt relationem phosphorylationi cum demissione temporis imperium
Progressus et gene expressio lineamenta cum primo capite lineamentum Weining siliginis
8. Mining of chromosomal regionum et locorum potentialiter implicati in domesticis siliginis
Summa 123.647 SNPs usus est analysis selevtivae ducere inter culturam siliginis et S. vavilovii.11 scopa selectiva signa quae per reductionem index (DRI), index fixationis (FST) et methodus XP-CLR notantur.ScID1 inventum possibilem esse in constitutione capitis date.
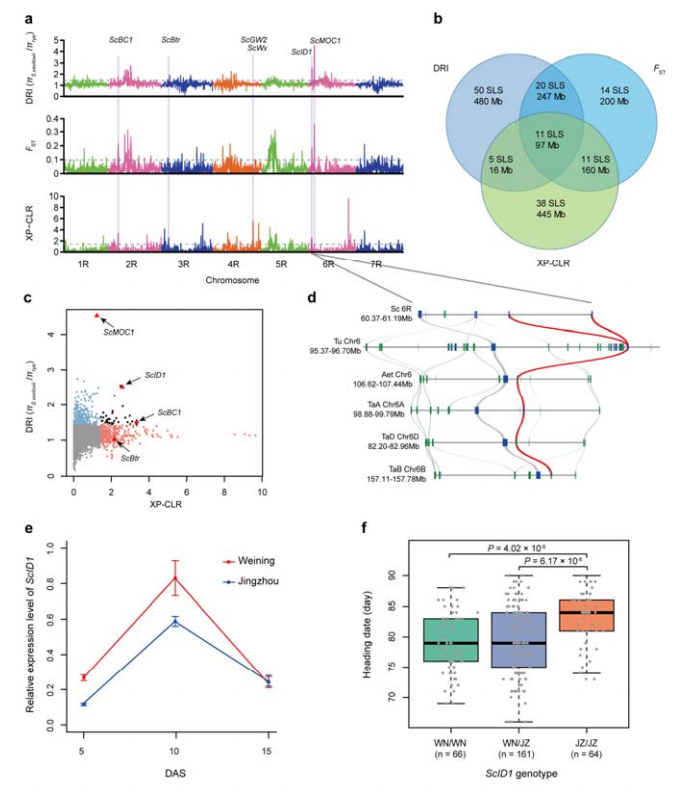
Sativum et analysis regionum chromosomalium et locorum potentialiter ad domesticationem siliginis pertinentia
Reference
Li GW et al.Princeps qualitas genome conventus elucidat indolem genomicam siliginis et genes agronomicae momenti.Natura Genetics (2021).
News and Highlights intendit ad communicandas causas novissimas prosperos cum Biomarker Technologies, res gestas scientificas novas capiendo necnon artes principales in studio applicatas.
Post tempus: Jan-05-2022

