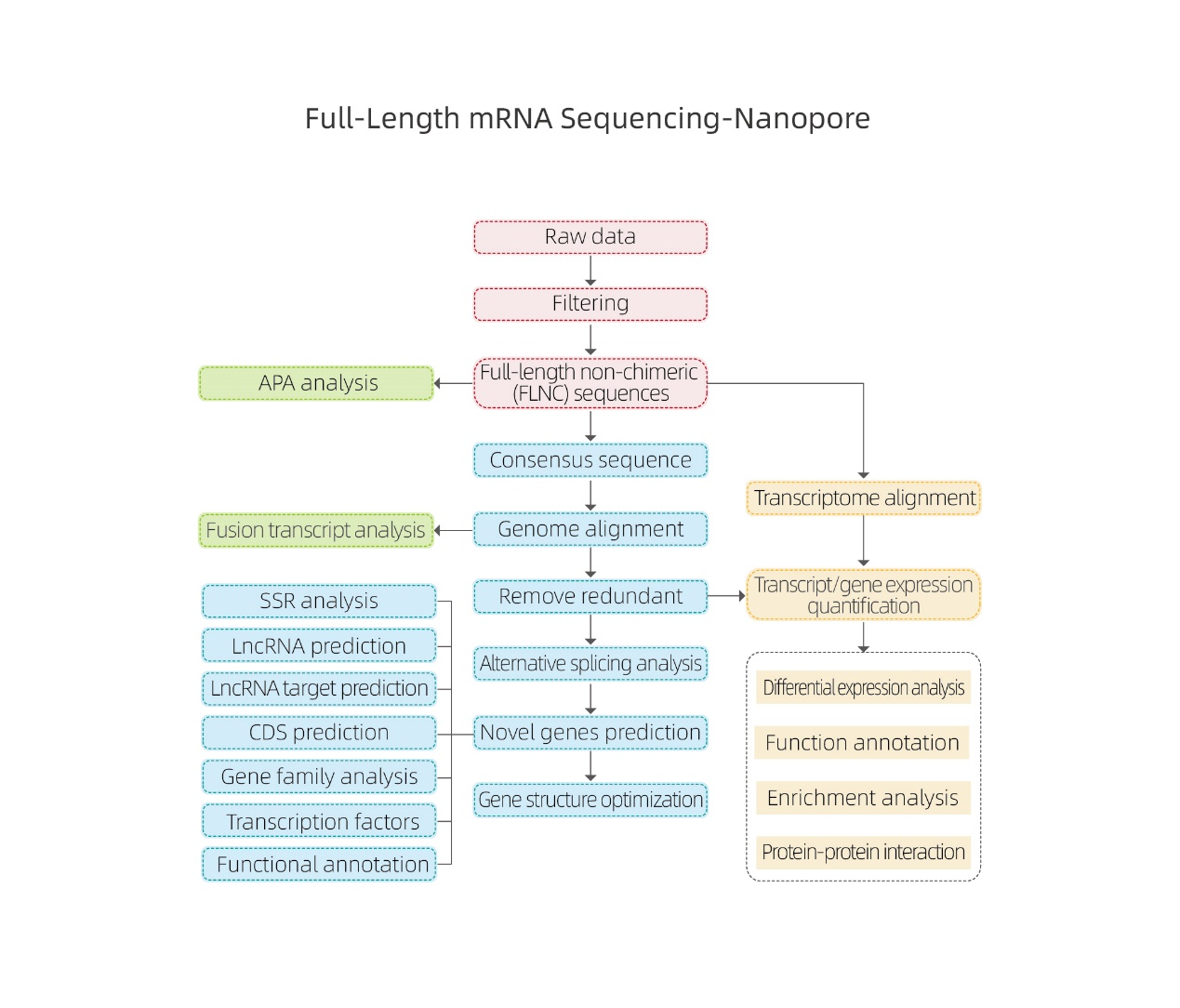
ប្រវែងពេញ mRNA Sequencing-Nanopore
អត្ថប្រយោជន៍សេវាកម្ម
● ភាពលំអៀងនៃលំដាប់ទាប
● បង្ហាញម៉ូលេគុល cDNA ពេញប្រវែង
● ត្រូវការទិន្នន័យតិចដើម្បីគ្របដណ្តប់ចំនួនប្រតិចារិកដូចគ្នា។
● ការកំណត់អត្តសញ្ញាណអ៊ីសូហ្វមច្រើនក្នុងមួយហ្សែន
● បរិមាណកន្សោមក្នុងកម្រិត isoform
លក្ខណៈបច្ចេកទេសនៃសេវាកម្ម
| បណ្ណាល័យ | វេទិកា | ទិន្នផលទិន្នន័យដែលបានណែនាំ (Gb) | ការត្រួតពិនិត្យគុណភាព |
| cDNA-PCR (Poly-A សំបូរទៅដោយ) | Nanopor PromethION P48 | 6 Gb / គំរូ (អាស្រ័យលើប្រភេទសត្វ) | សមាមាត្រប្រវែងពេញ 70% ពិន្ទុគុណភាពមធ្យម៖ Q10
|
ការវិភាគជីវវិទ្យា
●ដំណើរការទិន្នន័យឆៅ
● ការកំណត់អត្តសញ្ញាណប្រតិចារិក
● ការភ្ជាប់ជម្មើសជំនួស
● បរិមាណនៃការបញ្ចេញមតិក្នុងកម្រិតហ្សែន និងកម្រិតអ៊ីសូហ្វម
● ការវិភាគការបញ្ចេញមតិឌីផេរ៉ង់ស្យែល
● ចំណារពន្យល់មុខងារ និងការពង្រឹង (DEGs និង DETs)
តម្រូវការគំរូ និងការដឹកជញ្ជូន
តម្រូវការគំរូ៖
នុយក្លេអូទីត៖
| Conc.(ng/μl) | បរិមាណ (μg) | ភាពបរិសុទ្ធ | សុចរិតភាព |
| ≥ 100 | ≥ 0.6 | OD260/280=1.7-2.5 OD260/230=0.5-2.5 មានកំណត់ ឬគ្មានការចម្លងរោគ DNA បង្ហាញនៅលើជែល។ | សម្រាប់រុក្ខជាតិ៖ RIN≥7.0; សម្រាប់សត្វ: RIN≥7.5; 5.0≥28S/18S≥1.0; កម្រិតមូលដ្ឋាន ឬគ្មានការលើកឡើង |
ជាលិកា៖ ទំងន់ (ស្ងួត)៖ ≥1 ក្រាម។
*សម្រាប់ជាលិកាដែលតូចជាង 5mg យើងសូមណែនាំឱ្យផ្ញើគំរូជាលិកាដែលកក (ក្នុងអាសូតរាវ) flash ។
ការផ្អាកកោសិកា៖ ចំនួនកោសិកា = 3 × 106- 1 × 107
* យើងសូមផ្តល់អនុសាសន៍ឱ្យដឹកជញ្ជូន lysate កោសិកាទឹកកក។ក្នុងករណីដែលក្រឡានោះតូចជាង 5 × 105, flash បង្កកក្នុងអាសូតរាវត្រូវបានណែនាំ ដែលល្អសម្រាប់ការទាញយកខ្នាតតូច។
គំរូឈាម៖ បរិមាណ≥ 1 មីលីលីត្រ
ការចែកចាយគំរូដែលបានណែនាំ
កុងតឺន័រ: បំពង់ centrifuge 2 មីលីលីត្រ (ក្រដាសសំណប៉ាហាំងមិនត្រូវបានណែនាំទេ)
ការដាក់ស្លាកគំរូ៖ ក្រុម + ចម្លងឧទាហរណ៍ A1, A2, A3;B1, B2, B3 ......
ការដឹកជញ្ជូន៖ 2, ទឹកកកស្ងួត៖ គំរូត្រូវវេចខ្ចប់ក្នុងថង់ ហើយកប់ក្នុងទឹកកកស្ងួត។
- បំពង់ RNAstable: សំណាក RNA អាចត្រូវបានសម្ងួតនៅក្នុងបំពង់ស្ថេរភាព RNA (ឧទាហរណ៍ RNAstable®) និងដឹកជញ្ជូនក្នុងសីតុណ្ហភាពបន្ទប់។
លំហូរការងារសេវាកម្ម
នុយក្លេអូទីត៖

ការចែកចាយគំរូ

ការសាងសង់បណ្ណាល័យ

លំដាប់

ការវិភាគទិន្នន័យ

សេវាកម្មក្រោយពេលលក់
លំហូរការងារសេវាកម្ម
ជាលិកា៖

ការរចនាពិសោធន៍

ការចែកចាយគំរូ

ការទាញយក RNA

ការសាងសង់បណ្ណាល័យ

លំដាប់

ការវិភាគទិន្នន័យ

សេវាកម្មក្រោយពេលលក់
1.ការវិភាគការបញ្ចេញមតិឌីផេរ៉ង់ស្យែល - គ្រោងភ្នំភ្លើង
ការវិភាគកន្សោមឌីផេរ៉ង់ស្យែលអាចត្រូវបានដំណើរការក្នុងកម្រិតហ្សែនទាំងពីរដើម្បីកំណត់អត្តសញ្ញាណហ្សែនដែលបង្ហាញដោយឌីផេរ៉ង់ស្យែល (DEGs) និងក្នុងកម្រិត isoform ដើម្បីកំណត់អត្តសញ្ញាណឌីផេរ៉ង់ស្យែល។
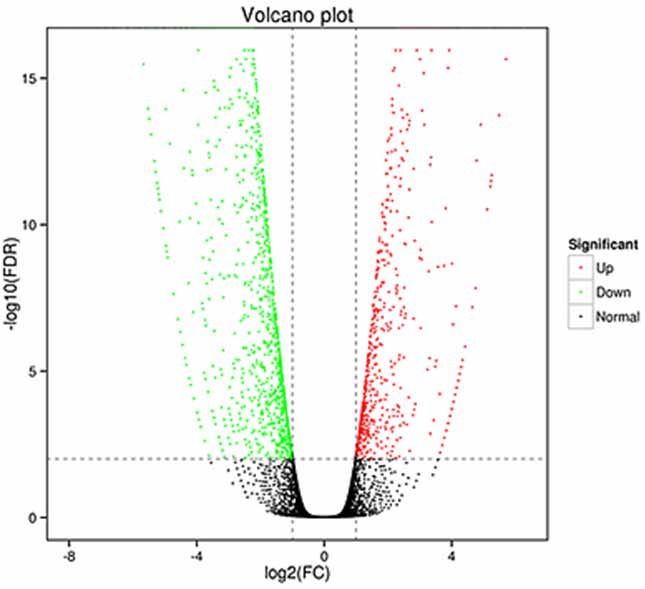
ប្រតិចារិកដែលបានសម្តែង (DETs)
2.ផែនទីកំដៅចង្កោមឋានានុក្រម
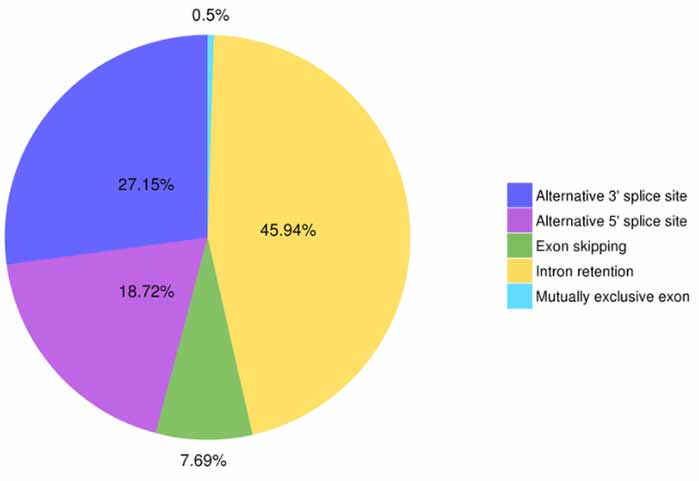
3. ការកំណត់អត្តសញ្ញាណ និងការចាត់ថ្នាក់ជំនួស
ព្រឹត្តិការណ៍បំបែកជម្រើសប្រាំប្រភេទអាចត្រូវបានព្យាករណ៍ដោយ Astalavista ។
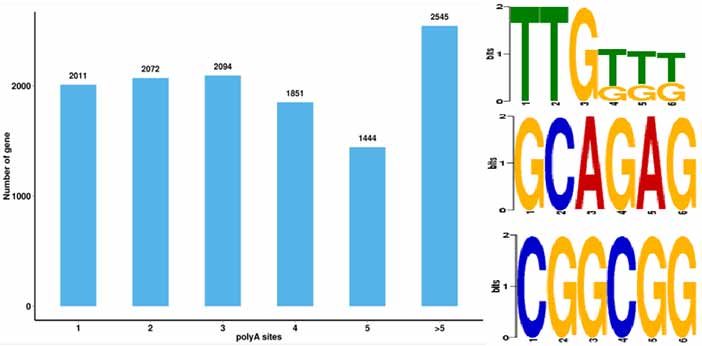
4.ការកំណត់អត្តសញ្ញាណព្រឹត្តិការណ៍ poly-adenylation ជំនួស (APA) និង Motif នៅ 50 bp ខាងលើនៃ poly-A
ករណី BMK
ការកំណត់អត្តសញ្ញាណជំនួស និងការកំណត់កម្រិត isoform ដោយ nanopore full-length transscriptome sequencing
បោះពុម្ពផ្សាយ៖ទំនាក់ទំនងធម្មជាតិឆ្នាំ ២០២០
យុទ្ធសាស្ត្រតម្រៀប៖
ការដាក់ជាក្រុម៖ 1. CLL-SF3B1(WT);2. ការផ្លាស់ប្តូរ CLL-SF3B1(K700E);3. កោសិកា B ធម្មតា។
យុទ្ធសាស្ត្រលំដាប់លំដោយ៖ ការរៀបចំបណ្ណាល័យ 2D តូច, តម្រៀបបណ្ណាល័យ PromethION 1D;ទិន្នន័យខ្លីៗពីគំរូដូចគ្នា។
វេទិកាលំដាប់លំដោយ៖ Nanopore MinION;ណាណូប៉ូ ប្រូមេទីយ៉ូន;
លទ្ធផលសំខាន់
1.Isoform-level Alternative Splicing Identification
លំដាប់ដែលបានអានយូរ ផ្តល់អំណាចដល់ការកំណត់អត្តសញ្ញាណនៃ SF3B1 ដែលផ្លាស់ប្តូរK700E- ផ្លាស់ប្តូរទីតាំង splice នៅកម្រិត isoform ។35 ជម្រើស 3′SSs និង 10 ជម្រើស 5′SSs ត្រូវបានរកឃើញថាត្រូវបានបំបែកយ៉ាងសំខាន់រវាង SF3B1K700Eនិង SF3B1WT.ការកែប្រែចំនួន 33 ក្នុងចំណោម 35 ត្រូវបានគេរកឃើញថ្មីដោយលំដាប់ដែលបានអានជាយូរមកហើយ។
2.Isoform-level Alternative Splicing quantification
ការបង្ហាញនៃ isoforms ការរក្សាទុក intron (IR) នៅក្នុង SF3B1K700Eនិង SF3B1WTត្រូវបានគេកំណត់បរិមាណដោយផ្អែកលើលំដាប់ nanopore ដែលបង្ហាញពីការចុះក្រោមជាសកលនៃ isoforms IR នៅក្នុង SF3B1K700E.
ឯកសារយោង
Tang AD, Soulette CM, Baren MJV, et al.ការកំណត់លក្ខណៈប្រតិចារឹកពេញប្រវែងនៃការផ្លាស់ប្តូរ SF3B1 នៅក្នុងជំងឺមហារីកឈាម lymphocytic រ៉ាំរ៉ៃបង្ហាញឱ្យឃើញពីការថយចុះនៃ introns ដែលបានរក្សាទុក[J]។ទំនាក់ទំនងធម្មជាតិ។