Secuenciación de ARN pequeno-Illumina
características
● Selección do tamaño do ARN antes da preparación da biblioteca
● Análise bioinformática centrada na predición de miRNA e os seus obxectivos
Vantaxes do servizo
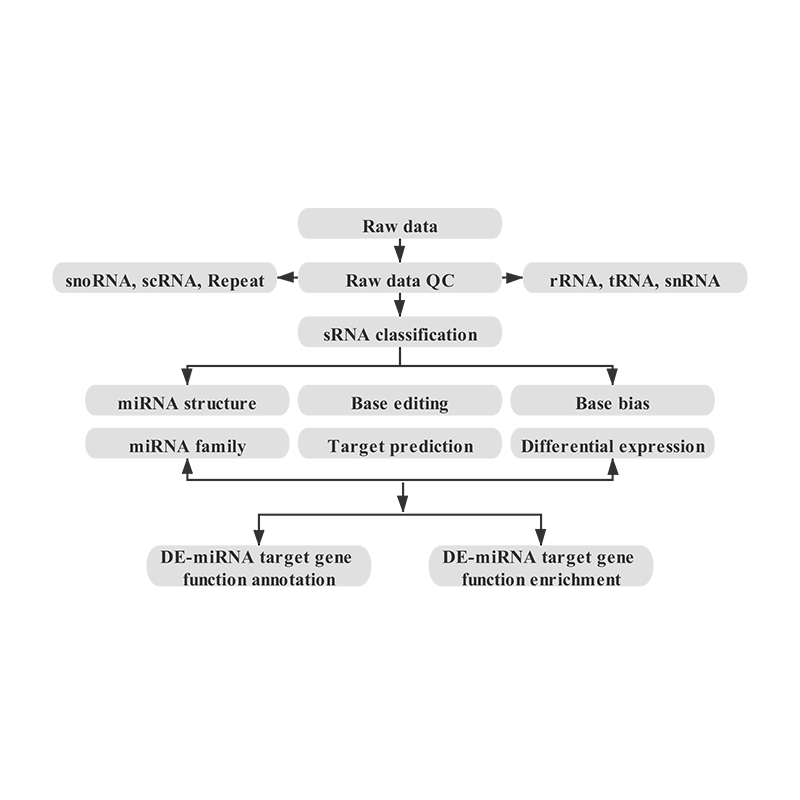
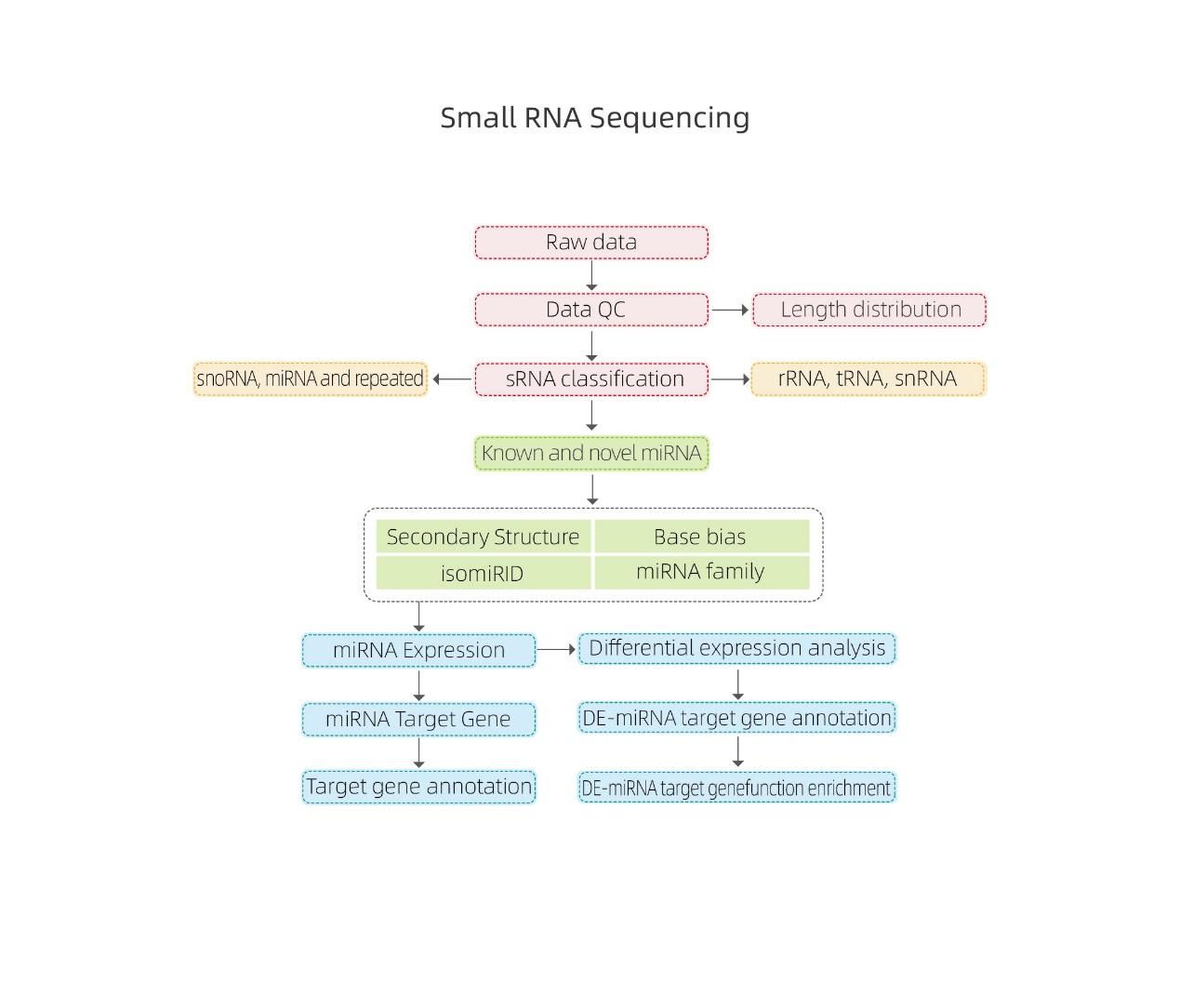
●Análise bioinformática integral:permitindo a identificación de miRNAs coñecidos e novos, identificación de obxectivos de miRNAs e a correspondente anotación funcional e o enriquecemento con múltiples bases de datos (KEGG, GO)
●Control de Calidade Riguroso: implementamos puntos de control básicos en todas as etapas, desde a preparación de mostras e bibliotecas ata a secuenciación e a bioinformática.Este seguimento minucioso garante a entrega de resultados de alta calidade constantemente.
●Soporte posvenda: O noso compromiso vai máis aló da finalización do proxecto cun período de servizo posvenda de 3 meses.Durante este tempo, ofrecemos seguimento do proxecto, asistencia para a resolución de problemas e sesións de preguntas e respostas para resolver calquera dúbida relacionada cos resultados.
●Amplia experiencia: cun historial de pechar con éxito varios proxectos de ARNs que abarcan máis de 100 especies en varios dominios de investigación, o noso equipo achega unha gran experiencia a cada proxecto.
Requisitos de mostra e entrega
| Biblioteca | Plataforma | Datos recomendados | QC de datos |
| Tamaño seleccionado | Illumina SE50 | 10M-20M lecturas | Q30≥85% |
Requisitos de mostra:
Nucleótidos:
| Conc. (ng/μl) | Cantidade (μg) | Pureza | Integridade |
| ≥ 80 | ≥ 0,5 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Contaminación limitada ou nula de proteínas ou ADN no xel. | RIN≥6,5; 5,0≥28S/18S≥1,0; elevación de referencia limitada ou nula |
● Plantas:
Raíz, talo ou pétalo: 450 mg
Folla ou semente: 300 mg
Froita: 1,2 g
● Animal:
Corazón ou Intestino: 450 mg
Vísceras ou cerebro: 240 mg
Músculo: 600 mg
Ósos, cabelo ou pel: 1,5 g
● Artrópodos:
Insectos: 9 g
Crustáceos: 450 mg
● Sangue enteiro: 2 tubos
● Células: 106 células
● Soro e Plasma:6 ml
Entrega de mostras recomendada
Envase:
Tubo de centrífuga de 2 ml (non se recomenda papel de aluminio)
Etiquetaxe da mostra: grupo+réplica, por exemplo, A1, A2, A3;B1, B2, B3....
Envío:
1. Xeo seco: as mostras deben ser embaladas en bolsas e enterradas en xeo seco.
2.Tubos RNAstable: as mostras de ARN pódense secar nun tubo de estabilización de ARN (por exemplo, RNAstable®) e enviarse a temperatura ambiente.
Fluxo de traballo do servizo

Deseño de experimentos

Entrega da mostra

extracción de ARN

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
Bioinformática
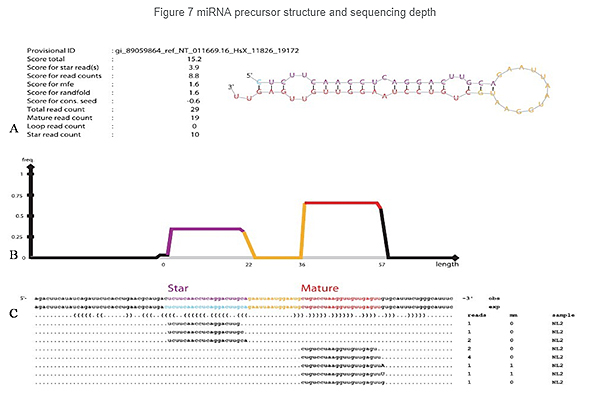
Identificación do miARN: estrutura e profundidade
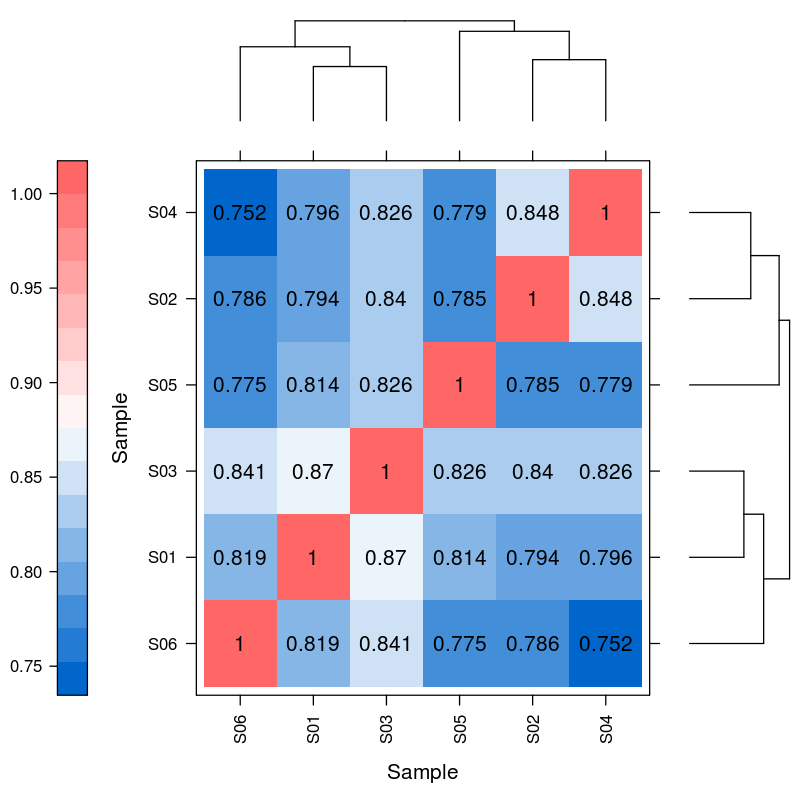
Expresión diferencial de miARN - agrupación jerárquica
Anotación funcional da diana de miARN expresados de forma diferencial
Explore os avances na investigación facilitados polos servizos de secuenciación de ARNs de BMKGene a través dunha colección de publicacións seleccionada.
Chen, H. et al.(2023) "As infeccións víricas inhiben a biosíntese e a fotosíntese da saponina en Panax notoginseng", Fisioloxía e Bioquímica Vexetal, 203, p.108038. doi: 10.1016/J.PLAPHY.2023.108038.
Li, H. et al.(2023) "A proteína FREE1 que contén o dominio FYVE da planta asóciase con compoñentes do microprocesador para reprimir a bioxénese do miARN", informa EMBO, 24 (1).doi: 10.15252/EMBR.202255037/SUPPL_FILE/EMBR202255037-SUP-0004-SDATAFIG4.TIF.
Yu, J. et al.(2023) "O MicroRNA Ame-Bantam-3p controla o desenvolvemento da pupa larvaria ao dirixirse ao xene 8 de dominios similares a factores de crecemento epidérmico múltiple (megf8) na abella, Apis mellifera", International Journal of Molecular Sciences, 24 (6), p. .5726. doi: 10.3390/IJMS24065726/S1.
Zhang, M. et al.(2018) "A análise integrada de MiRNA e xenes asociados á calidade da carne revela que Gga-MiR-140-5p afecta a deposición de graxa intramuscular en pollos", Fisioloxía celular e bioquímica, 46 (6), páxinas 2421-2433.doi: 10.1159/000489649.