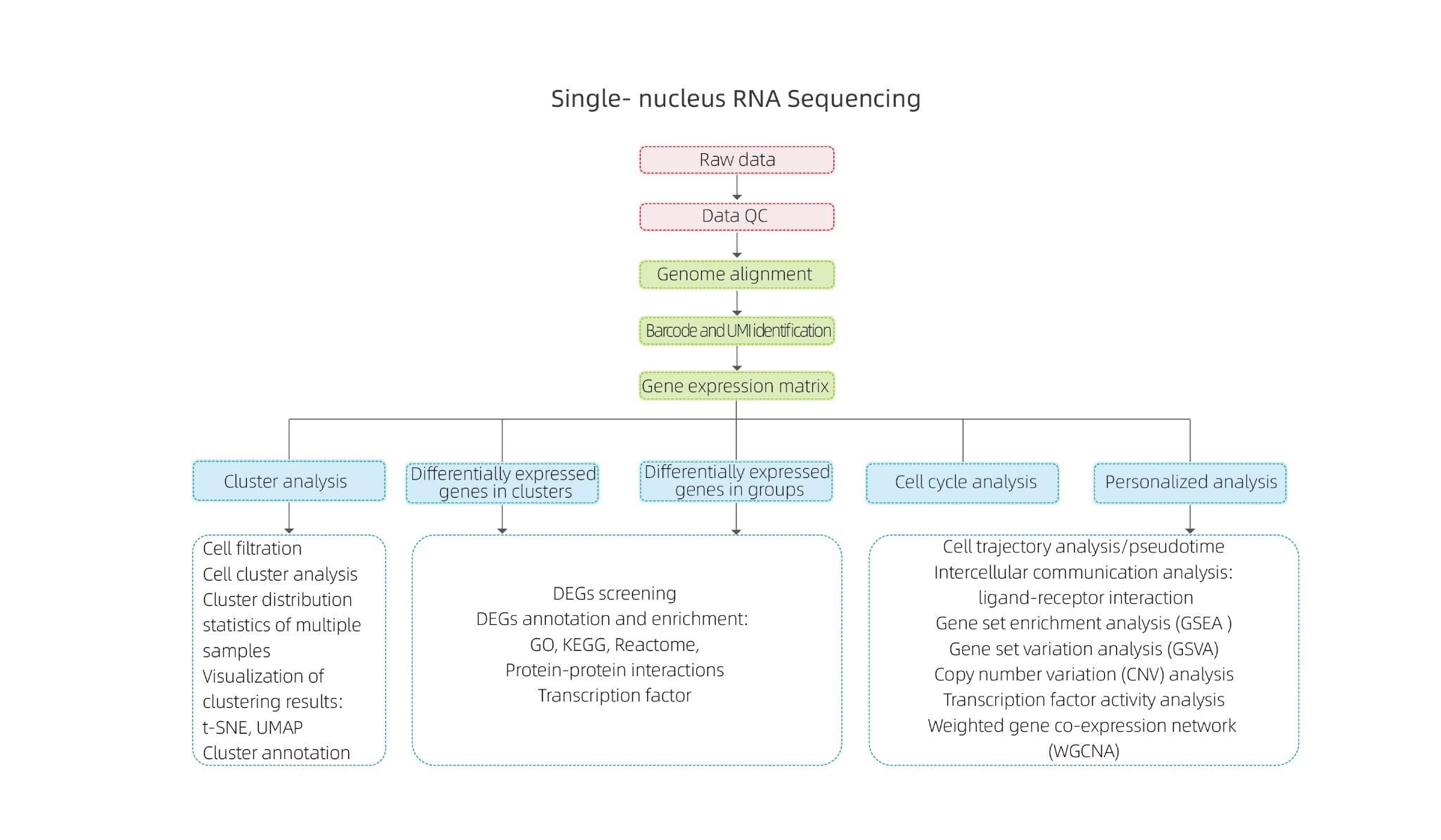
Secuenciación de ARN dun só núcleo
Principio técnico
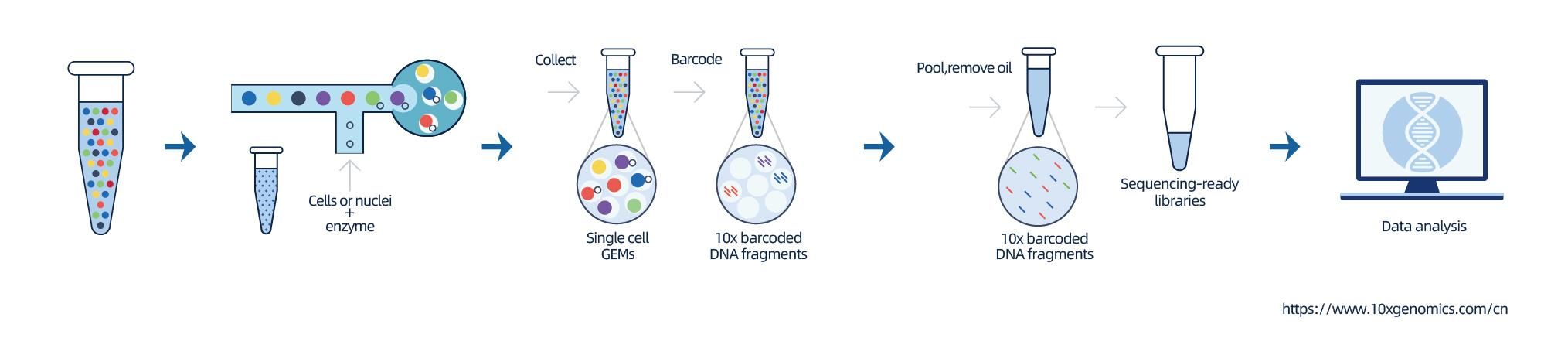
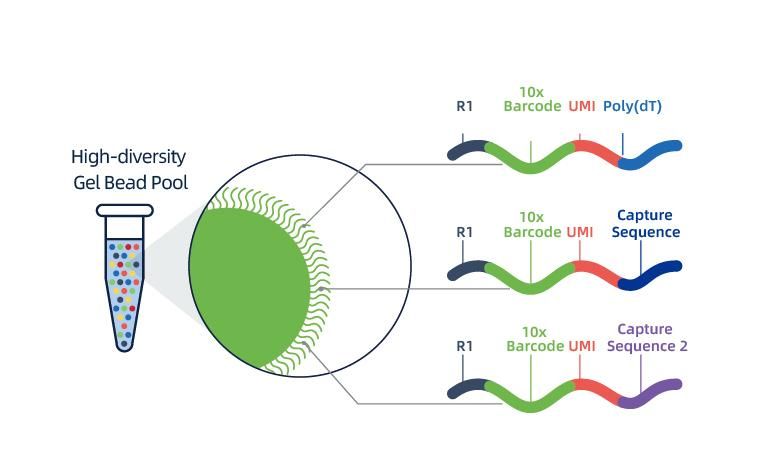
O illamento dos núcleos conséguese mediante 10× Genomics ChromiumTM, que consiste en un sistema microfluídico de oito canles con dobres cruzamentos.Neste sistema, unhas esferas de xel con códigos de barras e imprimación, encimas e un só núcleo están encapsulados nunha gota de aceite do tamaño dun nanolitro, xerando Gel Bead-in-Emulsion (GEM).Unha vez que se forman os GEM, realízase a lise celular e a liberación de códigos de barras en cada GEM.O ARNm transcríbense inversamente en moléculas de ADNc con códigos de barras 10× e UMI, que están ademais suxeitos á construción de bibliotecas de secuenciación estándar.
O tecido non é apto para a preparación dunha suspensión celular
| Célula/Tecido | Razón |
| Tecido conxelado non fresco | Non se poden obter organizacións novas ou gardadas desde hai moito tempo |
| Célula muscular, megacariocito, graxa... | O diámetro da cela é demasiado grande para entrar no instrumento |
| Fígado… | Demasiado fráxil para romper, incapaz de distinguir células individuais |
| Célula neuronal, cerebro... | Máis sensible, fácil de estresar, cambiará os resultados da secuenciación |
| Páncreas, Tiroides... | Rico en enzimas endóxenas, que afectan á produción de suspensión celular única |
Núcleo único vs
| Un único núcleo | Monocélula |
| Diámetro celular ilimitado | Diámetro celular: 10-40 μm |
| O material pode ser tecido conxelado | O material debe ser tecido fresco |
| Baixo estrés das células conxeladas | O tratamento con enzimas pode provocar reaccións de estrés celular |
| Non é necesario eliminar glóbulos vermellos | Os glóbulos vermellos deben ser eliminados |
| Nuclear expresa bioinformación | Toda a célula expresa bioinformación |
Especificacións do servizo
| Biblioteca | Estratexia de secuenciación | Volume de datos | Requisitos de mostra | Tecido |
| 10× Biblioteca de núcleo único de xenómica | 10x Genomics -Illumina PE150 | 100.000 lecturas/célula aprox.100-200 Gb | Número de móbil: > 2 × 105 Conc. celulara 700-1.200 células/μL | ≥ 200 mg |
Para obter máis detalles sobre a orientación de preparación de mostras e o fluxo de traballo do servizo, non dubide en falar con aExperto en BMKGENE
Fluxo de traballo do servizo

Deseño de experimentos

Entrega da mostra
Illamento dos núcleos

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
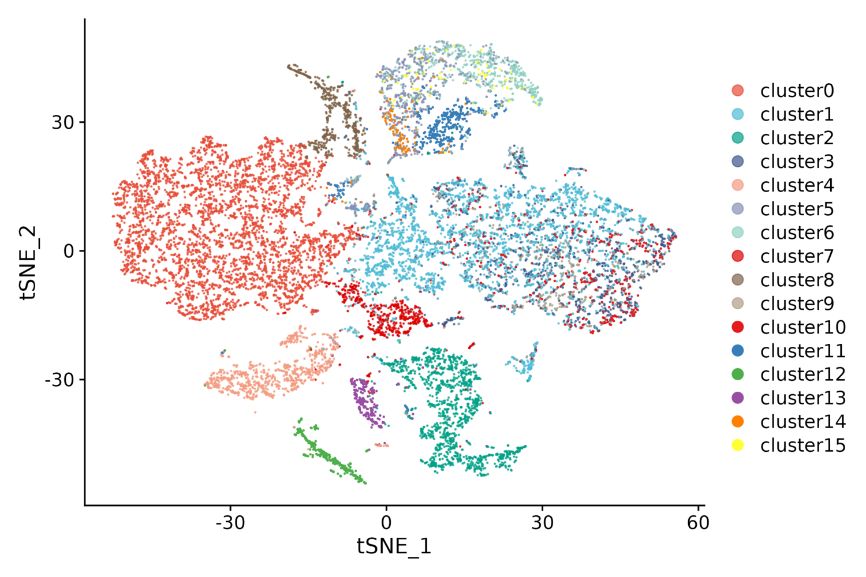
1.Agrupación puntual
2.Mapa de calor de agrupación de abundancia de expresións de marcadores
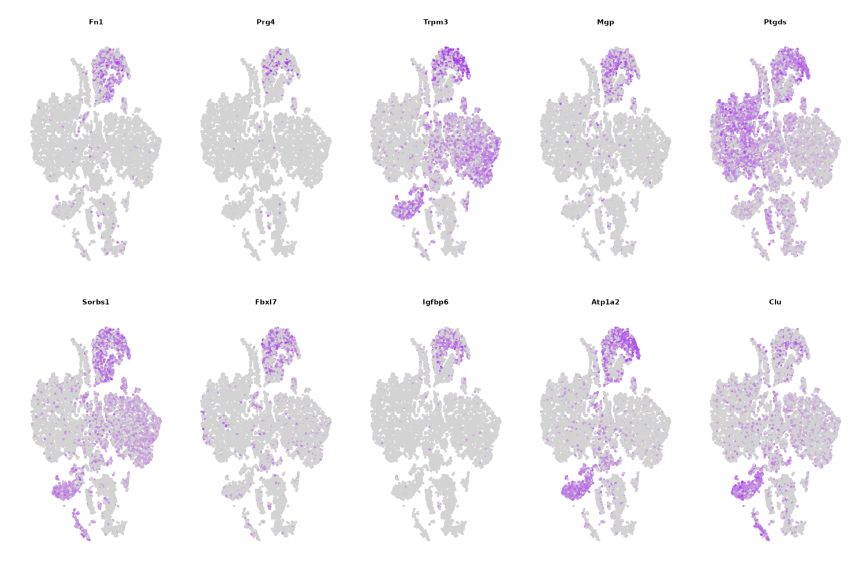
 3.Distribución de xenes Maker en diferentes clusters
3.Distribución de xenes Maker en diferentes clusters
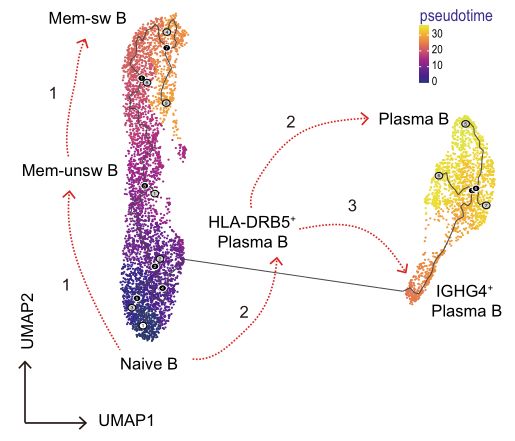
4.Análise da traxectoria celular/pseudotempo