
Mapa de calor
O ficheiro de datos da matriz úsase para o debuxo de mapas térmicos, que pode filtrar, normalizar e agrupar os datos da matriz.Utilízase principalmente para a análise de clusters do nivel de expresión xénica entre diferentes mostras.
Anotación xenética
A anotación da función xenética realízase mapeando secuencias no ficheiro FASTA con varias bases de datos.

Anotación xenética
Ferramenta básica de busca de aliñamento local

CDS_UTR_Predicción
Esta ferramenta está deseñada para predecir rexións codificantes (CDS) e rexións non codificantes (UTR) en determinadas secuencias de transcrición baseadas na explosión contra a base de datos de proteínas coñecidas e a predición de ORF.
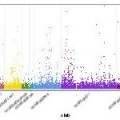
Trama Manhattan
O diagrama de Manhattan permite a visualización de datos cun gran número de puntos de datos.Úsase habitualmente nos estudos de asociación de todo o xenoma (GWAS).

Circos
O diagrama CIRCOS permite a presentación directa das distribucións de SNP, InDeL, SV e CNV no xenoma.

GO_Enriquecemento
TopGO é unha ferramenta deseñada para o enriquecemento funcional.O paquete TopGO-Bioconductor consiste en análise de expresión diferencial, análise de enriquecemento GO e visualización dos resultados.Xerará un cartafol con saída chamado "Gráfico", que contén resultados para topGO_BP, topGO_CC e topGO_MF.

WGCNA
WGCNA é un método de extracción de datos moi utilizado para descubrir módulos de coexpresión xénica.É aplicable a varios conxuntos de datos de expresión, incluíndo datos de microarrays e datos de expresión xénica orixinados a partir da secuenciación de próxima xeración.

InterProScan
Análise e clasificación de secuencias de proteínas InterPro

GO_KEGG_Enriquecemento
Esta ferramenta está deseñada para xerar histograma de enriquecemento GO, histograma de enriquecemento KEGG e vía de enriquecemento KEGG en función do conxunto xenético proporcionado e da anotación correspondente.
