XENOMÍA HUMANA
xenética da natureza
A secuenciación de lectura longa identifica expansións repetidas de GGC en NOTCH2NLC asociadas á enfermidade de inclusión intranuclear neuronal
Resecuenciación ONT |Ilumina |Secuenciación do exoma enteiro |Secuenciación dirixida a CRISPR-Cas9 ONT |ARN-seq |ONT 5mC chamada de metilación
Destacados
1. Mediante a análise de vinculación nunha gran familia de NIID, identificáronse dúas rexións vinculadas.
A secuenciación de lectura longa baseada en ONT e a secuenciación ONT de enriquecemento mediado por Cas-9 descubriu unha causa xenética potencial de expansións repetidas de NIID, GGC en 5′ UTR de NOTCH2NLC.Este estudo informou por primeira vez de expansións repetidas en xenes específicos humanos que evolucionaron a través de duplicacións segmentarias.
3. A secuenciación do ARN revelou transcritos antisentido anormais ao comezo ou dentro das rexións de expansión repetidas de GGC en NOTCH2NLC.
Fondo
NA enfermidade de inclusión intranuclear euronal (NIID) é unha enfermidade neurodexenerativa progresiva e mortal, que se caracteriza pola presenza de inclusións intranucleares hialinas eosinofílicas no sistema nervioso central e periférico.As súas manifestacións clínicas moi variables xeran grandes dificultades no diagnóstico ata a introdución da biopsia cutánea.Non obstante, os métodos baseados na histopatoloxía seguen sufrindo un diagnóstico erróneo, o que require unha comprensión xenética da NIID.
Logros
Análise de vinculación
SRealizouse a secuenciación do xenoma completo (WGS) e a secuenciación do exoma completo (WES) baseada en hort-read nunha gran familia de NIID (13 membros afectados e 7 non afectados).A análise de enlace nos SNP extraídos destes datos revelou só dúas rexións ligadas: unha rexión de 3,5 Mb en 1p36.31-p36.22 (LOD máximo = 2,32) e unha rexión de 58,1 Mb en 1p22.1-q21.3 (LOD máximo: 4,21). ).Non obstante, non se identificaron SNP ou CNV patóxenos nestas rexións vinculadas.
GGC repite as expansións en NOTCH2NLC
NA secuenciación baseada en anoporos foi procesada en 13 membros afectados e 4 non afectados de 8 familias (outro membro afectado foi secuenciado pola plataforma de secuenciación de lectura longa Pacbio).Os datos de lectura longas revelaron expansións repetidas de GGC asociadas á enfermidade na cartografía do xene NOTCH2NLC 5′ UTR na rexión ligada a 58,1 Mb (figura 1).Estas expansións repetidas tamén foron identificadas nos 40 casos esporádicos de NIID probados por RP-PCR.
CEmpregouse a secuenciación de obxectivos mediada por as-9 na plataforma de nanoporos para acadar unha maior cobertura de lectura na repetición NOTCH2NLC (100 X-1.795 X).Estas secuencias de consenso coincidiron ben cos descubrimentos anteriores sobre as expansións repetidas de GGC.Ademais, as repeticións {(GGA)n (GGC)n}n foron identificadas como un posible marcador xenético para o fenotipo dominante na debilidade (Figura 2).
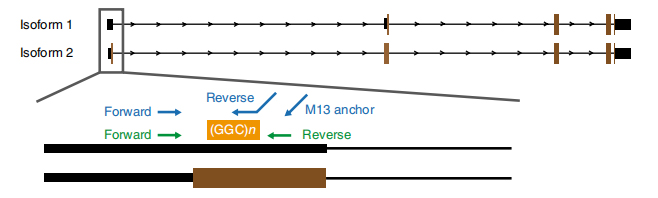
Figura 1. Expansión repetida asociada á enfermidade identificada no exón 1 das isoformas NOTCH2NLC.
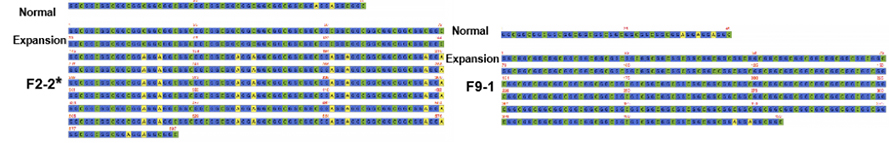
Figura 2. Secuencias consenso de repetición de NPTCH2NLC en pacientes con NIID con (*) ou sen fenotipo dominante de debilidade
NOs xenes OTCH2NL son xenes específicos de humanos, que se cre que desempeñan un papel vital na evolución do cerebro humano e nas enfermidades neurolóxicas.Non obstante, tres xenes relacionados con NOTCH2 (NOTCH2NLA, NOTCH2NLB e NOTCH2NLC) cunha identidade de secuencia > 99,1 % non se resolveron ata a última montaxe do xenoma humano.A secuenciación sen síntese e de lectura longa na plataforma de nanoporos mostrou vantaxes notables na resolución de rexións de gran semellanza e repeticións (GGC)n cun 100% rico en GC.
GGC repite as expansións en NOTCH2NLC
TA secuenciación do ranscriptoma foi procesada en 2 membros afectados e 2 non afectados.Calculouse a profundidade de lectura normalizada nas cadeas de sentido e antisentido en augas arriba dos primeiros exóns dos parálogos NOTCH2NL.Atopáronse transcricións anti-sentido anormais só nos casos afectados, que se sitúan ao comezo ou dentro da rexión de expansión repetida (picos morados en F1-14 e F1-16 na Figura 3).Ademais, identificáronse 54 DEG e todos foron enriquecidos en termos GO e MPO relacionados coas funcións neuronais.
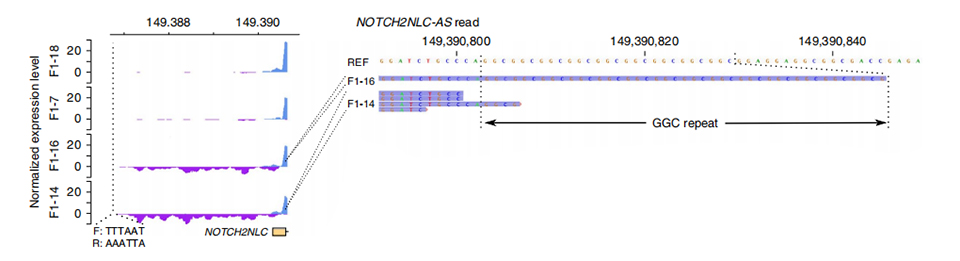
Figura 3. Profundidade de lectura normalizada augas arriba do primeiro exón de NOTCH2NLC nos casos non afectados (arriba) e afectados (abaixo).
Tecnoloxía
Oxford Nanopore Teghnologies (ONT)
NA secuenciación de anopores distínguese doutras plataformas de secuenciación, xa que os nucleótidos son lidos directamente sen proceso de síntese de ADN.A medida que unha única cadea de ADN pasa por un poro de proteína de tamaño nanométrico (nanoporo), diferentes nucleótidos xeran diferentes correntes iónicas, que poden ser capturadas e transferidas a unha secuencia de bases.A propia plataforma de secuenciación ONT non mostra un límite técnico aparente na duración da lectura do ADN.Polo tanto, as lecturas ultralongas (ULR) están dispoñibles para a ensamblaxe do xenoma de alta calidade.Ademais, estas lecturas extremadamente longas, que son o suficientemente longas como para cruzar características de secuencias complexas ou variacións estruturais, axudan a superar aquí as limitacións da secuenciación de lecturas curtas.

Secuenciación de nanoporos
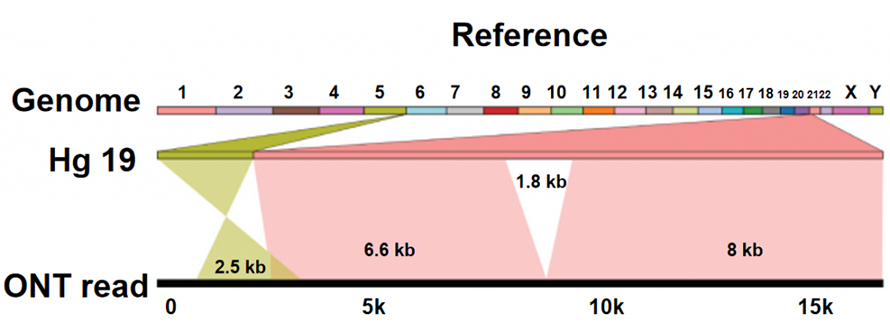
Identificación da variación de estrutura (SV).
SA secuenciación sen síntese conservou en gran parte a información de metilación do ADN no molde.A, T, C e G metilados xeran correntes iónicas distintas das non metiladas, que poden ser lidas directamente pola plataforma.A secuenciación de nanoporos permite o perfil do xenoma completo de 5 mC e 6 mA con resolución dun só nucleótido.
Referencia
Xuño Sone, et.al.A secuenciación de lectura longa identifica expansións repetidas de GGC en NOTCH2NLC asociadas á enfermidade de inclusión intranuclear neuronal.Xenética da natureza (2019)
Tecnoloxía e aspectos destacados ten como obxectivo compartir a aplicación exitosa máis recente de diferentes tecnoloxías de secuenciación de alto rendemento en varios ámbitos de investigación, así como ideas brillantes en deseño experimental e minería de datos.
Hora de publicación: 06-xan-2022

