TRANSCRIPTOMICA
natureza
COMUNICACIÓNS
A caracterización da transcrición completa da mutación SF3B1 na leucemia linfocítica crónica revela a regulación á baixa dos intróns retidos
Transcricións completas|Secuenciación de nanoporos|Análise de isoformas alternativas
Fondo
SAs mutacións omáticas no factor de splicing SF3B1 foron amplamente relacionadas con varios cancros, incluíndo a leucemia linfocítica crónica (LLC), o melanoma úveal, o cancro de mama, etc. Ademais, estudos transcriptómicos de lectura curta revelaron patróns de splicing aberrantes inducidos por mutacións de SF3B1.Non obstante, os estudos sobre estes patróns de empalme alternativos limitáronse durante moito tempo ao nivel de evento e á falta de coñecemento a nivel de isoforma debido á limitación das transcricións ensambladas de lectura curta.Aquí, introduciuse a plataforma de secuenciación de nanoporos para xerar transcricións completas, o que permitiu a inverstigación nas isoformas AS.
Deseño Experimental
Experimentos
Agrupación:1. CLL-SF3B1(WT) 2. CLL-SF3B1 (mutación K700E);3. Células B normais
Estratexia de secuenciación:Secuenciación de bibliotecas MinION 2D, secuenciación de bibliotecas PromethION 1D;lectura curta de datos das mesmas mostras
Plataforma de secuenciación:ONT Minion;ONT PromethION;
Análise Bioinformática
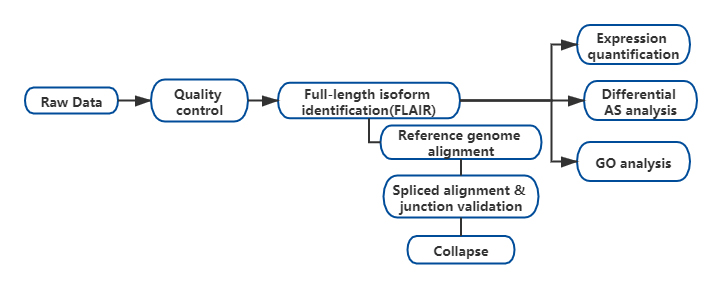
Resultados
AEn total, xeráronse 257 millóns de lecturas a partir de 6 mostras de CLL e 3 células B.De media, o 30,5% destas lecturas identificáronse como transcricións completas.
FDesenvolveuse a análise alternativa de isoformas completas de ARN (FLAIR) para xerar un conxunto de isoformas de alta confianza.FLAIR pódese resumir como:
Nanopore lee o aliñamento: identifica a estrutura xeral da transcrición baseándose no xenoma de referencia;
Scorrección de unión de plice: corrixe os erros de secuencia (vermello) co sitio de empalme de intróns anotados, intróns de datos de lectura curta ou ambos;
Collapse: resume isoformas representativas baseadas en cadeas de unión de empalme (conxunto de primeiro paso).Seleccione a iso de alta confianza en función do número de lecturas compatibles (Limiar: 3).
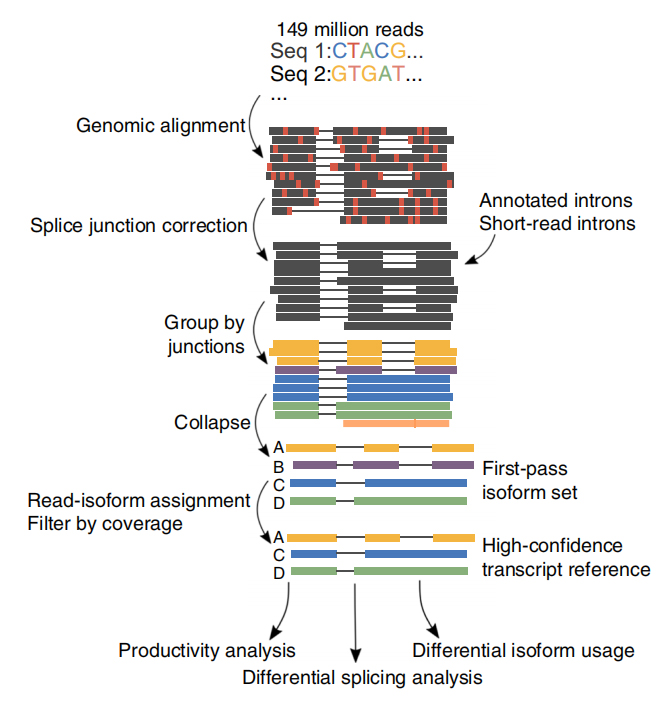
Figura 1. Análise FLAIR para identificar isoformas de lonxitude completa asociadas á mutación de SF3B1 en LLC
FLAIR identificou 326.699 isoformas empalmadas de alta confianza, o 90% das cales son isoformas novas.Descubriuse que a maioría destas isoformas non anotadas eran novas combinacións de unións de empalme coñecidas (142.971), mentres que o resto de isoformas novas contiñan intrón retido (21.700) ou exón novo (3594).
LAs secuencias de lectura prolongada permiten a identificación de sitios de empalme alterados por SF3B1-K700E mutantes a nivel de isoforma.35 alternativas 3'SS e 10 alternativas 5'SS atopáronse empalmadas de forma significativamente diferencial entre SF3B1-K700E e SF3B1-WT.33 das 35 alteracións foron recentemente descubertas por secuencias de lectura longa.Nos datos de Nanopore, a distribución da distancia entre os 3'SS alterados por SF3B1-K700E aos picos dos sitios canónicos é de aproximadamente -20 pb, o que difire significativamente dunha distribución de control, similar ao que se informou nas secuencias de lectura curta de CLL.Analizáronse as isoformas do xene ERGIC3, onde se atopou unha nova isoforma que contén o sitio de empalme proximal máis abundante en SF3B1-K700E.Tanto a SS 3 proximal como a distal asociáronse con patróns de SA diferenciados que xeraban múltiples isoformas.
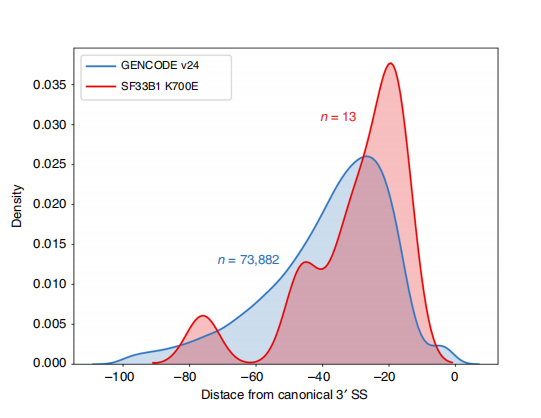
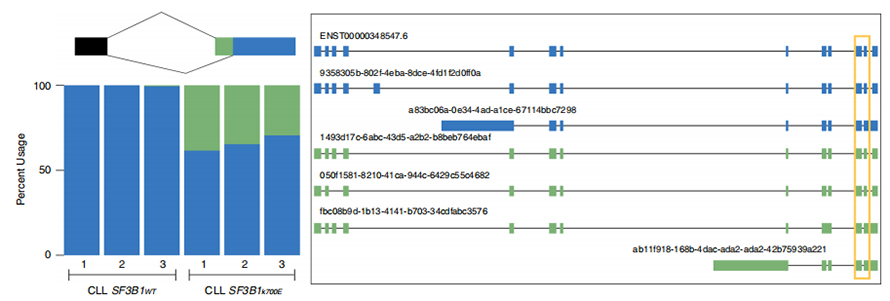
Figura 2. Patróns de empalme alternativos de 3′ identificados con datos de secuenciación de nanoporos
A análise do uso de eventos IR estivo limitada durante moito tempo na análise baseada en lecturas curtas debido á confianza na identificación e cuantificación de IR.A expresión das isoformas IR en SF3B1-K700E e SF3B1-WT cuantificáronse en base a secuencias de nanoporos, revelando unha regulación global á baixa das isoformas IR en SF3B1-K700E.
Figura 4. Intensidade da agricultura e conectividade de rede en tres sistemas agrícolas (A e B);Análise forestal aleatoria (C) e relación entre a intensidade agrícola e a colonización de AMF (D)
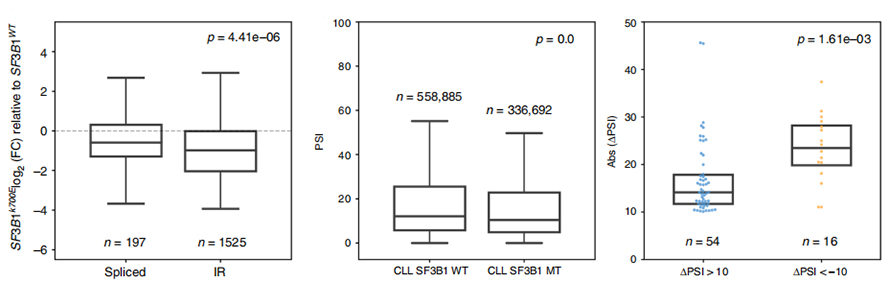
Figura 3. Os eventos de retención de intróns están máis fortemente regulados en CLL SF3B1-K700E
Tecnoloxía
Secuenciación de lectura longa de Nanopore
NA secuenciación de anopores é unha tecnoloxía de secuenciación de sinais eléctricos en tempo real dunha soa molécula.
DO ADN ou ARN de dobre cadea unirase á proteína nanoporosa incrustada na biopelícula e desenrolarase baixo o liderado da proteína motora.
DAs cadeas de NA/ARN atravesan a proteína da canle de nanoporos a un ritmo determinado baixo a acción da diferenza de voltaxe.
Mas moléculas xeran diferentes sinais eléctricos segundo a estrutura química.
Ra detección en tempo real de secuencias conséguese mediante a chamada base.
Realización da secuenciación de transcriptoma de lonxitude completa
√ Saturación de datos
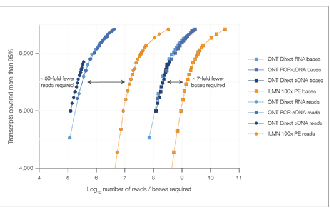
Se necesitan lecturas 7 veces menos para alcanzar unha saturación de datos comparable.
√ Identificación da estrutura da transcrición
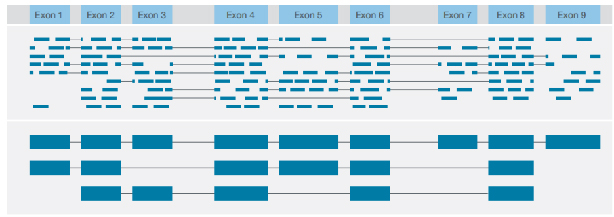
Identificación de diversas variantes estruturais con lectura consensuada completa de cada transcrición
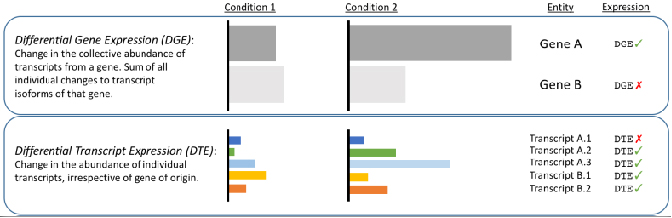
√ Análise diferencial a nivel de transcrición -Revela os cambios que ocultan as lecturas curtas
Referencia
Tang AD, Soulette CM, Baren MJV, et al.A caracterización de transcrición completa da mutación SF3B1 na leucemia linfocítica crónica revela a regulación negativa dos intróns retidos [J].Comunicacións da natureza.
Tecnoloxía e aspectos destacados ten como obxectivo compartir a aplicación exitosa máis recente de diferentes tecnoloxías de secuenciación de alto rendemento en varios ámbitos de investigación, así como ideas brillantes en deseño experimental e minería de datos.
Hora de publicación: Xaneiro-08-2022

