Secuenciación de ARNm de lonxitude completa-Nanopore
Vantaxes do servizo
● Baixo sesgo de secuencia
● Revelación de moléculas de ADNc de lonxitude total
● Menos datos necesarios para cubrir o mesmo número de transcricións
● Identificación de múltiples isoformas por xene
● Cuantificación de expresións a nivel de isoformas
Especificacións do servizo
| Biblioteca | Plataforma | Rendemento de datos recomendado (Gb) | Control de calidade |
| cDNA-PCR (enriquecido con poli-A) | Nanopore PromethION P48 | 6 Gb/mostra (dependendo da especie) | Relación de lonxitude total> 70% Puntuación media de calidade: Q10
|
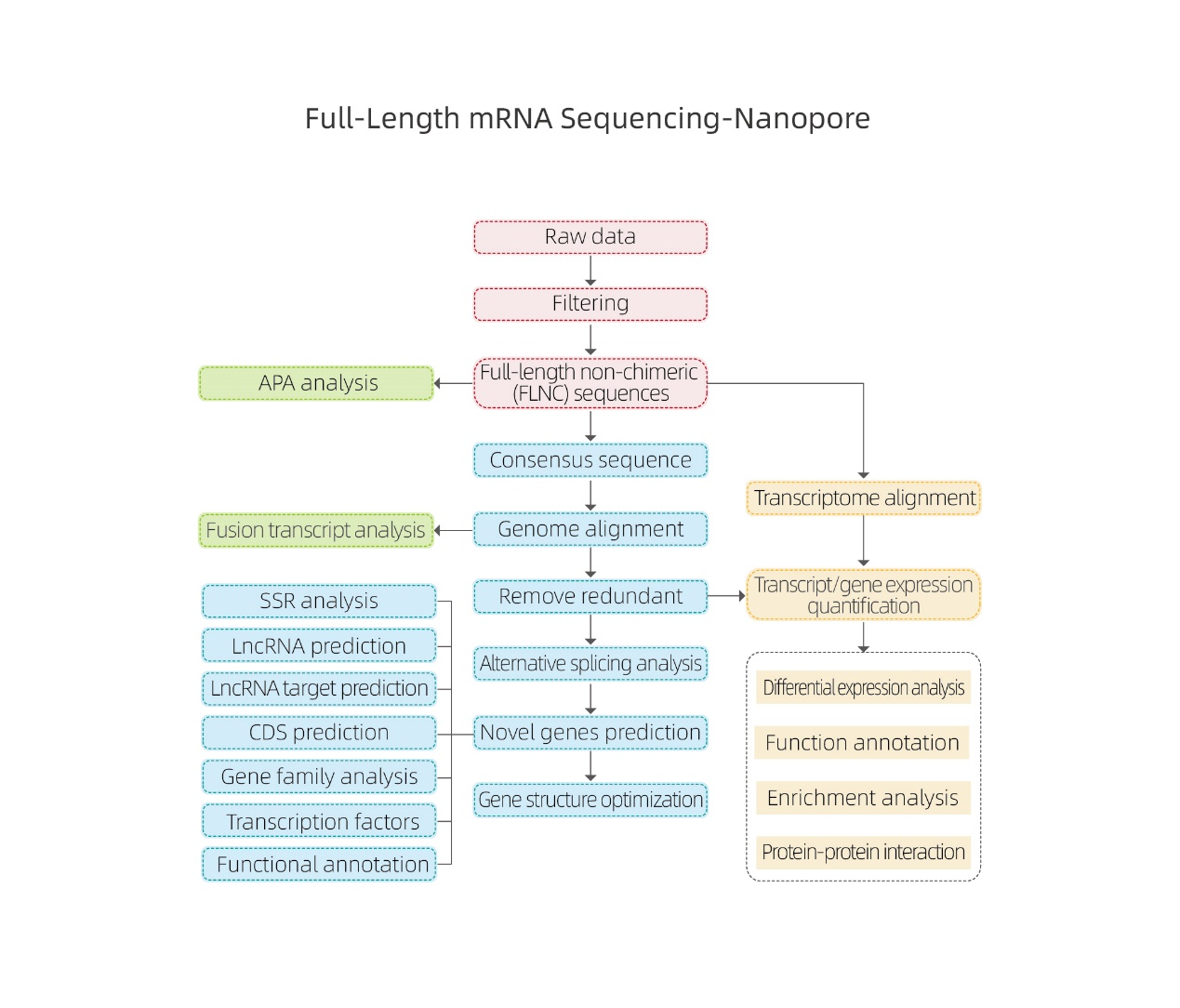
Análise bioinformática
●Procesamento de datos brutos
● Identificación da transcrición
● Empalme alternativo
● Cuantificación da expresión a nivel de xenes e nivel de isoformas
● Análise da expresión diferencial
● Anotación e enriquecemento de funcións (DEG e DET)
Requisitos de mostra e entrega
Requisitos de mostra:
Nucleótidos:
| Conc. (ng/μl) | Cantidade (μg) | Pureza | Integridade |
| ≥ 100 | ≥ 0,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Contaminación limitada ou nula de proteínas ou ADN no xel. | Para plantas: RIN≥7,0; Para animais: RIN≥7,5; 5,0≥28S/18S≥1,0; elevación de referencia limitada ou nula |
Tecido: Peso (seco): ≥1 g
*Para tecidos inferiores a 5 mg, recomendamos enviar mostras de tecido conxelada instantáneamente (en nitróxeno líquido).
Suspensión celular: reconto celular = 3×106- 1×107
*Recomendamos enviar lisado celular conxelado.No caso de que esa cela conte menos de 5×105, recoméndase conxelar flash en nitróxeno líquido, o que é preferible para a micro extracción.
Mostras de sangue: Volume ≥1 ml
Entrega de mostra recomendada
Recipiente: tubo de centrífuga de 2 ml (non se recomenda papel de aluminio)
Etiquetaxe da mostra: grupo+réplica, por exemplo, A1, A2, A3;B1, B2, B3....
Envío: 2、 Xeo seco: as mostras deben ser embaladas en bolsas e enterradas en xeo seco.
- Tubos RNAstable: as mostras de ARN pódense secar nun tubo de estabilización de ARN (por exemplo, RNAstable®) e enviarse a temperatura ambiente.
Fluxo de traballo do servizo
Nucleótidos:

Entrega da mostra

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
Fluxo de traballo do servizo
Tecido:

Deseño de experimentos

Entrega da mostra

extracción de ARN

Construción da biblioteca

Secuenciación

Análise de datos

Servizos posvenda
1.Análise de expresión diferencial -Gráfica do volcán
A análise da expresión diferencial pódese procesar tanto a nivel de xenes para identificar xenes expresados diferencialmente (DEG) como a nivel de isoformas para identificar de forma diferencial.
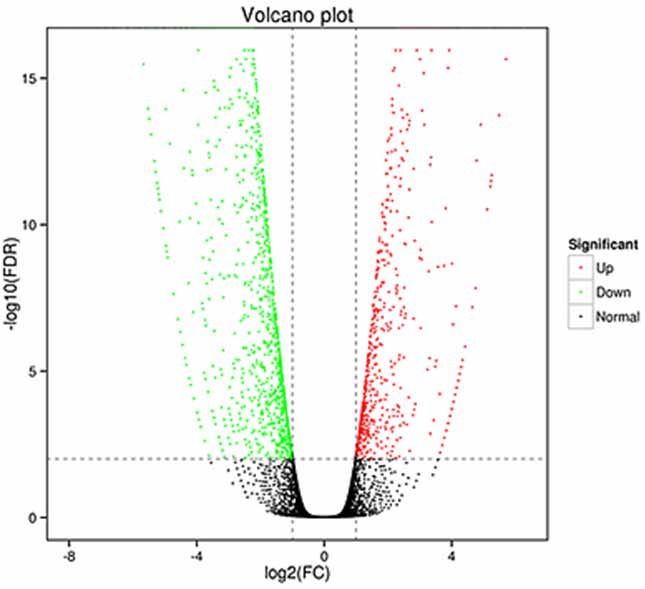
transcricións expresadas (DET)
2.Mapa de calor de agrupación xerárquica
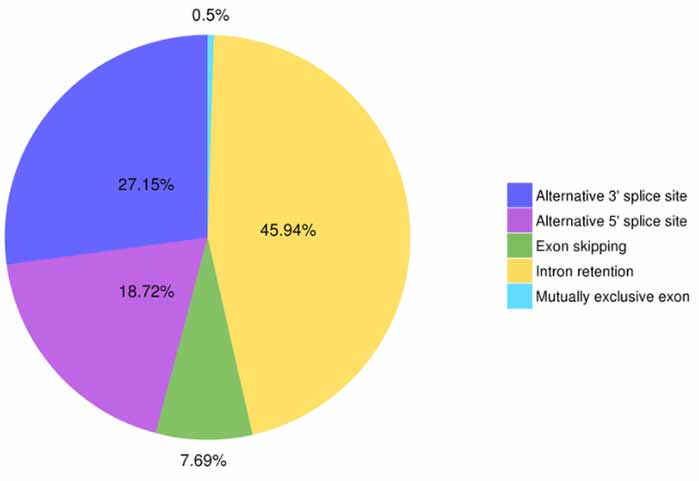
3.Identificación e clasificación de empalmes alternativos
Astalavista pode prever cinco tipos de eventos de empalme alternativos.
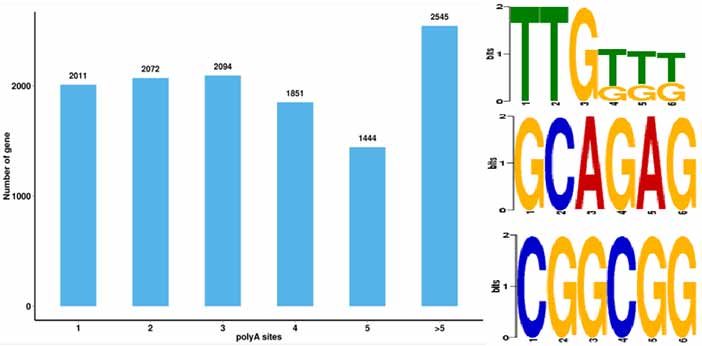
4.Identificación de eventos de poliadenilación alternativa (APA) e motivo a 50 bp aguas arriba de poli-A
Caso BMK
Identificación de empalme alternativo e cuantificación a nivel de isoforma mediante secuenciación de transcriptoma de lonxitude total de nanoporos
Publicado:Nature Communications, 2020
Estratexia de secuenciación:
Agrupación: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (mutación K700E);3. Células B normais
Estratexia de secuenciación: secuenciación da biblioteca MinION 2D, secuenciación da biblioteca PromethION 1D;lectura curta de datos das mesmas mostras
Plataforma de secuenciación: Nanopore MinION;Nanopore PromethION;
Resultados clave
1.Identificación de empalme alternativo a nivel de isoforma
As secuencias de lectura longa permiten a identificación do mutante SF3B1K700E- sitios de empalme alterados a nivel de isoforma.Atopáronse 35 alternativas 3′SS e 10 alternativas 5′SS empalmadas de forma significativa entre SF3B1K700Ee SF3B1WT.33 das 35 alteracións foron recentemente descubertas por secuencias de lectura longa.
2. Cuantificación de empalme alternativo a nivel de isoforma
Expresión das isoformas de retención de intróns (IR) en SF3B1K700Ee SF3B1WTcuantificáronse en base a secuencias de nanoporos, revelando unha regulación global á baixa das isoformas IR en SF3B1K700E.
Referencia
Tang AD, Soulette CM, Baren MJV, et al.A caracterización de transcrición completa da mutación SF3B1 na leucemia linfocítica crónica revela a regulación negativa dos intróns retidos [J].Comunicacións da natureza.