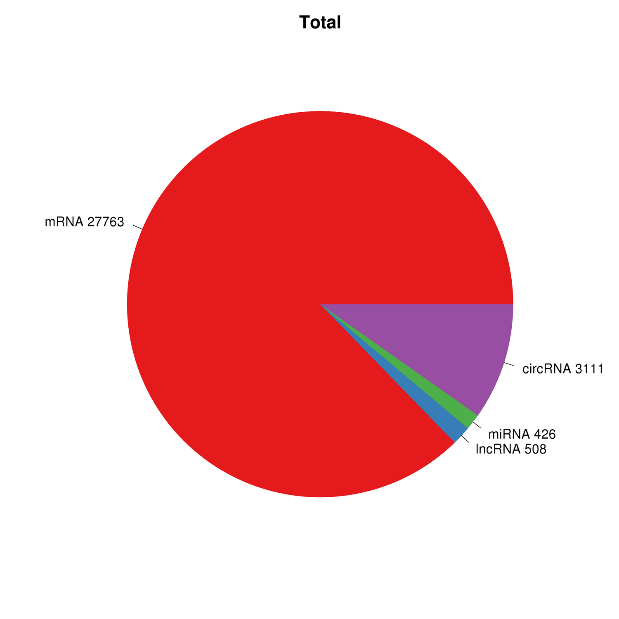
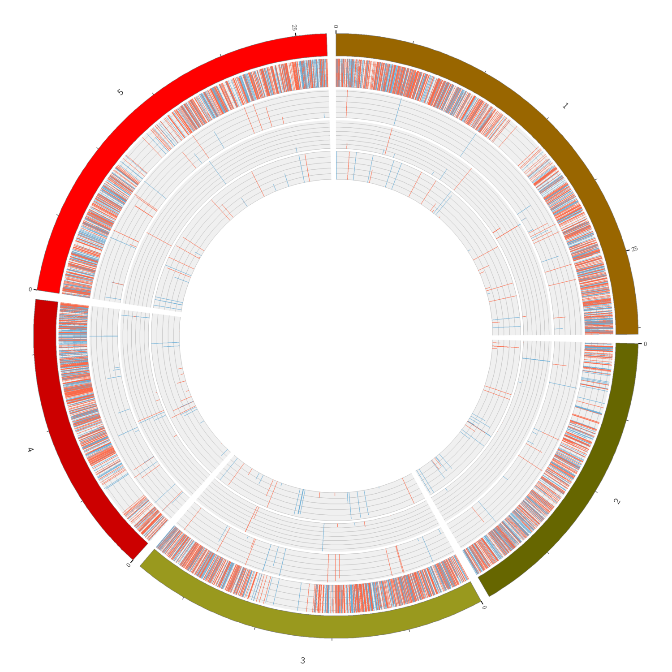
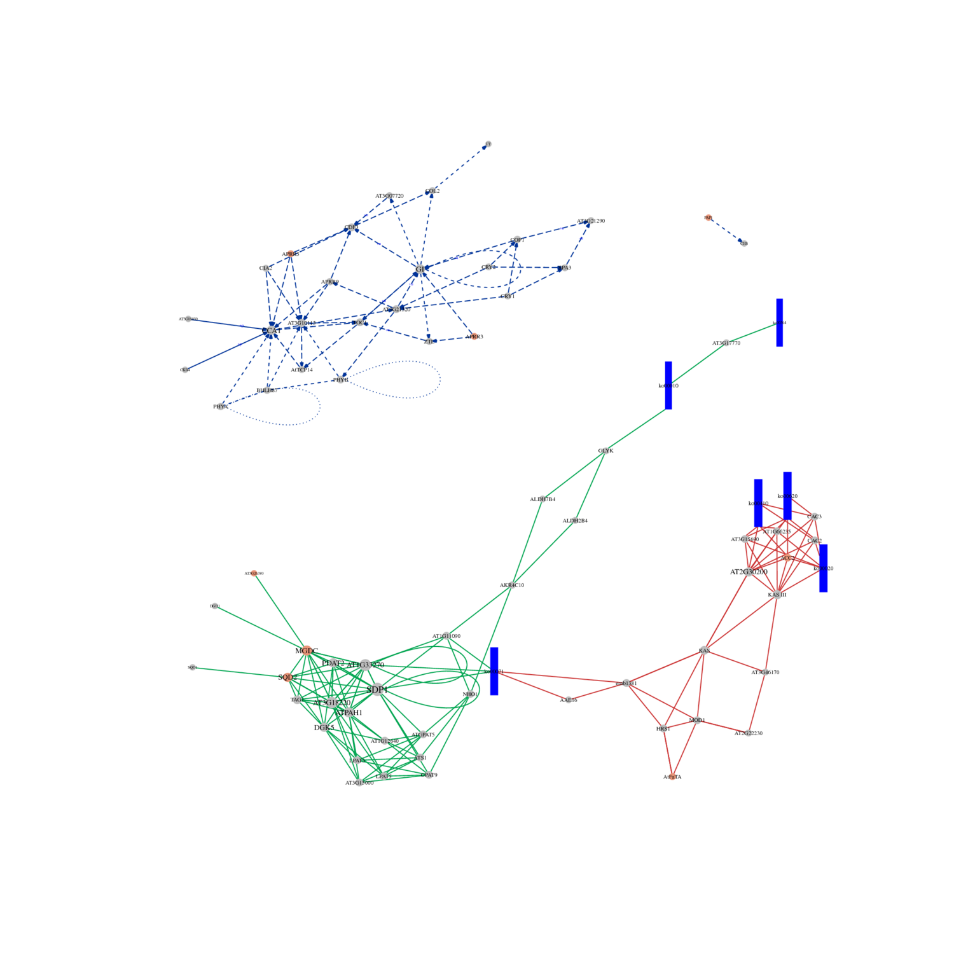
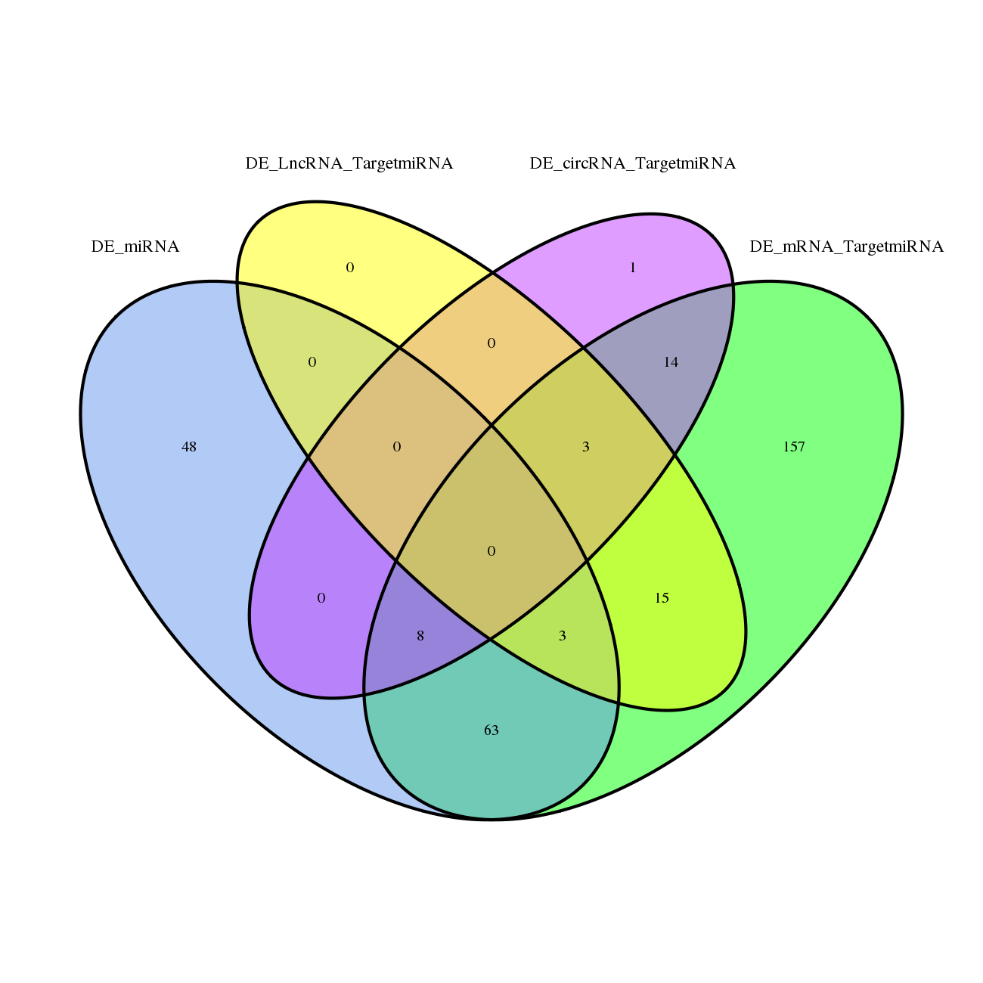
Folsleine transkriptome-sekwinsje - Illumina
Features
● Dûbele bibleteek om it folsleine transkriptoom te foltôgjen: rRNA-útputting folge troch PE150-biblioteektarieding en grutte seleksje folge troch SE50-biblioteektarieding
● Folsleine bioinformatika-analyze fan mRNA, lncRNA, circRNA en miRNA yn aparte bioinformatika-rapporten
● Joint analyze fan alle RNA ekspresje yn kombinearre rapport, ynklusyf ceRNA netwurken analyze.
Service Foardielen
●Yngeande analyze fan regeljouwing netwurken: ceRNA-netwurkanalyse wurdt ynskeakele troch de mienskiplike sequencing fan mRNA, lncRNA, circRNA en miRNA en troch in útputtende bioinformatyske workflow.
●Wiidweidige annotaasje: wy brûke meardere databases om funksjoneel te annotearjen fan 'e Differential Expressed Genes (DEG's) en de oerienkommende ferrikingsanalyse út te fieren, en jouwe ynsjoch oer de sellulêre en molekulêre prosessen dy't de transkriptoom-antwurd ûnderlizze.
●Wiidweidige Expertise: mei in spoarrekord fan it suksesfol sluten fan mear dan 2000 hiele transkriptoomprojekten yn ferskate ûndersyksdomeinen, bringt ús team in skat oan ûnderfining nei elk projekt.
●Rigorous Quality Control: wy ymplementearje kearnkontrôlepunten yn alle stadia, fan sample- en biblioteektarieding oant sequencing en bioinformatika.Dizze sekuere tafersjoch soarget foar de levering fan konsekwint resultaten fan hege kwaliteit.
● Wiidweidige Annotaasje: wy brûke meardere databases om funksjoneel te annotearjen fan 'e Differential Expressed Genes (DEG's) en de oerienkommende ferrikingsanalyse út te fieren, en jouwe ynsjoch oer de sellulêre en molekulêre prosessen dy't de transkriptoom-antwurd ûnderlizze.
●Post-Sales Support: Us ynset giet fierder as projektfoltôging mei in 3-moanne tsjinstperioade nei ferkeap.Yn dizze tiid biede wy projektopfolging, help by it oplossen fan problemen, en Q&A-sesjes om alle fragen oan te pakken yn ferbân mei de resultaten
Sample easken en levering
| Biblioteek | Sequencing strategy | Gegevens oanrikkemandearre | Kwaliteitsbeweitsing |
| rRNA fermindere | Illumina PE150 | 16 gb | Q30≥85% |
| Grutte selektearre | Illumina SE50 | 10-20M lêzen |
Sample easken:
Nukleotiden:
| Konk. (ng/μl) | Bedrach (μg) | Purity | Yntegriteit |
| ≥ 100 | ≥ 1 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Beheinde as gjin proteïne- of DNA-fersmoarging werjûn op gel. | Planten: RIN≥6.5 Dier: RIN≥7.0 5.0≥28S/18S≥1.0; beheind of gjin baseline hichte |
Oanrikkemandearre Sample Delivery
Kontener:
2 ml sintrifugebuis (Tin folie wurdt net oanrikkemandearre)
Sample labeling: Groep + replikearje bgl. A1, A2, A3;B1, B2, B3......
Ferstjoering:
1.Dry-ice: Samples moatte wurde ynpakt yn sekken en begroeven yn droech-iis.
2.RNAstable buizen: RNA-monsters kinne wurde droege yn RNA-stabilisaasjebuis (bgl. RNAstable®) en ferstjoerd yn keamertemperatuer.
Service Work Flow

Eksperimint ûntwerp

Sample levering

RNA-ekstraksje

Biblioteekbou

Sequencing

Data analyze

Nei-ferkeap tsjinsten
Bioinformatika
RNA ekspresje oersjoch
Differinsjaal útdrukt genen
ceRNA analyze
Ferkenne de foarútgong fan ûndersyk fasilitearre troch BMKGene 'hele tsjinsten foar transkriptoomsekwinsjes fia in gearstalde samling publikaasjes.
Dai, Y. et al.(2022) 'Utwreide ekspresjeprofilen fan mRNA's, lncRNA's en miRNA's yn Kashin-Beck-sykte identifisearre troch RNA-sequencing', Molecular Omics, 18(2), pp. 154-166.doi: 10.1039/D1MO00370D.
Liu, N. Nan et al.(2022) 'Folle lingte transkriptomen analyze fan kjeldresistinsje fan Apis cerana yn Changbai Mountain yn' e oerwinteringperioade.', Gene, 830, s. 146503–146503.doi: 10.1016/J.GENE.2022.146503.
Wang, XJ et al.(2022) 'Multi-Omics-yntegraasje-basearre prioritearring fan konkurrearjende endogene RNA-regelingsnetwurken yn lytse sellen longkanker: molekulêre skaaimerken en drugskandidaten', Frontiers in Oncology, 12, p.904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. et al.(2022) 'Yntegrearre analyze fan' e lncRNA / circRNA-miRNA-mRNA-ekspresjeprofilen ûntbleatet nije ynsjoch yn potinsjele meganismen yn antwurd op root-knotnematoden yn pinda', BMC Genomics, 23(1), pp. 1-12.doi: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. et al.(2022) 'Hele-transkriptome RNA-sekwinsje markeart de molekulêre meganismen dy't ferbûn binne mei it ûnderhâld fan postharvest kwaliteit yn brokkoli troch reade LED-bestraling', Postharvest Biology and Technology, 188, p.111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.