Séquençage du transcriptome entier – Illumina
Caractéristiques
● Double bibliothèque pour séquencer le transcriptome complet : déplétion de l'ARNr suivie de la préparation de la bibliothèque PE150 et sélection de la taille suivie de la préparation de la bibliothèque SE50
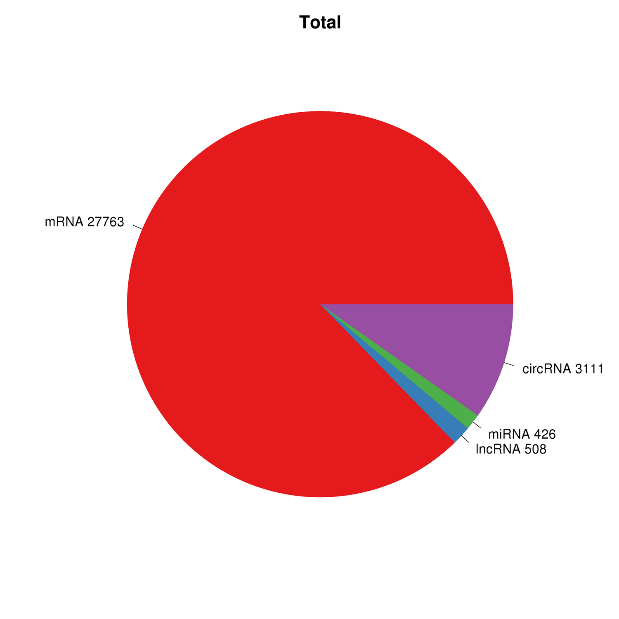
● Analyse bioinformatique complète de l'ARNm, de l'ARNnc, de l'ARNcirc et du miARN dans des rapports bioinformatiques distincts
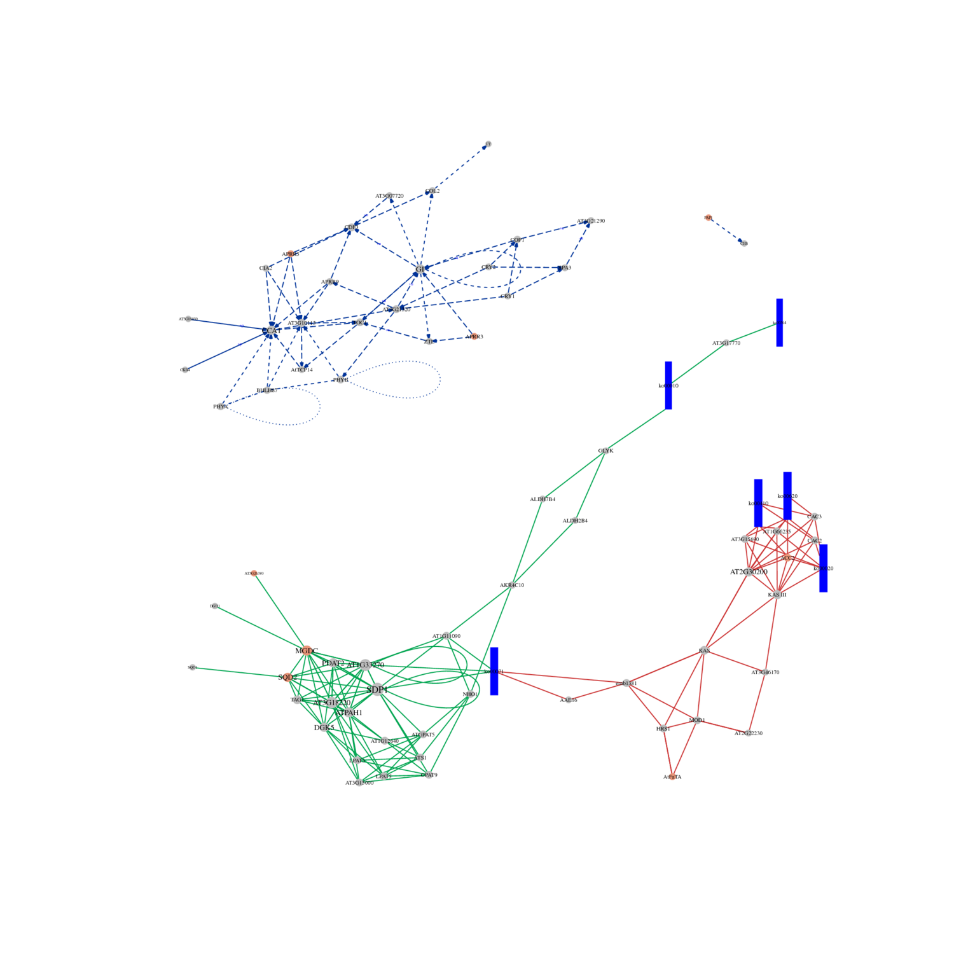
● Analyse conjointe de toutes les expressions d'ARN dans un rapport combiné, y compris l'analyse des réseaux ceRNA.
Avantages des services
●Analyse approfondie des réseaux de régulation: L'analyse du réseau ceRNA est rendue possible par le séquençage conjoint de l'ARNm, de l'ARNnc, du circARN et du miARN et par un flux de travail bioinformatique exhaustif.
●Annotation complète: nous utilisons plusieurs bases de données pour annoter fonctionnellement les gènes exprimés différentiellement (DEG) et effectuer l'analyse d'enrichissement correspondante, fournissant ainsi un aperçu des processus cellulaires et moléculaires sous-jacents à la réponse du transcriptome.
●Une expertise étendue: avec une expérience dans la clôture réussie de plus de 2000 projets complets de transcriptome dans divers domaines de recherche, notre équipe apporte une richesse d'expérience à chaque projet.
●Contrôle qualité rigoureux: nous mettons en œuvre des points de contrôle de base à toutes les étapes, de la préparation des échantillons et des bibliothèques au séquençage et à la bioinformatique.Ce suivi méticuleux garantit la fourniture de résultats constants de haute qualité.
● Annotation complète: nous utilisons plusieurs bases de données pour annoter fonctionnellement les gènes exprimés différentiellement (DEG) et effectuer l'analyse d'enrichissement correspondante, fournissant ainsi un aperçu des processus cellulaires et moléculaires sous-jacents à la réponse du transcriptome.
●Assistance après-vente: Notre engagement s'étend au-delà de la réalisation du projet avec une période de service après-vente de 3 mois.Pendant cette période, nous proposons un suivi de projet, une assistance au dépannage et des séances de questions-réponses pour répondre à toute question liée aux résultats.
Exigences et livraison des échantillons
| Bibliothèque | Stratégie de séquençage | Données recommandées | Contrôle de qualité |
| ARNr épuisé | Illumina PE150 | 16 GB | Q30≥85 % |
| Taille sélectionnée | Illumina SE50 | 10 à 20 millions de lectures |
Exigences de l'échantillon :
Nucléotides :
| Conc. (ng/μl) | Quantité (μg) | Pureté | Intégrité |
| ≥ 100 | ≥ 1 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contamination limitée ou inexistante en protéines ou en ADN indiquée sur le gel. | Plantes : RIN≥6,5 Animal : RIN≥7,0 5,0≥28S/18S≥1,0 ; élévation de la ligne de base limitée ou inexistante |
Livraison d’échantillon recommandée
Récipient:
Tube à centrifuger de 2 ml (le papier d’aluminium n’est pas recommandé)
Étiquetage des échantillons : Groupe + répétition, par exemple A1, A2, A3 ;B1, B2, B3... ...
Expédition:
1. Glace sèche : Les échantillons doivent être emballés dans des sacs et enterrés dans de la glace carbonique.
2.Tubes RNAstable : les échantillons d'ARN peuvent être séchés dans un tube de stabilisation d'ARN (par exemple RNAstable®) et expédiés à température ambiante.
Flux de travail des services

Conception d'expériences

Livraison d'échantillon

Extraction d'ARN

Construction d'une bibliothèque

Séquençage

L'analyse des données

Services après-vente
Bioinformatique
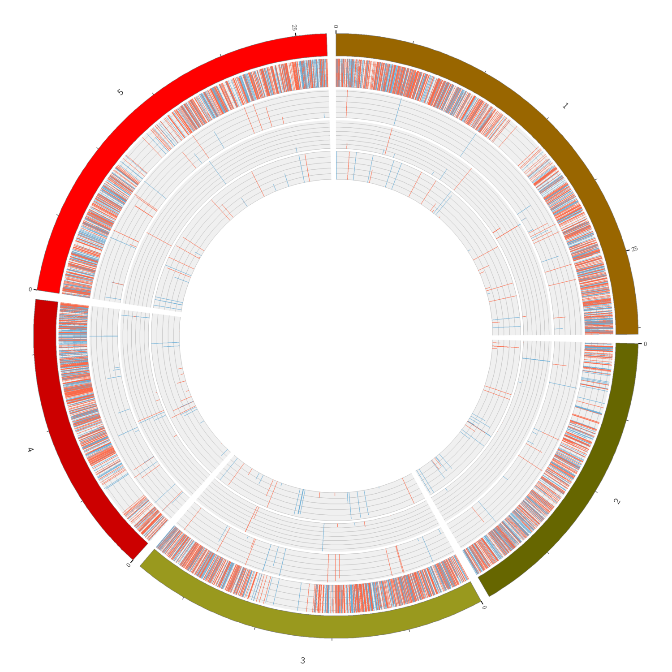
Aperçu de l'expression de l'ARN
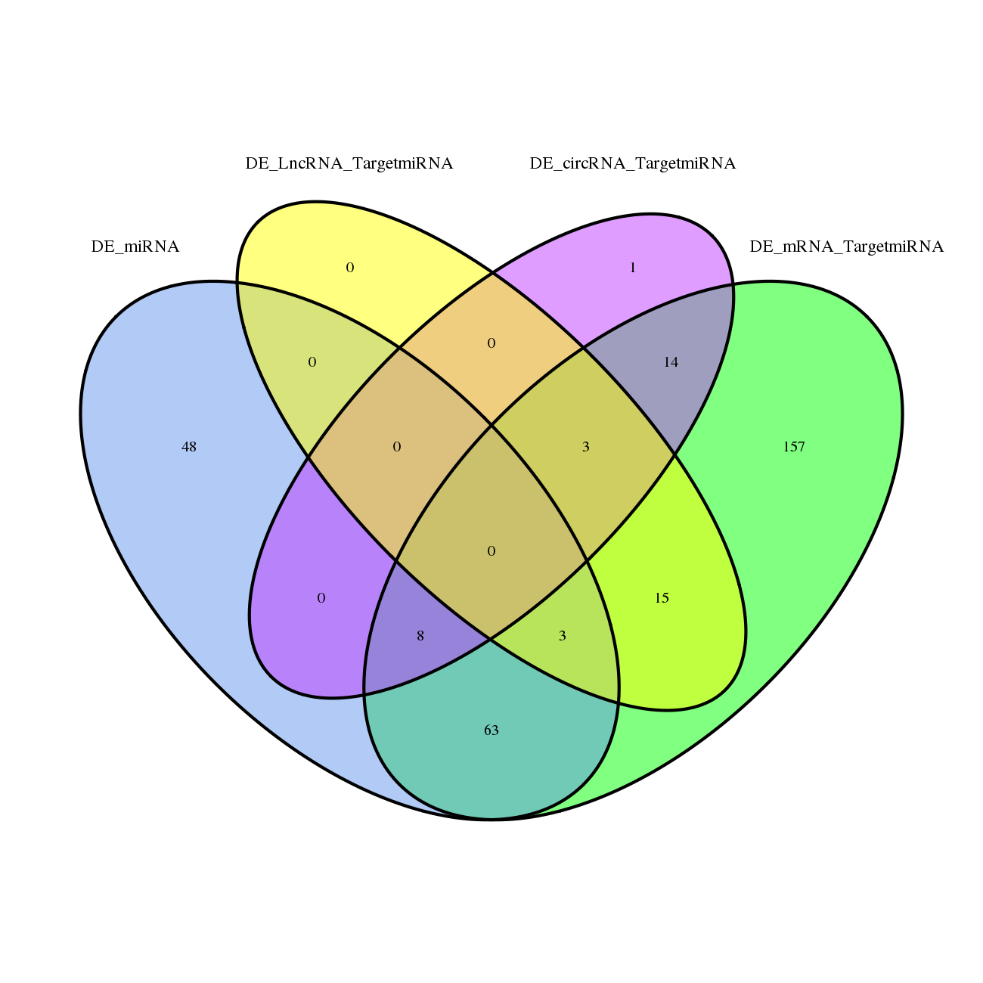
Gènes exprimés différentiellement
analyse d'ARNc
Explorez les progrès de la recherche facilités par les services de séquençage complet du transcriptome de BMKGene à travers une collection organisée de publications.
Dai, Y. et al.(2022) « Profils d'expression complets des ARNm, des ARNnc et des miARN dans la maladie de Kashin-Beck identifiés par séquençage d'ARN », Molecular Omics, 18(2), pp.est ce que je: 10.1039/D1MO00370D.
Liu, N. Nan et al.(2022) « Analyse des transcriptomes complets de la résistance au froid d'Apis cerana dans la montagne Changbai pendant la période d'hivernage. », Gene, 830, pp.est ce que je: 10.1016/J.GENE.2022.146503.
Wang, XJ et coll.(2022) « Priorisation basée sur l'intégration multi-omique des réseaux de régulation d'ARN endogènes concurrents dans le cancer du poumon à petites cellules : caractéristiques moléculaires et candidats médicaments », Frontiers in Oncology, 12, p.904865. est ce que je : 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. et al.(2022) « L'analyse intégrée des profils d'expression lncRNA/circRNA-miRNA-mRNA révèle de nouvelles informations sur les mécanismes potentiels en réponse aux nématodes à galles chez l'arachide », BMC Genomics, 23(1), pp.est ce que je: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. et coll.(2022) « Le séquençage de l'ARN du transcriptome entier met en évidence les mécanismes moléculaires associés au maintien de la qualité post-récolte du brocoli par irradiation par LED rouge », Postharvest Biology and Technology, 188, p.111878. est ce que je : 10.1016/J.POSTHARVBIO.2022.111878.