
Séquençage du génome végétal/animal de novo
Avantages des services

Développement de plateformes de séquençage et de bioinformatique ende novoassemblage du génome
(Amarasinghe SL et al.,Biologie du génome, 2020)
● Construire de nouveaux génomes et améliorer les génomes de référence existants pour les espèces d'intérêt.
● Plus grande précision, continuité et exhaustivité dans l'assemblage
● Construire des ressources fondamentales pour la recherche sur le polymorphisme de séquence, les QTL, l'édition de gènes, la sélection, etc.
● Équipé d'une gamme complète de plateformes de séquençage de troisième génération : solution unique d'assemblage du génome
● Stratégies flexibles de séquençage et d'assemblage répondant à divers génomes avec différentes caractéristiques
● Équipe de bioinformaticiens hautement qualifiés avec une grande expérience dans les assemblages de génomes complexes, notamment les polyploïdes, les génomes géants, etc.
● Plus de 100 cas réussis avec un facteur d'impact cumulé publié de plus de 900
● Délai d'exécution pouvant atteindre 3 mois pour l'assemblage du génome au niveau des chromosomes.
● Un support technique solide avec une série de brevets et de droits d'auteur sur les logiciels tant du côté expérimental que de la bioinformatique.
Spécifications des services
|
Contenu
|
Plate-forme
|
Lire la longueur
|
Couverture
|
| Enquête génomique
| Illumina NovaSeq
| PE150
| ≥ 50X
|
| Séquençage du génome
| PacBio Revio
| Lectures HiFi 15 Ko
| ≥ 30X
|
| Salut-C
| Illumina NovaSeq
| PE150
| ≥100X
|
Flux de travail

Exigences et livraison des échantillons
Exigences de l'échantillon :
| Espèces | Tissu | Pour PacBio | Pour Nanopore |
| Animaux | Organes viscéraux (foie, rate, etc.) | ≥ 1,0 g | ≥ 3,5g |
| Muscle | ≥ 1,5 g | ≥ 5,0 g | |
| Sang de mammifères | ≥ 1,5 ml | ≥ 5,0 ml | |
| Sang de poisson ou d'oiseau | ≥ 0,2 ml | ≥ 0,5 ml | |
| Plantes | Feuilles fraîches | ≥ 1,5 g | ≥ 5,0 g |
| Pétale ou tige | ≥ 3,5g | ≥ 10,0 g | |
| Racines ou graines | ≥ 7,0 g | ≥ 20,0 g | |
| Cellules | Culture de cellules | ≥ 3×107 | ≥ 1×108 |
Livraison d’échantillon recommandée
Récipient : tube à centrifuger de 2 ml (le papier d'aluminium n'est pas recommandé)
Pour la plupart des échantillons, nous recommandons de ne pas conserver dans l'éthanol.
Étiquetage des échantillons : les échantillons doivent être clairement étiquetés et identiques au formulaire d’informations sur les échantillons soumis.
Expédition : Glace carbonique : Les échantillons doivent d’abord être emballés dans des sacs et enterrés dans de la glace carbonique.
Flux de travail des services

Conception d'expériences

Livraison d'échantillon

Extraction d'ADN

Construction d'une bibliothèque

Séquençage

L'analyse des données

Services après-vente
*Les résultats de démonstration présentés ici proviennent tous de génomes publiés avec Biomarker Technologies
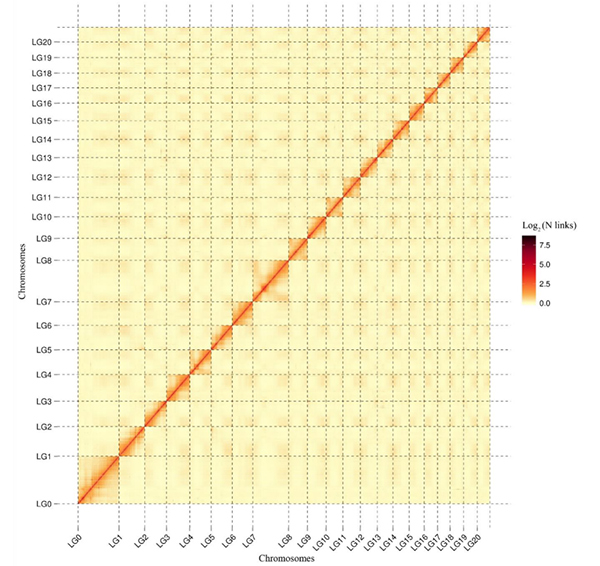
1.Circos sur l'assemblage du génome au niveau des chromosomes deG. rotundifoliumpar la plateforme de séquençage Nanopore
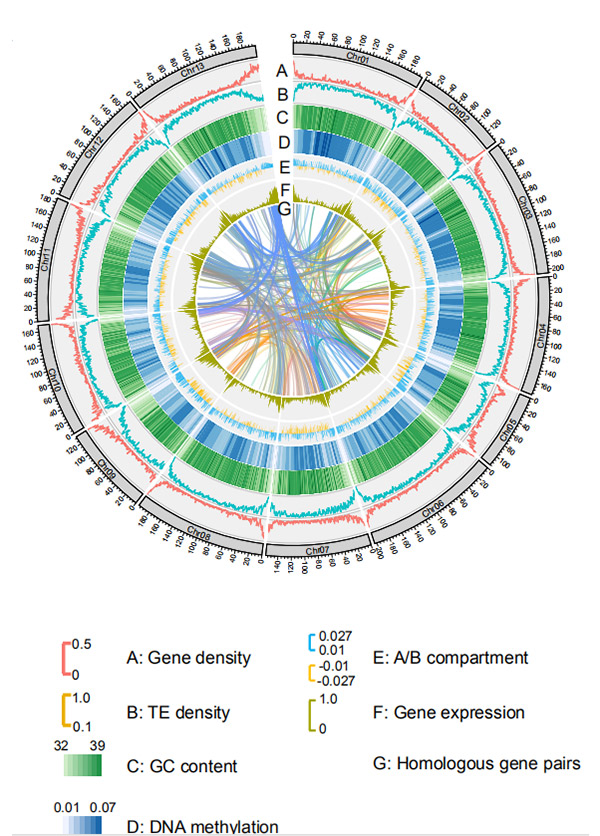
Wang M et coll.,Biologie moléculaire et évolution, 2021
2.Statistiques de l'assemblage et de l'annotation du génome du seigle Weining
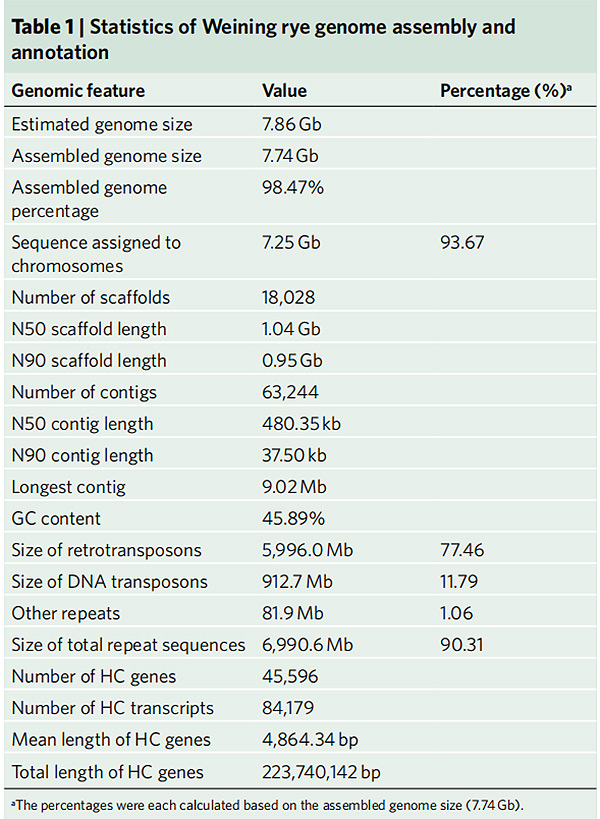
Li G et coll.,Génétique naturelle, 2021
3. Prédiction génétique deSéchium edulegénome, dérivé de trois méthodes de prédiction :De novoprédiction, prédiction basée sur l'homologie et prédiction basée sur les données RNA-Seq
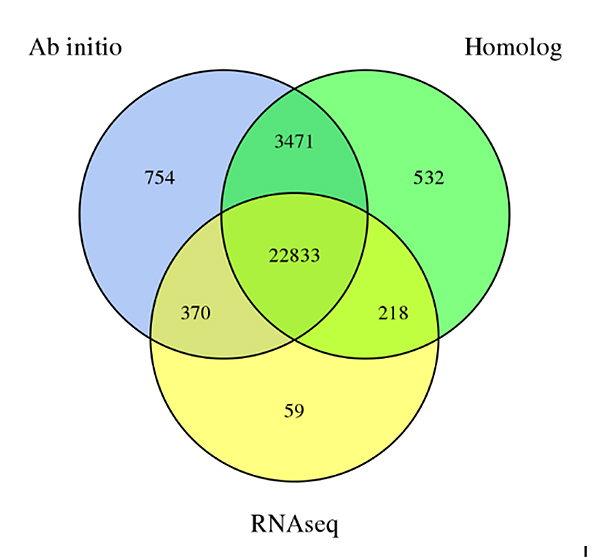
Fu A et coll.,Recherche horticole, 2021
4.Identification de longues répétitions terminales intactes dans trois génomes de coton
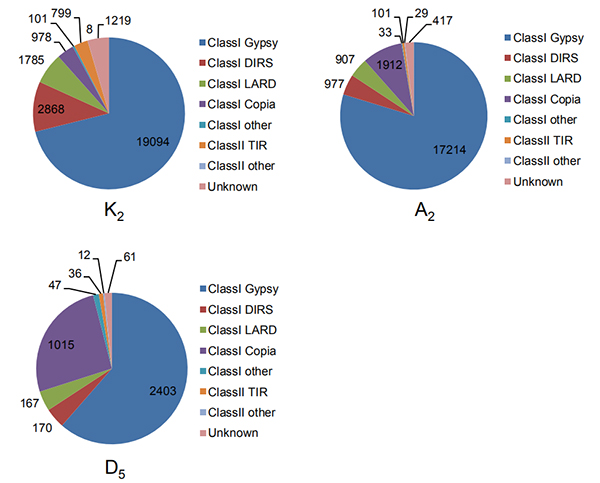
Wang M et coll.,Biologie moléculaire et évolution, 2021
5. Carte thermique Hi-C duC. acuminatagénome montrant des interactions globales à l'échelle du génome.L'intensité des interactions Hi-C est proportionnelle à la distance linéaire entre les contigs.Une ligne droite nette sur cette carte thermique indique un ancrage très précis des contigs sur les chromosomes.(Taux d'ancrage Contig : 96,03%)
Kang M et al.,Communications naturelles,2021
Affaire BMK
Un assemblage génomique de haute qualité met en évidence les caractéristiques génomiques du seigle et les gènes agronomiquement importants
Publié : Génétique naturelle, 2021
Stratégie de séquençage :
Assemblage du génome : mode PacBio CLR avec bibliothèque de 20 Ko (497 Go, environ 63×)
Correction de séquence : NGS avec bibliothèque d'ADN de 270 pb (430 Go, environ 54×) sur plateforme Illumina
Ancrage Contigs : bibliothèque Hi-C (560 Go, environ 71×) sur plateforme Illumina
Carte optique : (779,55 Go, environ 99×) sur Bionano Irys
Résultats clés
1.Un assemblage du génome du seigle Weining a été publié avec une taille totale du génome de 7,74 Go (98,74 % de la taille estimée du génome par cytométrie en flux).L'échafaudage N50 de cet assemblage atteint 1,04 Go.93,67% des contigs ont été ancrés avec succès sur 7 pseudo-chromosomes.Cet assemblage a été évalué par Linkage Map, LAI et BUSCO, ce qui a donné lieu à des scores élevés dans toutes les évaluations.
2. D'autres études sur la génomique comparative, la carte des liaisons génétiques et les études transcriptomiques ont été réalisées sur la base de ce génome.Une série de caractéristiques génomiques liées à des traits ont été révélées, notamment des duplications de gènes à l'échelle du génome et leur impact sur les gènes de biosynthèse de l'amidon ;organisation physique de loci complexes de prolamine, caractéristiques d'expression génique sous-jacentes au trait d'épiaison précoce et régions chromosomiques et locus putatifs associés à la domestication dans le seigle.
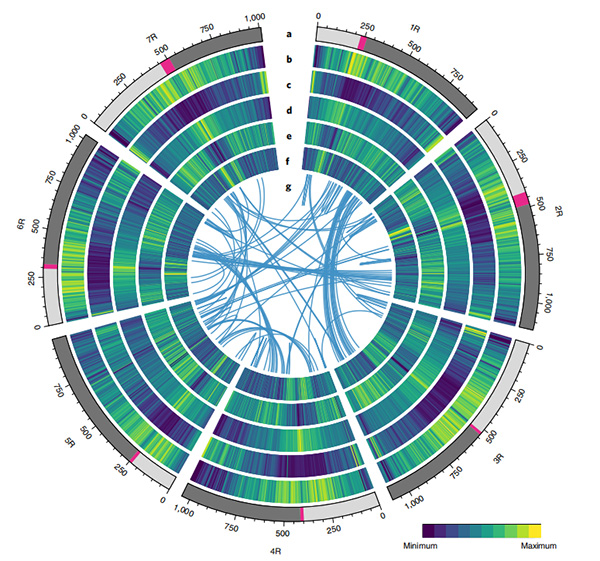 Diagramme Circos sur les caractéristiques génomiques du génome du seigle Weining | 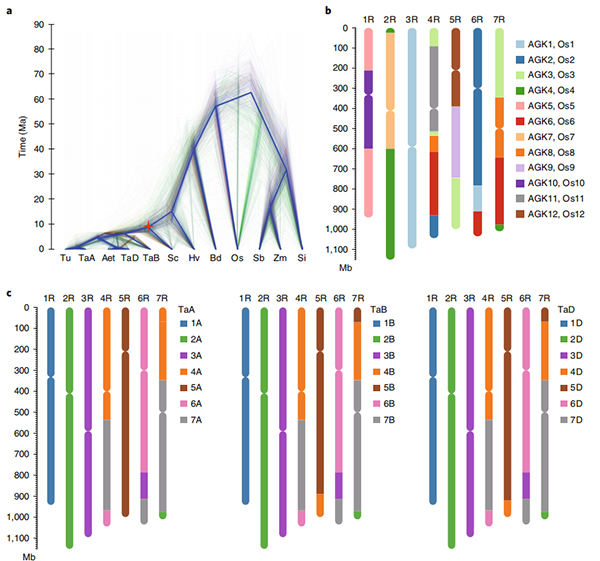 Analyses évolutives et de synthèse chromosomique du génome du seigle |
Li, G., Wang, L., Yang, J.et coll.Un assemblage génomique de haute qualité met en évidence les caractéristiques génomiques du seigle et les gènes agronomiquement importants.Nat Genet 53,574-584 (2021).
https://doi.org/10.1038/s41588-021-00808-z










