-

Des analyses comparatives du génome mettent en évidence l'expansion du génome médiée par les transposons et l'architecture évolutive du repliement génomique 3D du coton
ÉVOLUTION DU GÉNOME Les analyses comparatives du génome mettent en évidence l'expansion du génome médiée par les transposons et l'architecture évolutive du repliement génomique 3D dans le coton Séquençage des nanopores |Salut-C |PacBio...En savoir plus -
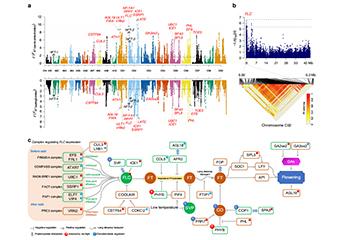
Des gènes associés à la tolérance au stress, à la teneur en huile, à la qualité des graines et à l'amélioration de l'écotype ont été identifiés par GWAS dans le colza
Titre GWAS : Le reséquençage du génome entier révèle l'origine de Brassica napus et les loci génétiques impliqués dans son amélioration Journal : Nature Communications NGS |WGS |Reséquençage |GWAS |Transcriptome |ARNseq |Brassica napus |Évolution |Domestique...En savoir plus -
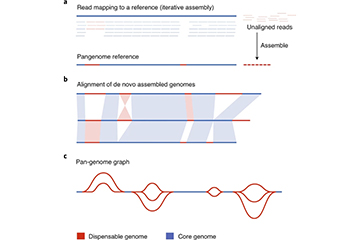
Les études pan-génomiques fournissent des vues génétiques approfondies et complètes d’une espèce
ÉVOLUTION DU GÉNOME, PANGENOME Qu'est-ce que le Pan-génome ?Les preuves accumulées montrent que la variance entre les différentes souches d’une espèce peut être énorme.Un seul génome est loin d’être suffisant pour obtenir une vue d’ensemble de l’information génétique d’une seule espèce.T...En savoir plus




