MICROBIEN

La compatibilité bactérienne et l'immobilisation avec le biocharbon ont amélioré la dégradation du tébuconazole, la composition et le fonctionnement du microbiome du sol
Séquençage d'amplicons 16S complet |PacBio HiFi |Diversité alpha |Diversité bêta
Dans cette étude, le séquençage complet de l’amplicon 16S par PacBio et l’analyse bioinformatique ont été fournis par Biomarker Technologies.
Points forts
La bactérie dégradant le tébuconazole immobilisée par Biochar, Alcaligenes faecalis WZ-2, a été étudiée sur l'efficacité de la biodégradation et ses effets sur le sol contaminé au tébuconazole par rapport à la souche libre dégradante WZ-2.
1. Le WZ-2 immobilisé au biochar a montré une dégradation plus efficace du tébuconazole par rapport au WZ-2 libre en réduisant la demi-vie du tébuconazole dans le sol de 18,7 jours à 13,3 jours.
2. Les WZ-2 immobilisés par du biochar ont pu restaurer les activités enzymatiques microbiennes natives du sol, notamment l'uréase, la déshydrogénase et l'invertase, etc.
3. Le profil microbien dans le sol traité au WZ-2 immobilisé par du biochar, déterminé par séquençage 16S complet, soutient fortement que ce système pourrait restaurer la santé du sol en améliorant la structure de la communauté bactérienne sous contamination par le tébuconazole.
Expérience (liée au séquençage)
Regroupement : CK : Sol naturel ;T : Sol enrichi en tébuconazole ;S : Le tébuconazole contenait du sol avec la souche libre WZ-2 ;BC : Le tébuconazole contenait de la terre avec du biocharbon ;BCS : Le tébuconazole contenait du sol avec du biocharbon WZ-2 immobilisé.
Échantillonnage : extraction de l'ADN total du sol amplifiée par des amorces d'ADNr 16S
27F (5′-AGAGTTTGATCCTGGCTCAG-3 ') et 1492R (5′-GGTTACCTTGTTACGA),représentant l'ADNr 16S complet
Plateforme de séquençage : PacBio RS II
Stratégie de séquençage : lectures CCS HIFI
L'analyse des données:BMKCloudLes plateformes bioinformatiques font du poisson rouge un excellent système de modèle génétique pour la physiologie et l'évolution des poissons.
Résultat
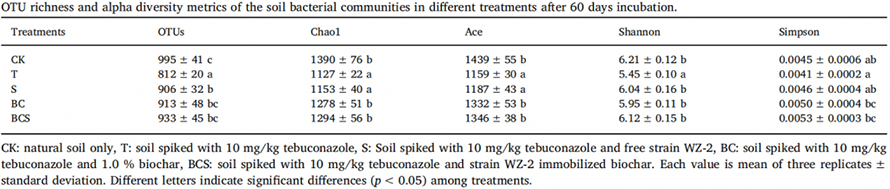
La structure de la communauté microbienne du sol a été déterminée par séquençage de l’ADNr 16S.La richesse OTU et l'indice de diversité alpha, y compris les indices Chao1, Ace, Shannon et Simpson, ont été évalués pour révéler la diversité des espèces dans chaque système.Après 60 jours d'incubation, tous les indices ont montré une tendance similaire, c'est-à-dire que le tébuconazole pourrait induire une réduction de la richesse et de la diversité des espèces dans le sol.Cependant, en ajoutant la souche WZ-2, la communauté bactérienne du sol a été partiellement récupérée en termes de richesse et de diversité.Une différence limitée a été observée entre BC, BCS et CK, indiquant que le biochar et le WZ-2 immobilisé par du biochar pourraient efficacement restaurer la santé biologique du sol.
La méthode de groupe de paires non pondérée avec moyennes arithmétiques (UPGMA) a été appliquée dans cette étude pour montrer la diversité bêta entre les groupes.Comme le montre la figure suivante, BC, BSC et CK partageaient un modèle de composition microbienne plus similaire à celui des groupes T et S, ce qui indiquait en outre que l'introduction de biocharbon dans la biorestauration des sols contaminés par le tébuconazole pourrait largement faciliter la récupération de la communauté microbienne dans le sol.
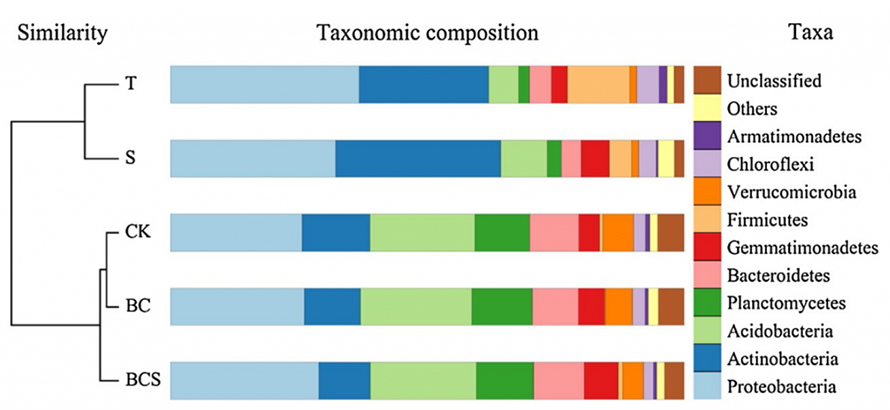
Chiffre.Regroupement UPGMA de la communauté bactérienne au niveau du phylum sous différents traitements
Référence
Sun, Tong et coll."La compatibilité bactérienne et l'immobilisation avec le biocharbon ont amélioré la dégradation du tébuconazole, la composition et le fonctionnement du microbiome du sol."Journal des Matériaux Dangereux398 (2020) : 122941.
Actualités et faits saillants vise à partager les derniers cas réussis avec Biomarker Technologies, en capturant de nouvelles réalisations scientifiques ainsi que des techniques importantes appliquées au cours de l'étude.
Heure de publication : 08 janvier 2022

