GWAS
Titre : Le reséquençage du génome entier révèle l’origine de Brassica napus et les locus génétiques impliqués dans son amélioration
Journal: Communications naturelles
NGS |WGS |Reséquençage |GWAS |Transcriptome |ARNseq |Brassica napus |Évolution |Domestication
Dans cette étude, Biomarker Technologies a fourni des services sur le séquençage NGS, ainsi qu'un support technique sur l'analyse bioinformatique des données de séquençage.
Arrière-plan
Brassica napus(colza) est une culture oléagineuse importante et un excellent modèle pour étudier les processus de spéciation, d'évolution et de sélection polyploïdes.Cependant, on ne sait toujours pas si les espèces sauvages ou les donneurs domestiqués étaient des géniteurs parentaux et les gènes qui ont contribué à la domestication et à l'amélioration du colza.
Matériels et méthodes
Matériaux:588B.napusles accessions ont été impliquées dans cette étude, dont 466 d’Asie, 102 d’Europe, 13 d’Amérique du Nord et 7 d’Australie.Sur la base des enregistrements d'habitudes de croissance, ces matériaux ont été divisés en trois écotypes :printemps (86 accessions), hiver (74 accessions) et semi-hivernal (428 accessions).
Séquençage :En moyenne env.5× (allant de 3,37× à 7,71×)
Plateforme de séquençage :Illumina Hiseq 4000
Production de données :4,03 To de données propres
Appel SNP :BWA + GATK.5 294 158 SNP et 1 307 151 InDels ont été obtenus.
Résultats
Origine de B. napus
B.napusUn sous-génome a évolué à partir de l’ancêtre du navet européen.Un événement de flux génétique du navet européen vers leB. napus Un sous-génome est apparu il y a environ 106 à 1 170 ans.B.napusLe sous-génome C pourrait avoir évolué à partir de l’ancêtre commun de ces lignées.L'ancêtre deB.napusséparé de l'ancêtre commun de quatre sous-espèces de B. oleracea, avec un flux génétique récent dansB.napusIl y a environ 108 à 898 ans.B.napusLe sous-génome C a une origine plus complexe que le sous-génome A.Un fort goulot d'étranglement s'est développé dans les deux sous-génomes au coursB.napusévolution.L'hiver et le semi-hiverB.napusles écotypes ont divergé il y a environ 60 ans, alors que l'hiver et le printempsB.napusont divergé il y a environ 416 ans, et les graines oléagineuses et non oléagineusesB.napusa divergé il y a environ 277 ans.
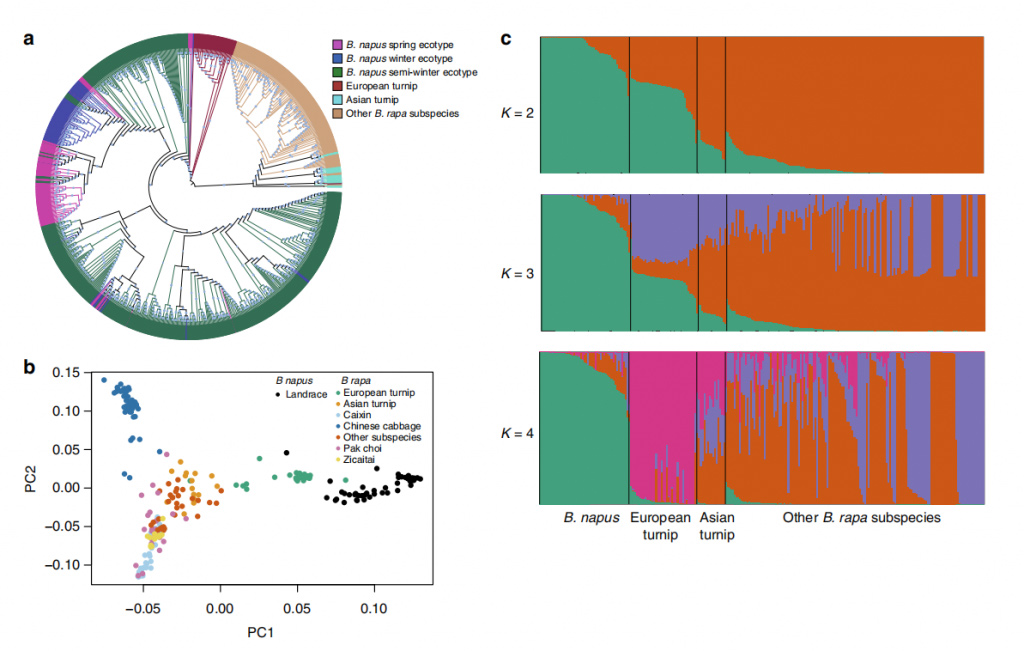
Fig. 2 Structure de la population de 588 accessions de B. napus et de 199 accessions de B. rapa.
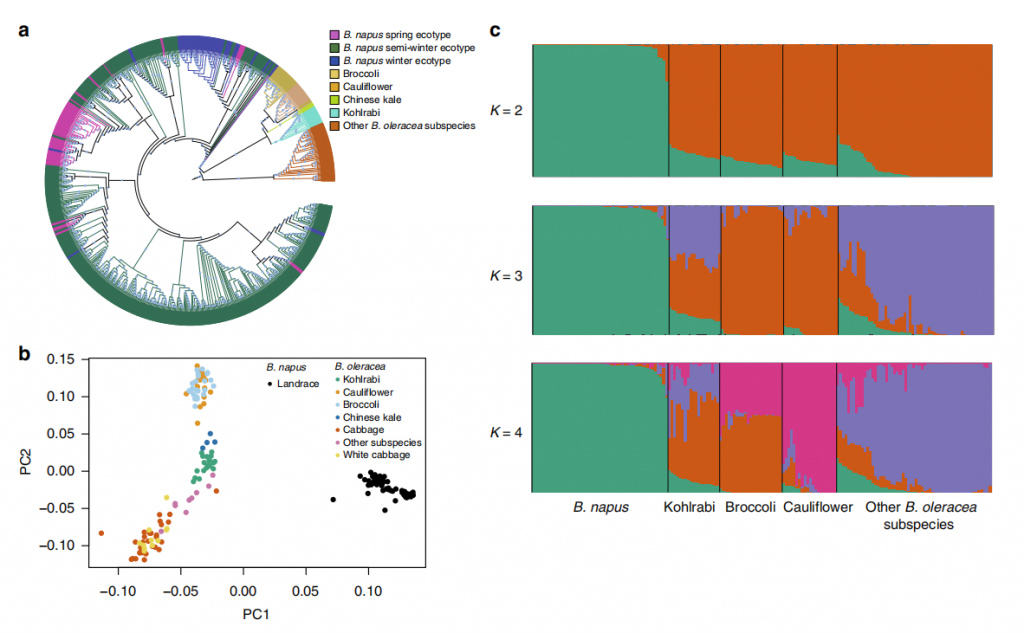
Fig. 3 Structure de la population de 588 accessions de B. napus et de 119 accessions de B. oleracea
Signaux de sélection et études d’association à l’échelle du génome.
Au cours de la première étape d’amélioration (FSI), davantage de diversité génétique a été perdue dans le sous-génome C de B. napus que dans le sous-génome A.Moins de différenciation génétique s'est produite au cours du FSI que lors de la deuxième étape d'amélioration (SSI).Les gènes des régions signal de sélection SSI ont été enrichis en termes de tolérance au stress, de développement et de voies métaboliques.60 locus significativement associés à 10 caractères cibles, dont 5 liés au rendement en graines, 3 à la longueur des siliques, 4 à la teneur en huile et 48 à la qualité des graines ont été identifiés.
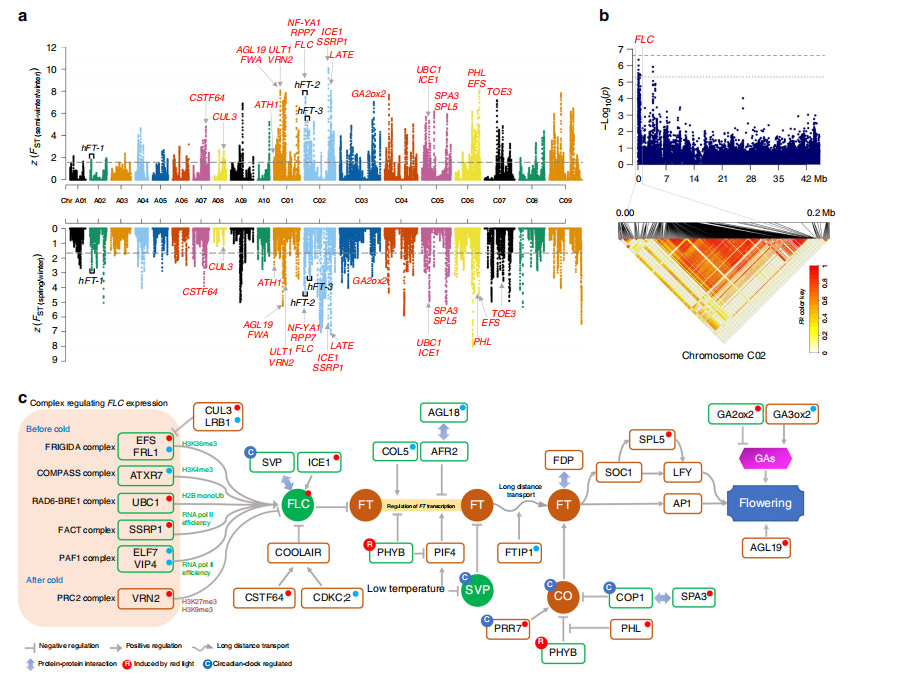
Fig. 4 Analyse du génome et annotations des régions sélectionnées lors du SSI de B. napus
Analyse du transcriptome
Les données RNAseq de 11 tissus provenant d'un cultivar à haute teneur en huile et doublement faible et d'un cultivar à faible teneur en huile et doublement élevée ont identifié des gènes liés au processus de biosynthèse des glucosinolates étaient significativement surreprésentées.
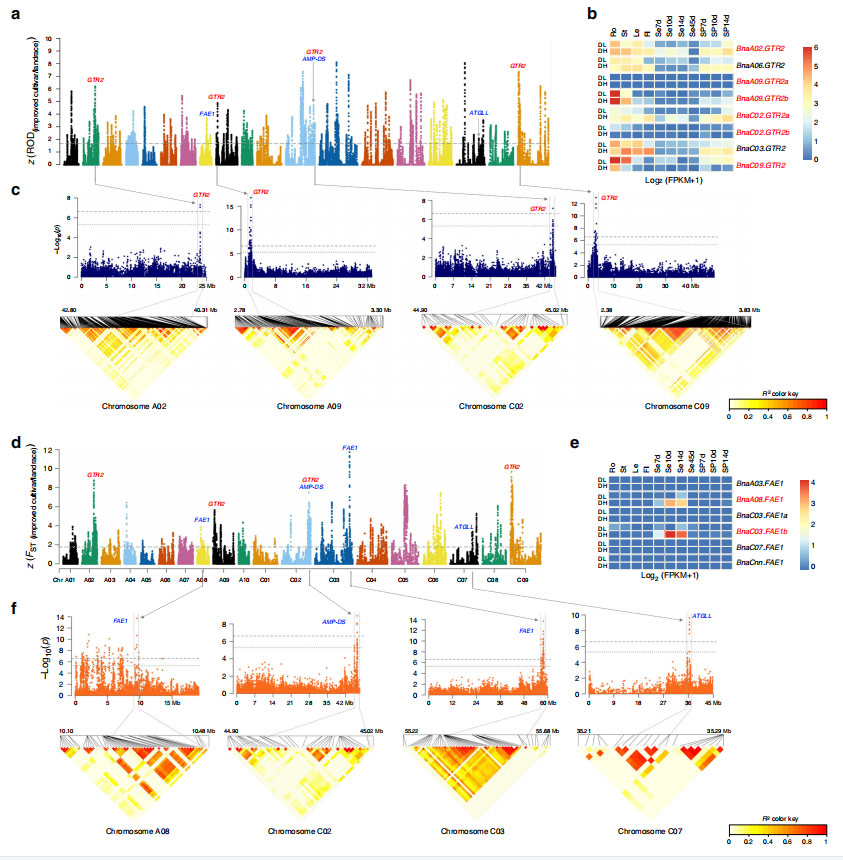
Fig. 5 Aperçu de la régulation de la période de floraison lors de la sélection de l'amélioration de l'écotype de B. napus
Discussion
Cette étude a fourni une ressource précieuse pour comprendre l’origine et l’historique d’amélioration deB.napuset facilitera la dissection des bases génétiques de caractères agronomiques complexes importants.Les SNP significatifs associés à des variantes favorables, des signaux de sélection et des gènes candidats contribueront largement à l'avenir, notamment à l'amélioration du rendement, de la qualité des graines, de la teneur en huile et de l'adaptabilité de cette culture allopolyploïde récente et de ses proches.
Référence
Le reséquençage du génome entier révèle l'origine de Brassica napus et les loci génétiques impliqués dans son amélioration[J].Communications naturelles, 2019, 10(1).
Actualités et faits saillants vise à partager les derniers cas réussis avec Biomarker Technologies, en capturant de nouvelles réalisations scientifiques ainsi que des techniques importantes appliquées au cours de l'étude.
Heure de publication : 05 janvier 2022

