Séquençage de l’exome humain entier
Avantages des services
● Région codante pour les protéines ciblées : en capturant et en séquençant la région codante pour les protéines, hWES est utilisé pour révéler des variantes liées à la structure des protéines ;
● Haute précision : avec une profondeur de séquençage élevée, hWES facilite la détection des variantes courantes et des variantes rares avec des fréquences inférieures à 1 % ;
● Rentable : le hWES génère environ 85 % des mutations de maladies humaines à partir de 1 % du génome humain ;
● Cinq procédures de contrôle qualité strictes couvrant l'ensemble du processus avec Q30>85 % garantis.
Exemples de spécifications
| Plate-forme
| Bibliothèque
| Stratégie de capture d'exons
| Recommander une stratégie de séquençage
|
|
Plateforme Illumina NovaSeq
| PE150 | Agilent SureSelect Humain Tous Exon V6 Panneau IDT xGen Exome Hyb V2 | 5 Go 10 Go |
Exemples d'exigences
| Échantillon type
| Montant(Qubit®)
| Volume
| Concentration
| Pureté (NanoDrop™) |
|
ADN génomique
| ≥ 300 ng | ≥ 15 µL | ≥ 20 ng/μL | OD260/280=1,8-2,0 aucune dégradation, aucune contamination
|
Profondeur de séquençage recommandée
Pour les troubles mendéliens/maladies rares : profondeur de séquençage efficace supérieure à 50×
Pour les échantillons de tumeurs : profondeur de séquençage efficace supérieure à 100 ×
Bioinformatique


Flux de travail des services

Livraison d'échantillon

Extraction d'ADN

Construction d'une bibliothèque

Séquençage

L'analyse des données

Livraison des données

Services après-vente
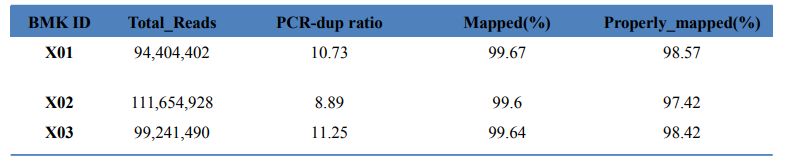
1.Statistiques d'alignement
Tableau 1 Statistiques du résultat de la carte
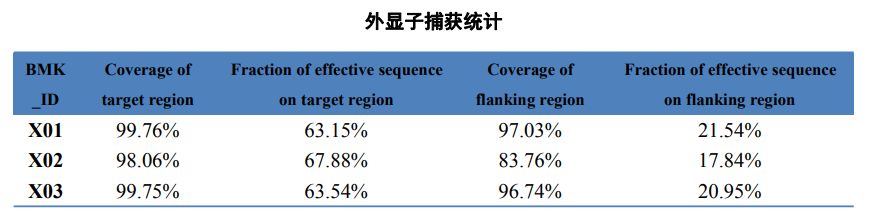
Tableau 2 Statistiques de capture d'exome
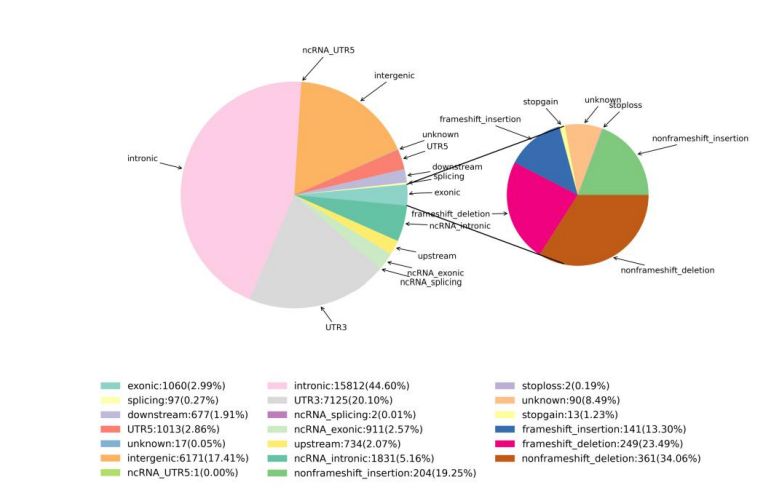
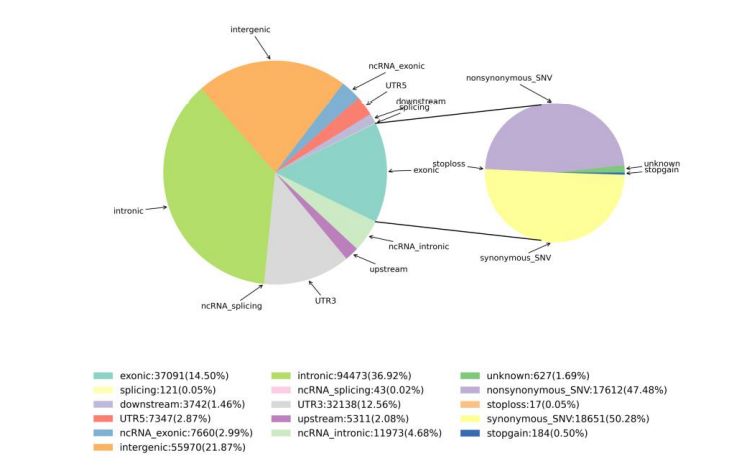
2. Détection des variations
Figure 1 Statistiques de SNV et InDel
3.Analyse avancée
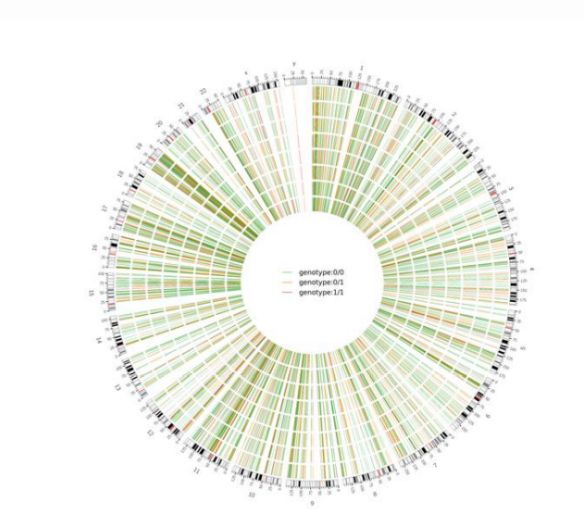
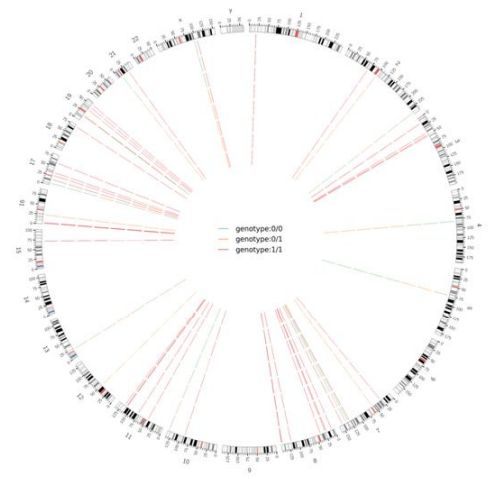
Figure 2 : Tracé Circos du SNV et de l'InDel nocifs à l'échelle du génome
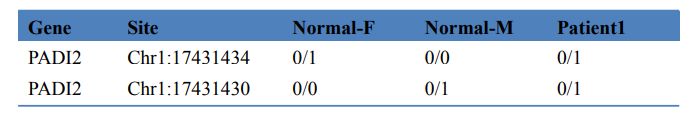
Tableau 3 Dépistage des gènes pathogènes