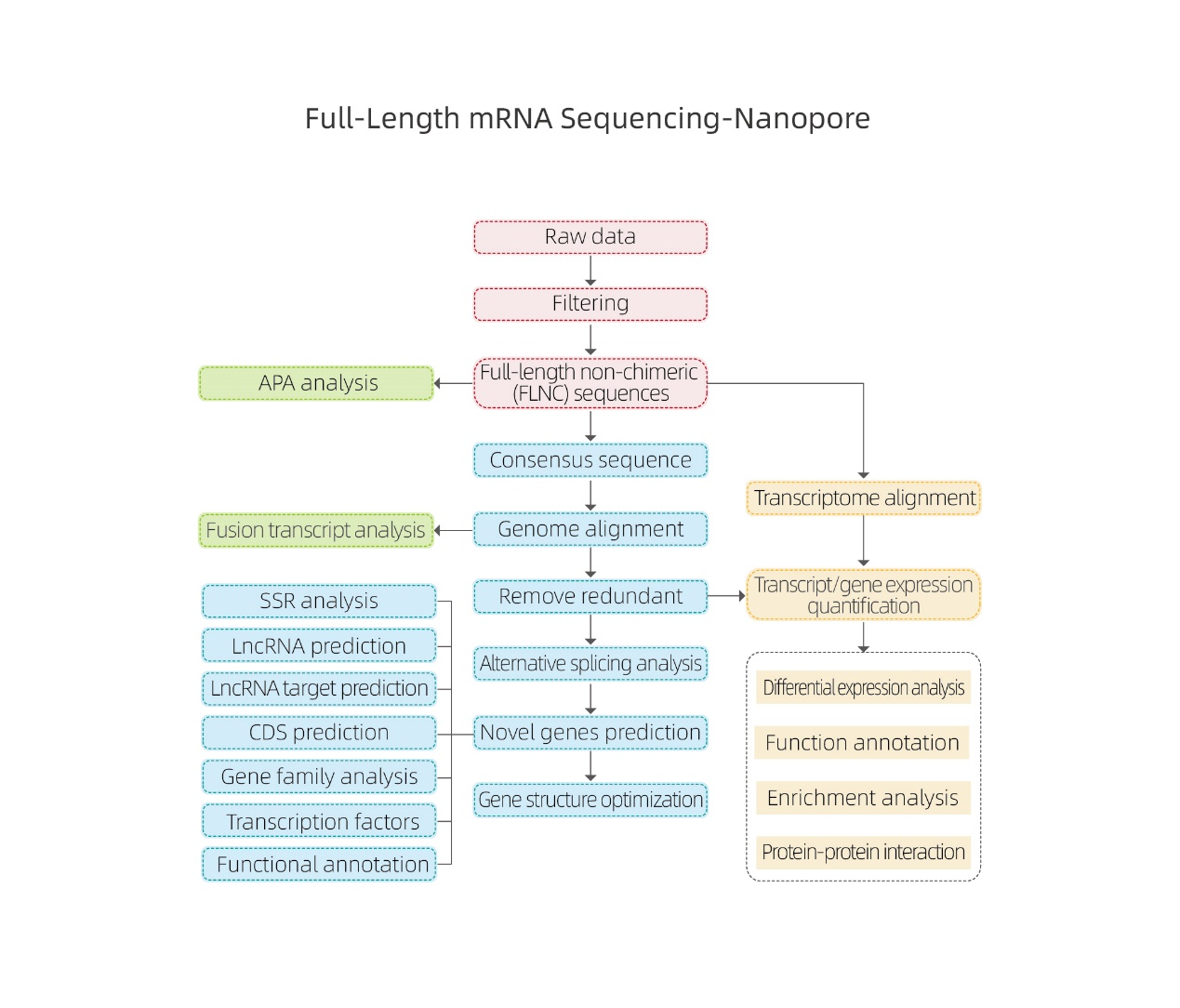
Séquençage complet de l'ARNm-Nanopore
Avantages des services
● Faible biais de séquence
● Révéler des molécules d'ADNc complètes
● Moins de données nécessaires pour couvrir le même nombre de relevés de notes
● Identification de plusieurs isoformes par gène
● Quantification d'expression au niveau isoforme
Spécifications des services
| Bibliothèque | Plate-forme | Rendement de données recommandé (Go) | Contrôle de qualité |
| ADNc-PCR (enrichi en Poly-A) | Nanopore ProméthION P48 | 6 Go/échantillon (selon les espèces) | Rapport sur toute la longueur > 70 % Score de qualité moyen : Q10
|
Analyses bioinformatiques
●Traitement des données brutes
● Identification du relevé de notes
● Épissage alternatif
● Quantification de l'expression au niveau du gène et au niveau des isoformes
● Analyse d'expression différentielle
● Annotation et enrichissement de fonctions (DEG et DET)
Exigences et livraison des échantillons
Exigences de l'échantillon :
Nucléotides :
| Conc. (ng/μl) | Quantité (μg) | Pureté | Intégrité |
| ≥ 100 | ≥ 0,6 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contamination limitée ou inexistante en protéines ou en ADN indiquée sur le gel. | Pour les plantes : RIN≥7,0 ; Pour les animaux : RIN≥7,5 ; 5,0≥28S/18S≥1,0 ; élévation de la ligne de base limitée ou inexistante |
Tissu : Poids (sec) : ≥1 g
*Pour les tissus inférieurs à 5 mg, nous recommandons d’envoyer un échantillon de tissu surgelé (dans l’azote liquide).
Suspension cellulaire : nombre de cellules = 3×106- 1×107
*Nous recommandons d’expédier du lysat cellulaire congelé.Dans le cas où cette cellule compte moins de 5×105, une surgélation dans l'azote liquide est recommandée, ce qui est préférable pour la micro-extraction.
Échantillons de sang : Volume≥1 ml
Livraison d’échantillon recommandée
Récipient : tube à centrifuger de 2 ml (le papier d'aluminium n'est pas recommandé)
Étiquetage des échantillons : Groupe + répétition, par exemple A1, A2, A3 ;B1, B2, B3... ...
Expédition : 2. Glace sèche : les échantillons doivent être emballés dans des sacs et enterrés dans de la glace sèche.
- Tubes RNAstable : les échantillons d'ARN peuvent être séchés dans un tube de stabilisation d'ARN (par exemple RNAstable®) et expédiés à température ambiante.
Flux de travail des services
Nucléotides :

Livraison d'échantillon

Construction d'une bibliothèque

Séquençage

L'analyse des données

Services après-vente
Flux de travail des services
Tissu:

Conception d'expériences

Livraison d'échantillon

Extraction d'ARN

Construction d'une bibliothèque

Séquençage

L'analyse des données

Services après-vente
1.Analyse d'expression différentielle - Tracé de volcan
L'analyse de l'expression différentielle peut être traitée à la fois au niveau des gènes pour identifier les gènes différentiellement exprimés (DEG) et au niveau des isoformes pour les identifier différentiellement.
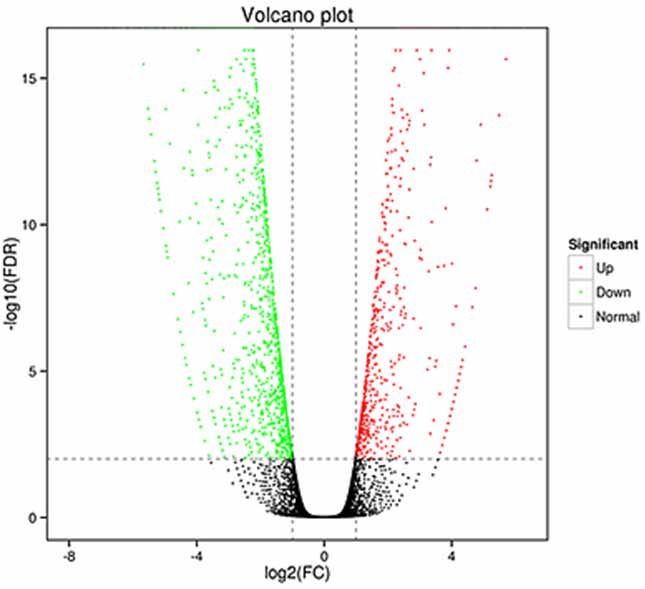
relevés de notes exprimés (DET)
2.Carte thermique de clustering hiérarchique
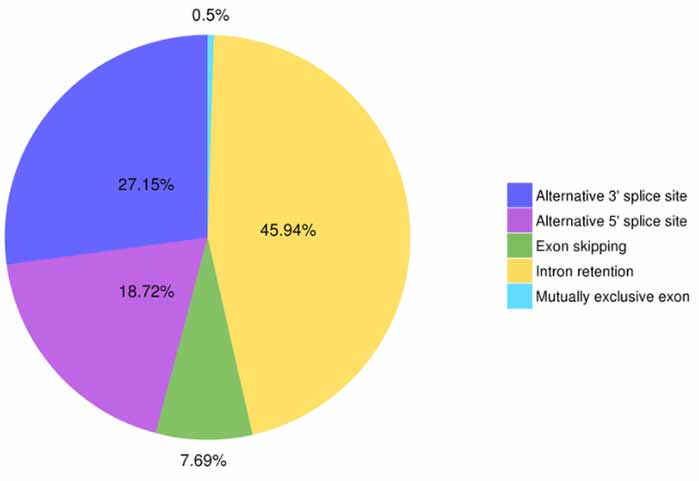
3. Identification et classification des épissages alternatifs
Cinq types d'événements d'épissage alternatifs peuvent être prédits par Astalavista.
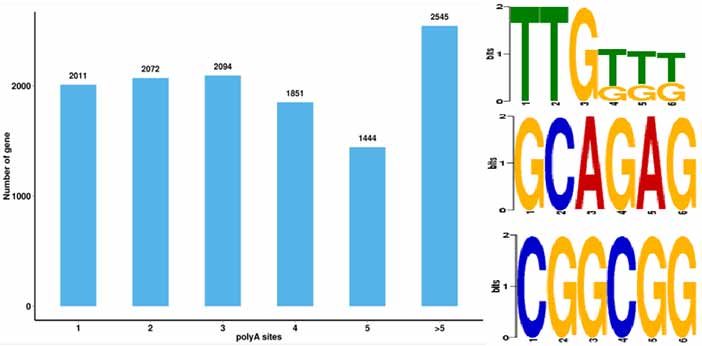
4.Identification des événements de polyadénylation alternative (APA) et motif à 50 pb en amont de poly-A
Affaire BMK
Identification d'épissage alternatif et quantification au niveau des isoformes par séquençage du transcriptome complet des nanopores
Publié :Communications naturelles, 2020
Stratégie de séquençage :
Regroupement : 1. CLL-SF3B1 (WT );2. CLL-SF3B1 (mutation K700E) ;3. Cellules B normales
Stratégie de séquençage : séquençage de bibliothèque MinION 2D, séquençage de bibliothèque PromethION 1D ;données à lecture courte à partir des mêmes échantillons
Plateforme de séquençage : Nanopore MinION ;Nanopore ProméthION ;
Résultats clés
1. Identification d'épissage alternatif au niveau isoforme
Les séquences à lecture longue permettent l'identification du mutant SF3B1K700E-sites d'épissage modifiés au niveau de l'isoforme.Il a été constaté que 35 3′SS alternatifs et 10 5′SS alternatifs étaient épissés de manière significativement différentielle entre SF3B1.K700Eet SF3B1WT.33 des 35 altérations ont été nouvellement découvertes par des séquences à lecture longue.
2.Quantification de l'épissage alternatif au niveau isoforme
Expression des isoformes de rétention d'intron (IR) dans SF3B1K700Eet SF3B1WTont été quantifiés sur la base de séquences de nanopores, révélant une régulation négative globale des isoformes IR dans SF3B1K700E.
Référence
Tang AD , Soulette CM , Baren MJV et al.La caractérisation complète de la transcription de la mutation SF3B1 dans la leucémie lymphoïde chronique révèle une régulation négative des introns retenus [J].Communication naturelle.