Séquençage de l'ARNm eucaryote-Illumina
Avantages
● Très expérimenté : plus de 200 000 échantillons ont été traités dans BMK couvrant divers types d'échantillons, notamment la culture cellulaire, les tissus, les fluides corporels, etc. et plus de 7 000 projets de séquençage d'ARNm clôturés couvrant divers domaines de recherche.
● Système de contrôle qualité strict : les principaux points de contrôle qualité à toutes les étapes, y compris la préparation des échantillons, la préparation de la bibliothèque, le séquençage et la bioinformatique, sont étroitement surveillés afin de fournir des résultats de haute qualité.
● Plusieurs bases de données disponibles pour les études d'annotation et d'enrichissement des fonctions afin de répondre à divers objectifs de recherche.
● Services après-vente : Services après-vente valables 3 mois après la fin du projet, comprenant le suivi des projets, le dépannage, les questions-réponses sur les résultats, etc.
Exigences et livraison des échantillons
| Bibliothèque | Stratégie de séquençage | Données recommandées | Contrôle de qualité |
| Poly A enrichi | Illumina PE150 | 6 Go | Q30≥85 % |
Exigences de l'échantillon :
Nucléotides :
| Conc. (ng/μl) | Quantité (μg) | Pureté | Intégrité |
| ≥20 | ≥ 0,5 | DO260/280=1,7-2,5 DO260/230=0,5-2,5 Contamination limitée ou inexistante en protéines ou en ADN indiquée sur le gel. | Pour les plantes : RIN≥6,5 ; Pour les animaux : RIN≥7,0 ; 5,0≥28S/18S≥1,0 ; élévation de la ligne de base limitée ou inexistante |
Tissu : Poids (sec) :≥1 g
*Pour les tissus inférieurs à 5 mg, nous recommandons d’envoyer un échantillon de tissu surgelé (dans l’azote liquide).
Suspension cellulaire :Nombre de cellules = 3×106- 1×107
*Nous recommandons d’expédier du lysat cellulaire congelé.Dans le cas où cette cellule compte moins de 5×105.
Échantillons de sang:Volume≥1 ml
Micro-organisme:Masse ≥ 1 g
Livraison d’échantillon recommandée
Récipient : tube à centrifuger de 2 ml (le papier d'aluminium n'est pas recommandé)
Étiquetage des échantillons : Groupe + répétition, par exemple A1, A2, A3 ;B1, B2, B3... ...
Expédition:
- Glace carbonique : les échantillons doivent être emballés dans des sacs et enterrés dans de la neige carbonique.
- Tubes RNAstable : les échantillons d'ARN peuvent être séchés dans un tube de stabilisation d'ARN (par exemple RNAstable®) et expédiés à température ambiante.
Flux de travail des services

Conception d'expériences

Livraison d'échantillon

Extraction d'ARN

Construction d'une bibliothèque

Séquençage

L'analyse des données

Services après-vente
Bioinformatique
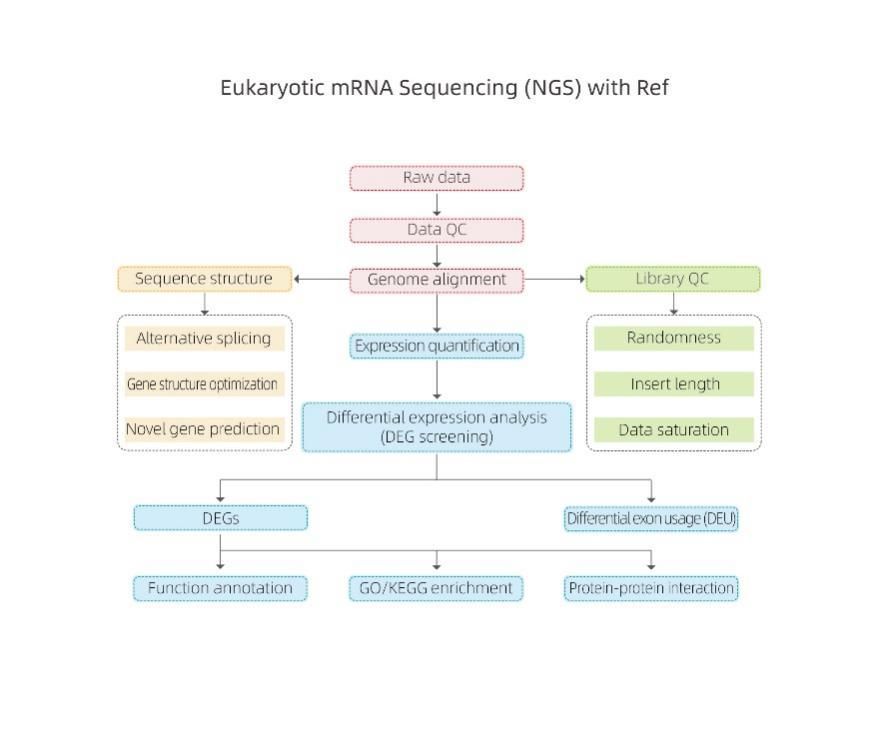
Eucaryote Flux de travail d'analyse de séquençage d'ARNm
Bioinformatique
ØContrôle qualité des données brutes
ØAlignement du génome de référence
ØAnalyse de la structure de la transcription
ØQuantification des expressions
ØAnalyse d'expression différentielle
ØAnnotation et enrichissement des fonctions
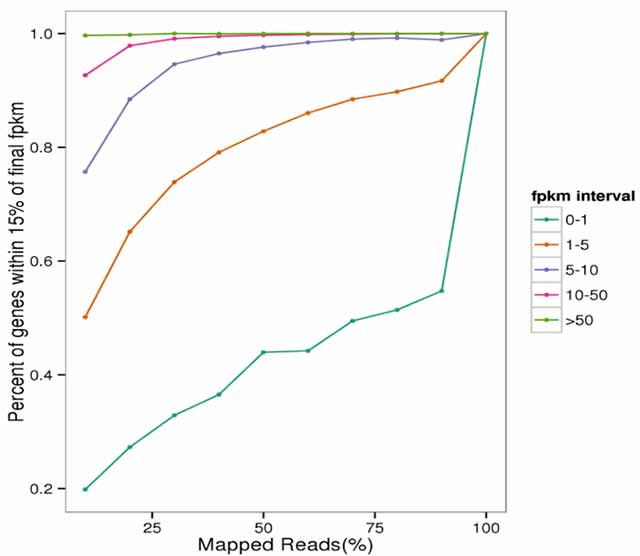
1.Courbe de saturation des données d'ARNm
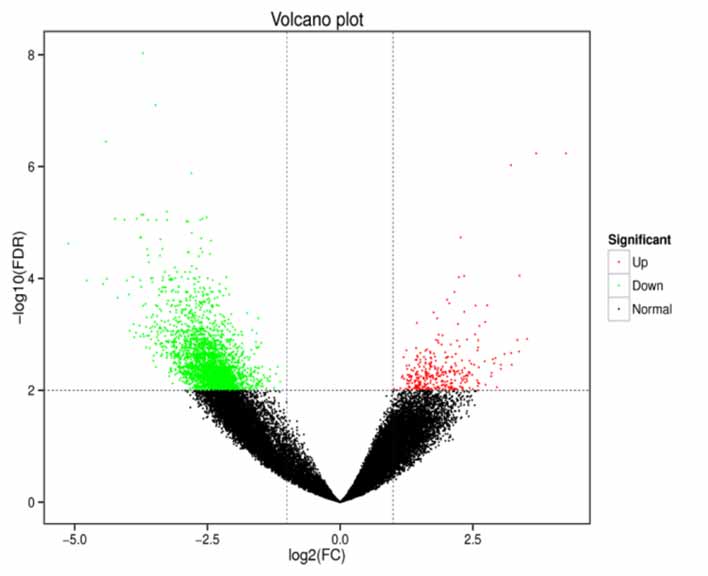
2.Analyse d'expression différentielle - Tracé de volcan
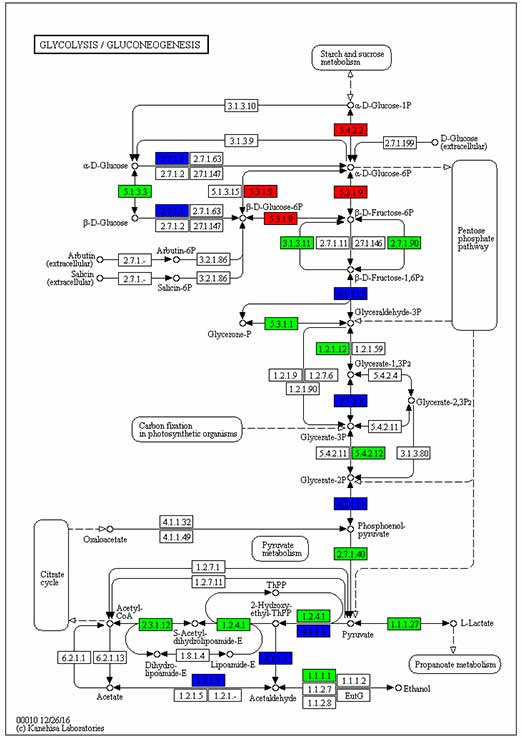
3.Annotation KEGG sur les DEG
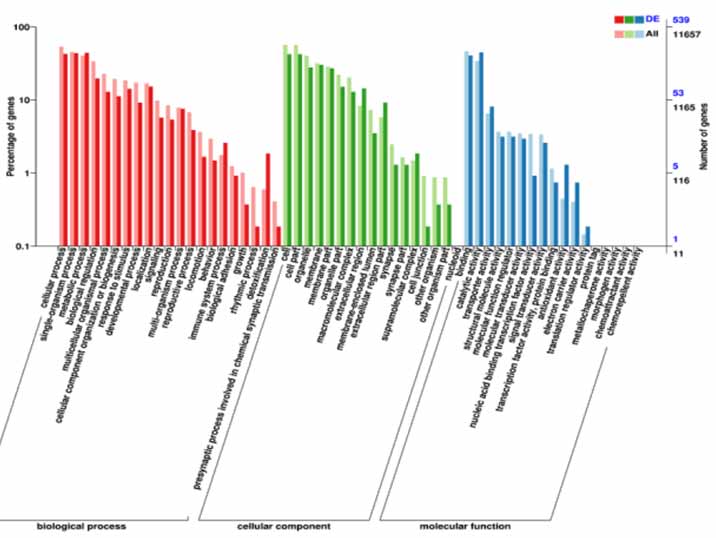
4.Classification GO sur les DEG