تکامل ژنوم
ژنتیک طبیعت
یک مجموعه ژنومی با کیفیت بالا ویژگی های ژنومی چاودار و ژن های مهم زراعی را برجسته می کند.
PacBio |ایلومینا |نقشه نوری Bionano |مونتاژ ژنوم Hi-C |نقشه ژنتیکی |جاروهای انتخابی |RNA-Seq |ISO-seq |SLAF-seq
Biomarker Technologies در این مطالعه از توالی یابی Pacbio، توالی یابی Hi-C و تجزیه و تحلیل داده ها پشتیبانی فنی کرد.
نکات برجسته
1. اولین ژنوم چاودار با کیفیت بالا در سطح کروموزومی به دست آمد که دارای یک کروموزوم منفرد بزرگتر از 1 گیگابایت است.
2. در مقایسه با ژنوم Tu، Aet و Hv، یک رویداد LTR-RT اخیر منحصر به فرد در ژنوم چاودار مشاهده شد که مسئول گسترش اندازه ژنوم چاودار بود.
3. واگرایی بین گندم چاودار و دیپلوئید پس از جداسازی جو از گندم اتفاق افتاد که زمان واگرایی برای این دو رویداد تقریباً 9.6 و 15 MYA بود.
فسفوریلاسیون ژن های FT ممکن است صفت اولیه را در چاودار کنترل کند.
4. تجزیه و تحلیل رفت و برگشت انتخابی نشان دهنده دخالت احتمالی ScID1 در تنظیم تاریخ سرفصل و انتخاب احتمالی آن توسط اهلی سازی در چاودار است.
زمینه
زمینه
چاودار یک محصول غذایی با ارزش و علوفه، یک منبع ژنتیکی مهم برای اصلاح گندم و تریتیکاله، و یک ماده ضروری برای مطالعات ژنومیک مقایسه ای کارآمد در علف ها است.چاودار Weining، گونهای زود گل که در چین کشت میشود، به دلیل مقاومت طیف گستردهای که در برابر کپک پودری و زنگ راه راه دارد، برجسته است.برای درک مبانی ژنتیکی و مولکولی صفات نخبه چاودار و ترویج مطالعات ژنومی و اصلاحی در چاودار و محصولات مرتبط، ما در اینجا ژنوم چاودار Weining را توالی یابی و تجزیه و تحلیل کردیم.
دستاوردها
ژنوم چاودار
ژنوم چاودار با ترکیب خواندن های PacBio SMRT، توالی خوانی کوتاه Illumina، و همچنین آنهایی که از گرفتن ترکیب کروماتین (Hi-C)، نقشه برداری ژنتیکی، و تجزیه و تحلیل BioNano ساخته شده است.کانتیگ های مونتاژ شده (7.74 گیگابیت) 98.47 درصد از اندازه ژنوم تخمینی (7.86 گیگابیت) را تشکیل می دهند، با 93.67 درصد از کانتیگ ها (7.25 گیگابایت) به هفت کروموزوم اختصاص داده شده است.عناصر تکراری 90.31 درصد از ژنوم مونتاژ شده را تشکیل می دادند.
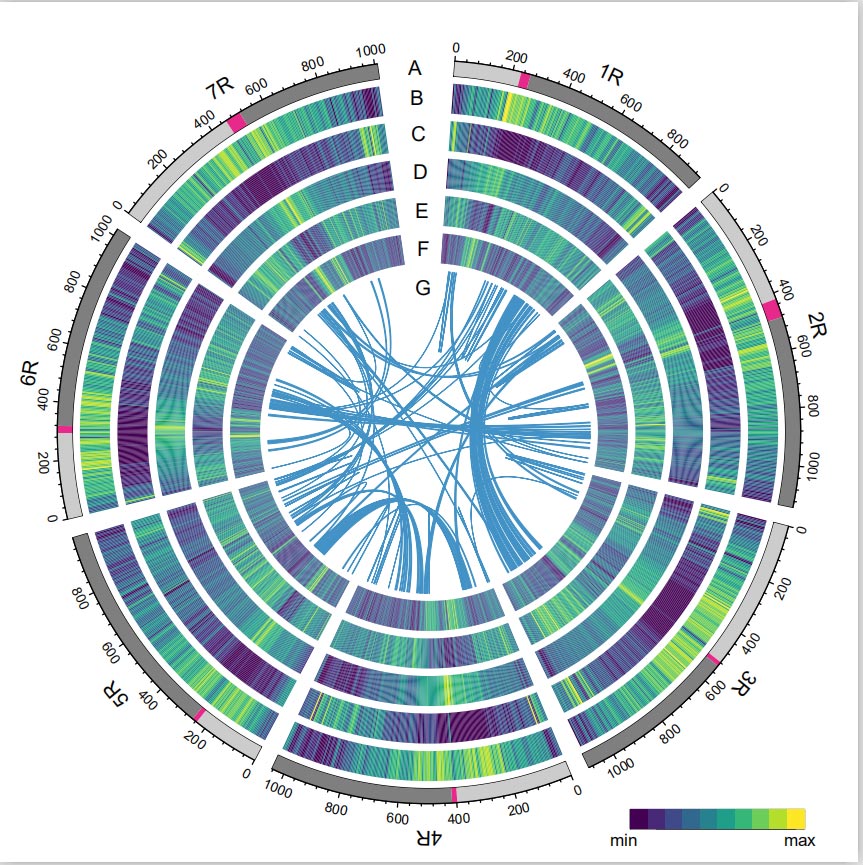
ژنوم چاودار
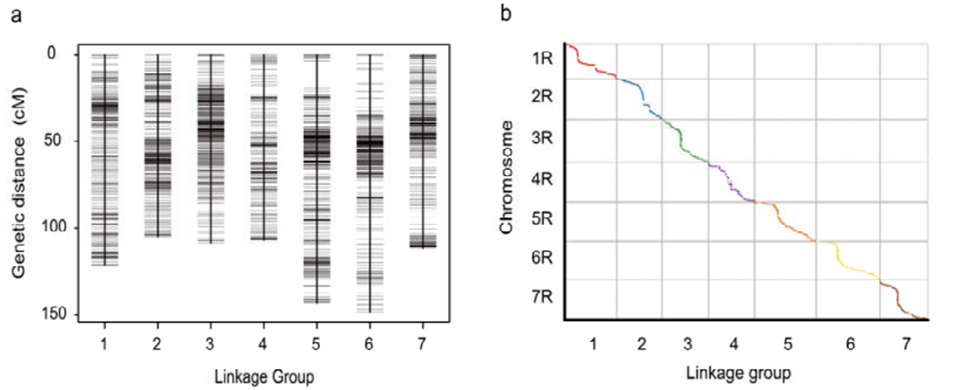
نقشه پیوند ژنتیکی (WJ) با استفاده از 295 گیاه F2 حاصل از تلاقی دو توده چاودار (Weining × Jingzhou) ایجاد شده است.
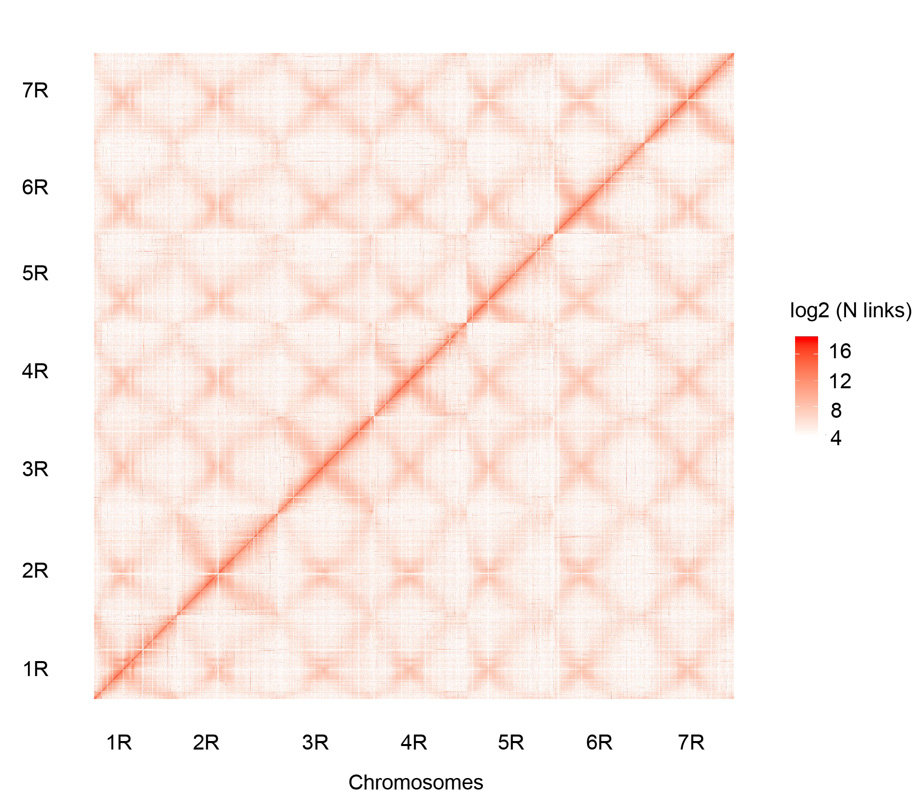
نقشه تماس Hi-C از هفت کروموزوم چاودار Weining مونتاژ شده (1R - 7R)
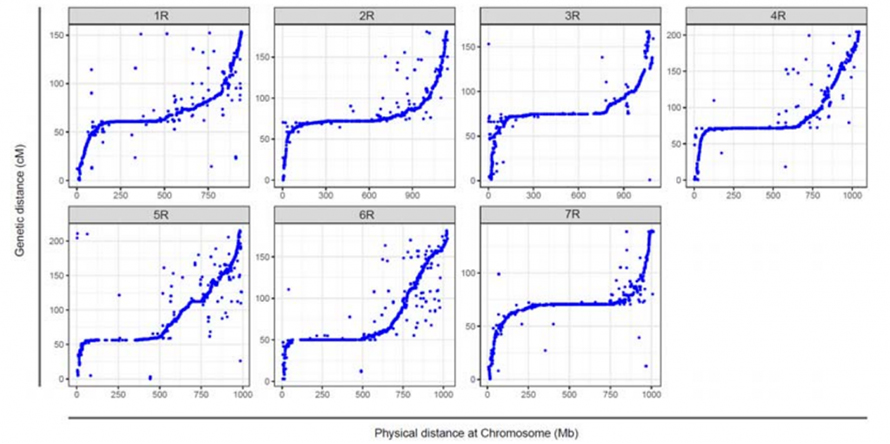
هم ترازی بین هفت کروموزوم مونتاژ شده چاودار Weining و هفت گروه پیوند چاودار با استفاده از جمعیت RIL Lo7 x Lo255 ایجاد شد.
مقدار شاخص مونتاژ LTR (LAI) ژنوم چاودار 18.42 و 1393 (96.74%) از 1440 ژن BUSCO بسیار حفاظت شده شناسایی شد. این نتایج نشان می دهد که توالی ژنوم چاودار Weining در هر دو بین ژنی از کیفیت بالایی برخوردار است و مناطق ژنتیکیدر مجموع 86991 ژن کد کننده پروتئین شامل 45596 ژن با اطمینان بالا (HC) و 41395 ژن با اطمینان پایین (LC) پیشبینی شد.
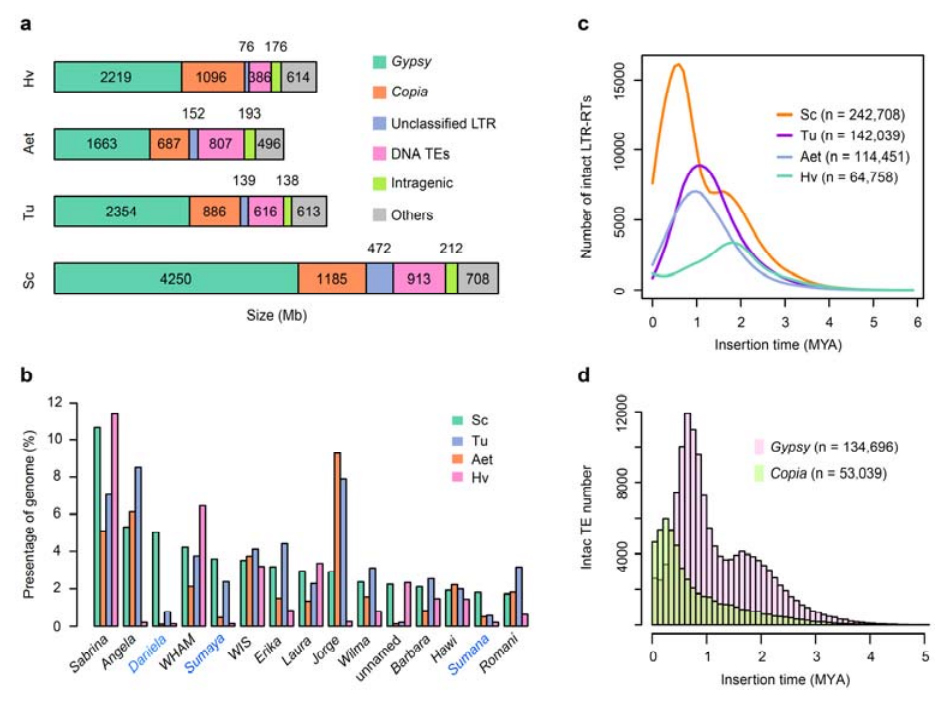
2. تجزیه و تحلیل TEs
تجزیه و تحلیل TEs.در مجموع 6.99 گیگابیت، که 90.31 درصد از مجموعه Weining را نشان می دهد، به عنوان TE مشروح شد که شامل 2,671,941 عنصر متعلق به 537 خانواده است.این محتوای TE به وضوح بالاتر از آنچه قبلا برای Ta (84.70٪)، Tu (81.42٪)، Aet (84.40٪)، WEW (82.20٪)، یا Hv (80.80٪) گزارش شده بود، بالاتر بود.رتروترانسپوزون های طولانی ترمینال تکرار (LTR-RTs)، از جمله Gypsy، Copia و عناصر RT طبقه بندی نشده، TE های غالب بودند و 84.49 درصد از محتوای TE مشروح و 76.29 درصد از ژنوم Weining مونتاژ شده را اشغال کردند.ترانسپوزون های DNA CACTA دومین TE های فراوان بودند که 11.68 درصد از محتوای TE مشروح شده و 10.55 درصد از ژنوم Weining مونتاژ شده را تشکیل می دادند.
تجزیه و تحلیل عناصر ترانسپوزون چاودار
چاودار Weining نسبت نسبتاً بالایی از درجهای اخیر LTR-RT با اوج تقویت حدود 0.5 میلیون سال پیش (MYA) داشت که جدیدترین در میان چهار گونه بود.پیک دیگر، تقریباً 1.7 MYA رخ داد، مسنتر بود و در جو نیز دیده میشد.در سطح ابرخانواده، انفجارهای بسیار اخیر عناصر Copia در چاودار Weining در 0.3 MYA پیدا شد، در حالی که تقویت RTs کولی غالباً الگوی توزیع دووجهی دینامیک انفجار LTR-RT را شکل داد.
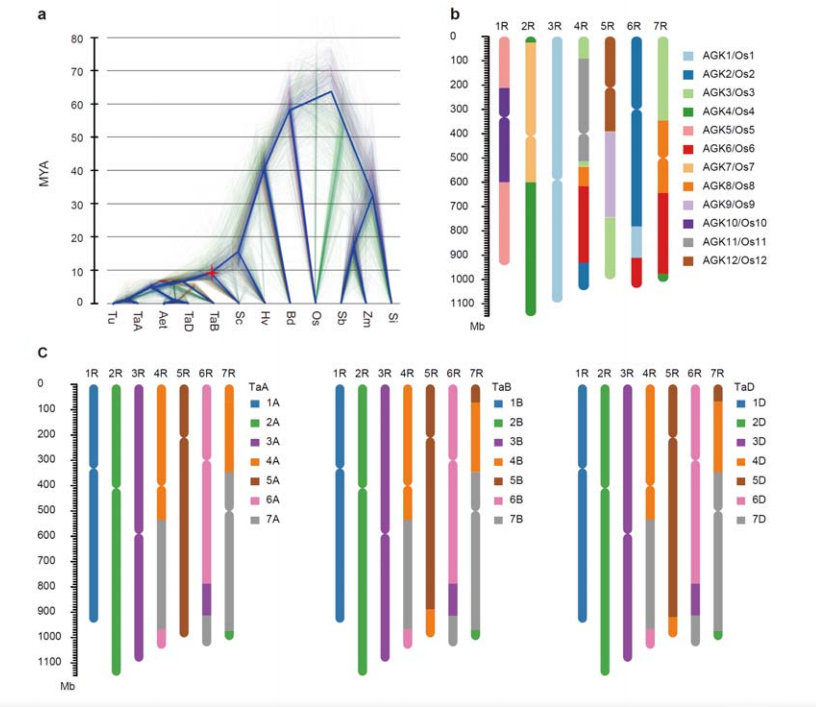
3. بررسی تکامل ژنوم چاودار و syntenies کروموزوم
واگرایی بین گندمهای چاودار و دیپلوئید پس از جداسازی جو از گندم صورت گرفت، با زمان واگرایی برای این دو رویداد به ترتیب تقریباً 9.6 و 15 MYA.1R، 2R، 3R به ترتیب با کروموزوم های 1، 2 و 3 گندم کاملاً هم خط بودند.4R، 5R، 6R، 7R یافت شد که همجوشی ها و قطعات در مقیاس بزرگ وجود دارد.
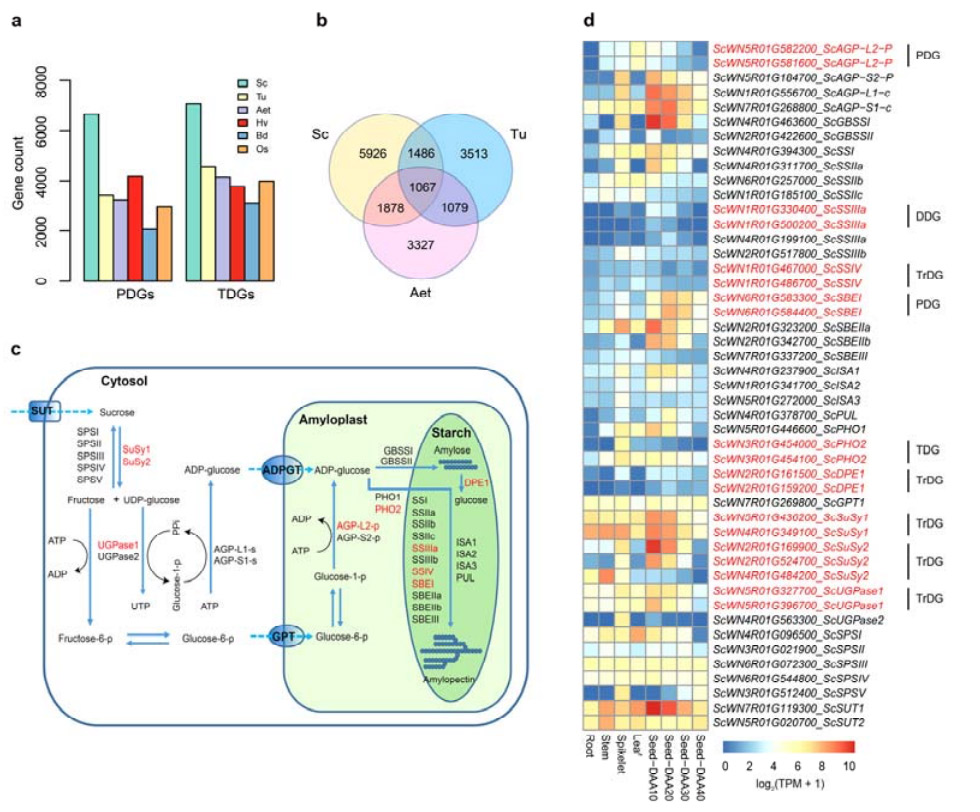
4. تجزیه و تحلیل تکرار ژن و تاثیر آنها بر ژن های بیوسنتز نشاسته
قابلتوجه، تعداد ژنهای تکراری پشت سر هم (TDGs) و ژنهای تکراری نزدیک (PDGs) چاودار Weining هر دو بیشتر از آنهایی بود که برای Tu، Aet، Hv، Bd و Os یافت شد.ژنهای تکراری جابجا شده (TrDGs) نیز از آنهایی که به طور خاص برای Tu و Aet یافت میشوند، بیشتر بودند.گسترش ژنوم چاودار با تعداد بیشتری از تکرار ژن همراه است.افزایش انفجار TE در چاودار ممکن است منجر به افزایش تعداد TrDG شود.
تجزیه و تحلیل سینتنی تکاملی و کروموزوم ژنوم چاودار
تجزیه و تحلیل تکثیر ژن چاودار و تأثیر آنها بر تنوع ژنهای مرتبط با بیوسنتز نشاسته (SBRGs)
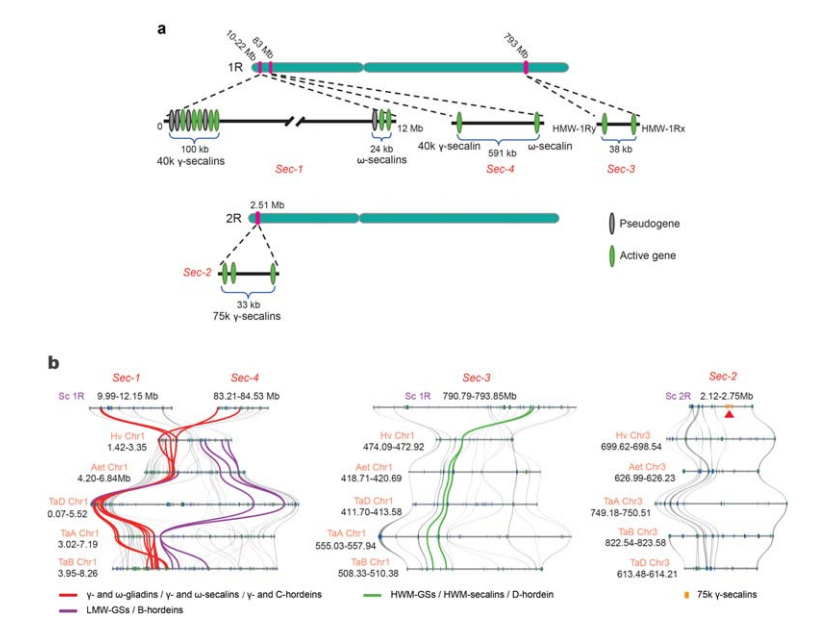
5. تشریح جایگاه های ژنی پروتئین ذخیره سازی دانه چاودار (SSP).
چهار جایگاه کروموزومی (Sec-1 تا Sec-4) که SSPs چاودار را مشخص میکنند در 1R یا 2R شناسایی شدهاند.ژن α-گلیادین تنها اخیراً در گندم و گونه های نزدیک به هم پس از واگرایی گندم از چاودار تکامل یافته است.
6. بررسی فاکتور رونویسی (TF) و ژن های مقاومت به بیماری
تجزیه و تحلیل جایگاه سکولین چاودار
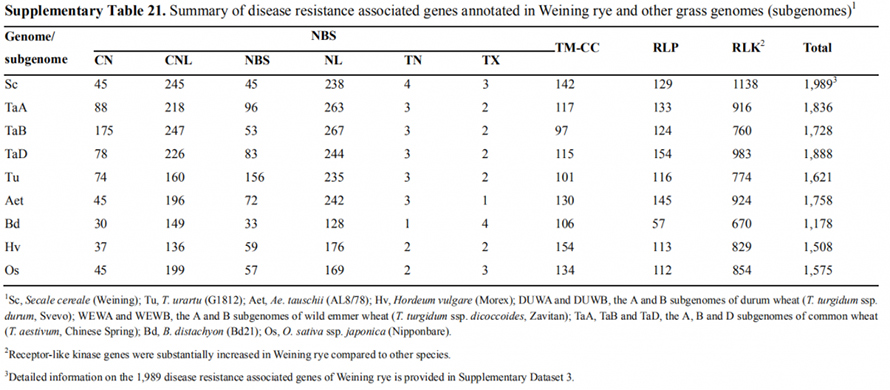
چاودار Weining دارای ژن های مرتبط با مقاومت به بیماری (DRA) (1989، داده های تکمیلی 3) نسبت به Tu (1621)، Aet (1758)، Hv (1508)، Bd (1178)، Os (1575) و A (1836) بود. )، B (1728) و D (1888) زیر ژنوم گندم معمولی.
7. بررسی ویژگی های بیان ژن مرتبط با صفت عنوان اولیه
دو ژن FT با بیان نسبتاً بالا در شرایط روز طولانی، ScFT1 و ScFT2، در مجموعه ژنوم Weining مشروح شدند.دو باقی مانده اسید آمینه فسفوریلاسیون ScFT2 (S76 و T132) با کاهش کنترل زمان ارتباط پیدا کردند.
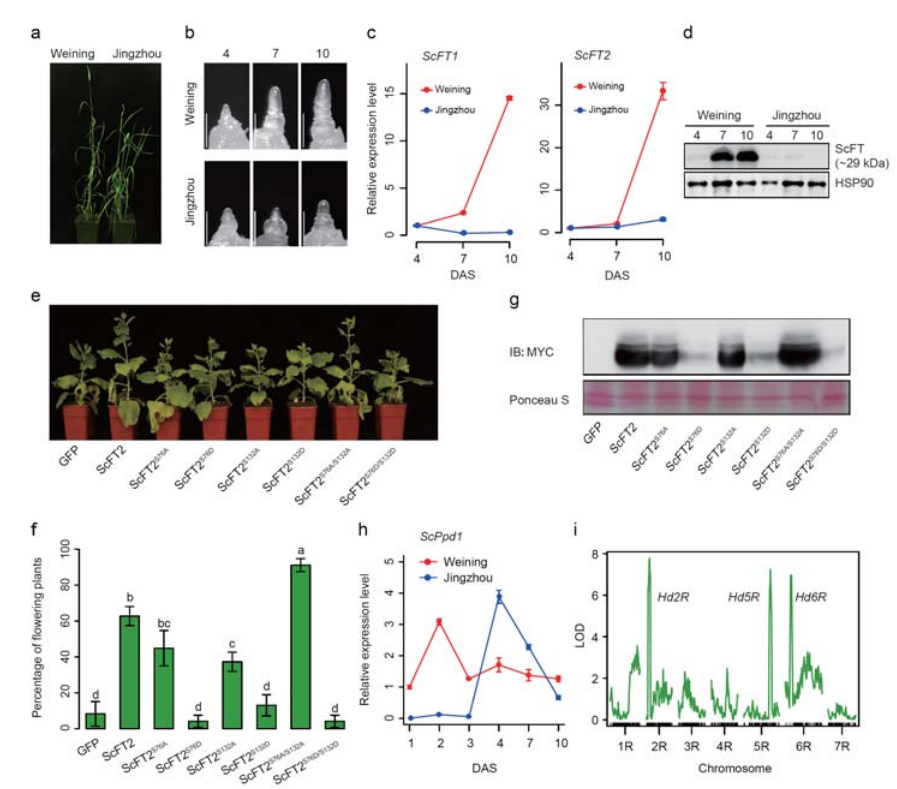
ویژگی های رشد و بیان ژن مرتبط با صفت اولیه چاودار Weining
8. استخراج نواحی کروموزومی و جایگاه هایی که بالقوه در اهلی سازی چاودار نقش دارند
در مجموع 123647 SNP برای انجام تجزیه و تحلیل رفت و برگشتی بین چاودار کشت شده و S. vavilovii استفاده شد.11 سیگنال رفت و برگشت انتخابی با شاخص کاهش (DRI)، شاخص تثبیت (FST) و روش XP-CLR شناسایی شدند.ScID1 دخالت احتمالی در تنظیم تاریخ عنوان پیدا شد.
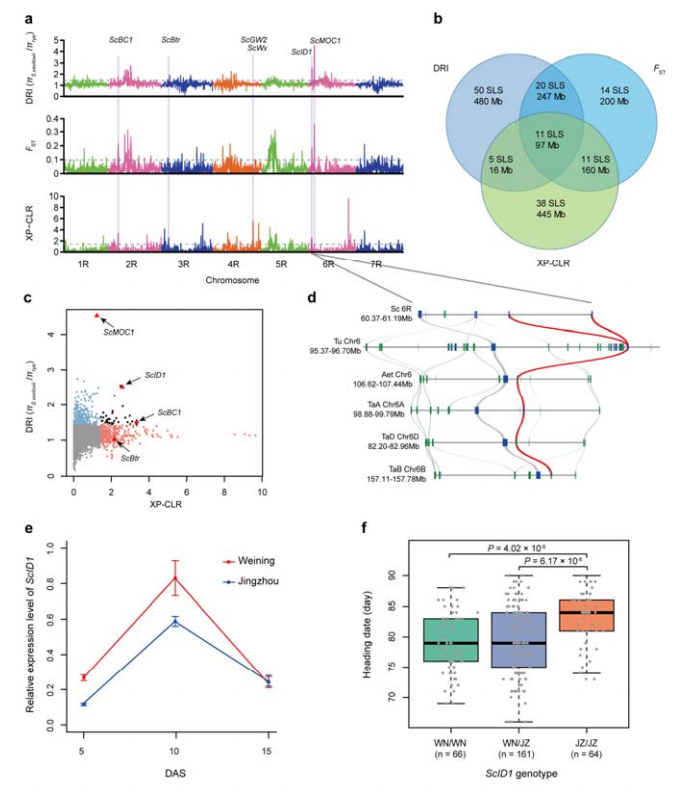
شناسایی و تجزیه و تحلیل نواحی کروموزومی و جایگاههای بالقوه مرتبط با اهلی شدن چاودار
ارجاع
لی GW و همکارانیک مجموعه ژنومی با کیفیت بالا ویژگی های ژنومی چاودار و ژن های مهم زراعی را برجسته می کند.ژنتیک طبیعت (2021)
اخبار و نکات مهم هدف آن به اشتراک گذاری آخرین موارد موفق با Biomarker Technologies، ثبت دستاوردهای علمی جدید و همچنین تکنیک های برجسته به کار رفته در طول مطالعه است.
زمان ارسال: ژانویه-05-2022

