
Transkriptoma osoaren sekuentziazioa - Illumina
Zerbitzu Abantailak
● RNA mota guztietako zenbatespena, zenbaketari, adierazpenari eta kromosometan oinarritutako adierazpen erlatiboei dagokienez.
● Diferentzialki adierazitako RNAak eta dagozkion adierazpenak identifikatzea
● Gene-adierazpenaren analisia
● ceRNA sarearen azterketa
● Funtsezko geneek bide-analisia inplikatu zuten
● BMKCloud-en oinarritutako emaitzak entrega: datu-meatzaritza pertsonalizatua eskuragarri dago plataforman
● Salmenta osteko zerbitzuak 3 hilabeterako balio du proiektua amaitzean
Lagin-baldintzak eta entrega
| Liburutegia | Plataforma | Gomendatutako datuak | Datuen QC |
| RNA osoa | Illumina PE150&SE50 | Circ/lnc/mRNA: 16G;miRNA: 10M irakurketa | Q30≥85% |
Lagin-baldintzak:
Nukleotidoak:
| Garbitasuna | Osotasuna | Kutsadura | Zenbatekoa |
| OD260/280≥1.7-2.5; OD260/230≥0.5-2.5; | Landareetarako: RIN≥6,5;Animalientzat: RIN≥7;28S/18S≥1.0;Oinarrizko kota mugatua edo ez | Gelan agertzen da proteina edo DNA kutsadura mugatua edo ez. | Konk.≥100 ng/μl;Bolumena ≥ 10 μl;Guztira ≥ 2 μg |
Ehuna: Pisua (lehorra): ≥1 g
*5 mg baino txikiagoak diren ehunetarako, izoztutako (nitrogeno likidoan) ehun lagina bidaltzea gomendatzen dugu.
Zelula esekidura: Zelula kopurua = 3×107
* Izoztutako zelula lisatua bidaltzea gomendatzen dugu.Zelula hori 5×105 baino txikiagoa bada, nitrogeno likidoan izoztutako flasha gomendatzen da.
Odol laginak:
PA×geneBloodRNATube;
6 mlTRIzol eta 2 ml odol (TRIzol:Odola=3:1)
Laginaren entrega gomendatua
Edukiontzia:
2 ml zentrifuga-hodi (ez da gomendagarria eztainu-papera)
Laginaren etiketatzea: Taldea+erreplika, adibidez, A1, A2, A3;B1, B2, B3....
Bidalketa:
1.Izotz lehorra: laginak poltsetan sartu eta izotz lehorrean lurperatu behar dira.
2.RNAstable hodiak: RNA laginak RNA egonkortzeko hodian lehortu daitezke (adibidez, RNAstable®) eta giro-tenperaturan bidal daitezke.
Zerbitzuaren Lan Fluxua

Esperimentuen diseinua

Laginaren entrega

RNA erauzketa

Liburutegiaren eraikuntza

Sekuentziazioa

Datuen azterketa

Salmenta osteko zerbitzuak
Bioinformatika
1. RNA mota guztietan oinarritutako adierazpen erlatiboaren estimazioa
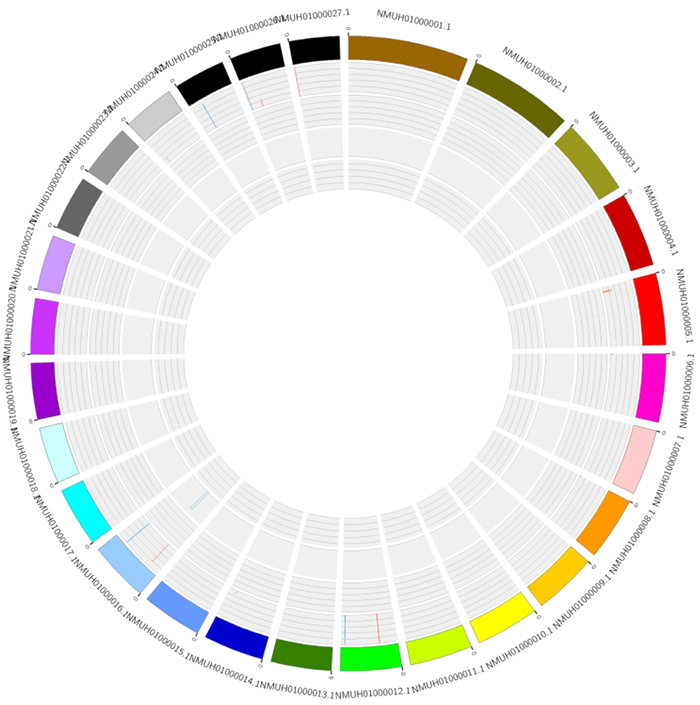
Zirkusuak RNAren adierazpenaren desberdintasunen esangurari buruz
2.KEGG bide-sare integratua
KEGG bide-sare integratua
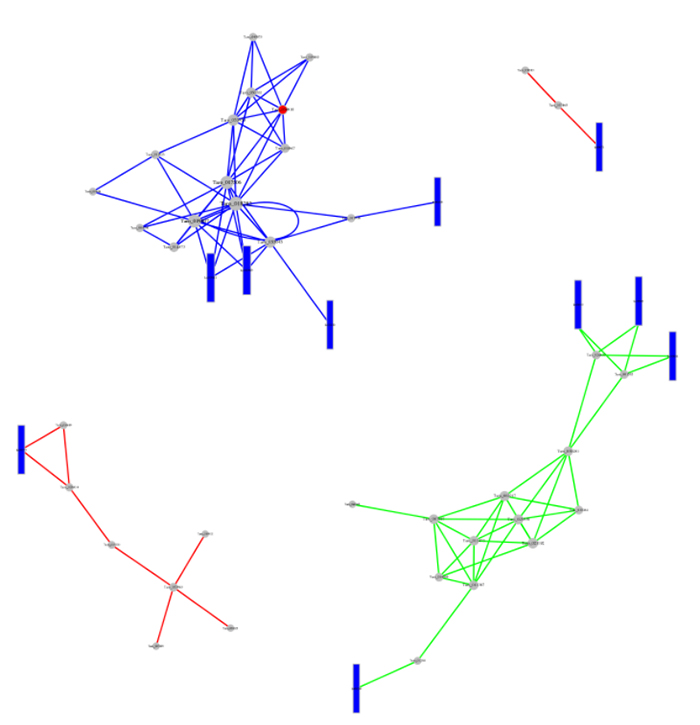
3.ceRNA sarearen analisia
ceRNA Sarean oinarritutako DE-circRNA-miRNA-mRNA elkarrekintzak
BMK kasua
CircRNA Adierazpen Eredua eta ceRNA eta miRNA-mRNA sareak Brassica campestris-en CMS lerroan anter garapenean parte hartzen dute
Argitaratu:Zientzia Molekularren Nazioarteko Aldizkaria,2019
NONMMUT015812-knockdown KP (shRNA-2) zelulak eta kontrol negatiboa (sh-Scr) zelulak infekzio biriko zehatz baten 6. egunean lortu ziren.
Emaitza nagusiak
Azterketa honek Polima zitoplasma maskulinoaren esterilitatea (CMS) "Bcpol97-05A" lerroa eta "Bcajh97-01B" lerro emankorra ezarri zituen Brassica campestris L. ssp.chinensis Makino, syn.B. rapa ssp.chinensis, eta RNA adierazpenaren profilaren konparaketak egin zituen lerro antzuaren eta lerro emankorren lore-begien artean, transkriptoma osoko sekuentziazioaren bidez.
1.Guztira 31 diferentzialki adierazitako (DE) circRNA, 47 DE miRNA eta 4779 DE mRNA identifikatu ziren.
2. Geneen ontologia (GO) azterketak frogatu zuen DE gene gehienak polen hormaren garapenean parte hartu zutela.
3.DE mRNAen KEGG bidearen aberastearen analisiak (lerro antzuaren aldean gora eta behera erregulatutako mRNAak barne) erakutsi zuen "almidoia eta sakarosemetabolismoa", "fenilpropanoideen biosintesia" eta "pentosa eta glukuronatoen arteko konbertsioak" direla. ” izan ziren bide metaboliko aberastuenak.
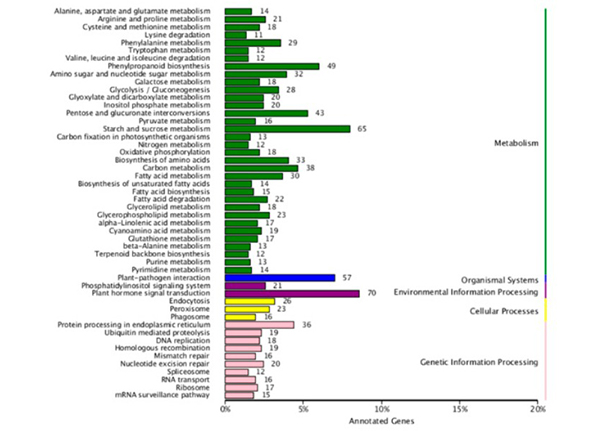 KEGG bidearen aberastearen azterketa diferentzial adierazitako mRNAen | 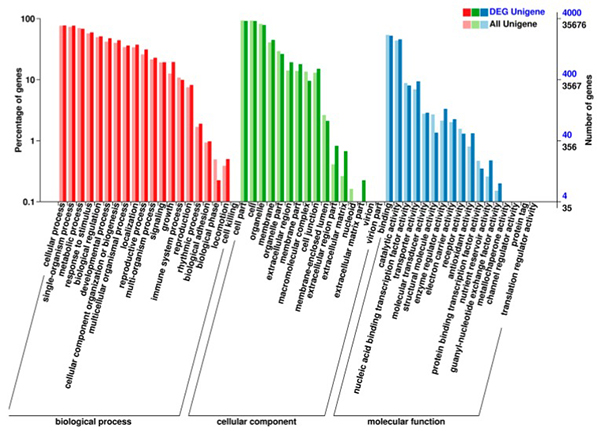 Gene ontologian (GO) sailkapen diferentzial adierazitako mRNA | 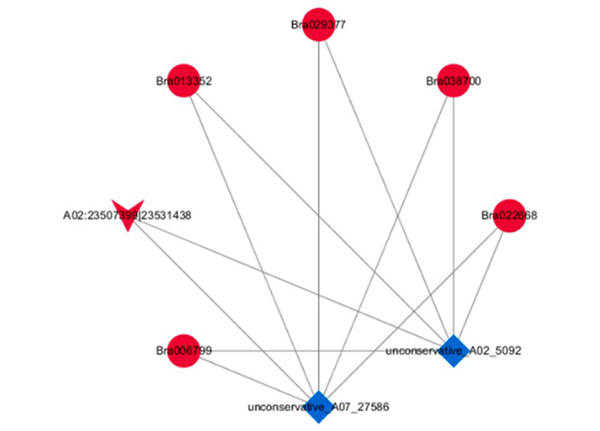 DEcircRNA–DEmiRNA–DEmRNA sare hirukoitzaren ikuspegia |
Erreferentzia
Liang Y, Zhang Y, Xu L, et al.CircRNA Adierazpen Eredua eta ceRNA eta miRNA-mRNA sareak Brassica campestris [J] CMS lerroan anter garapenean parte hartzen dute.Zientzia Molekularren Nazioarteko Aldizkaria, 2019, 20(19):4808-.DOI: 10.3390/ijms20194808














