GWAS
Pealkiri: Terve genoomi resekveneerimine paljastab Brassica napuse päritolu ja selle parandamisega seotud geneetilised lookused
Ajakiri: Looduskommunikatsioonid
NGS |WGS |Uuesti järjestamine |GWAS |Transkriptoom |RNAseq |Brassica napus |Evolutsioon |Kodustamine
Selles uuringus pakkus Biomarker Technologies NGS-i sekveneerimise teenuseid ja tehnilist tuge sekveneerimisandmete bioinformaatika analüüsimiseks.
Taust
Brassica napus(raps) on oluline õliseemnekultuur ja suurepärane mudel polüploidse spetsifikatsiooni, evolutsiooni ja selektsiooni protsesside uurimiseks.Siiski pole veel teada, kas looduslikud liigid või kodustatud doonorid olid vanemate eellased ja geenid, mis aitasid kaasa rapsiseemne kodustamisele ja paranemisele.
materjalid ja meetodid
Materjalid:588B. napusSellesse uuringusse kaasati liitujaid, sealhulgas 466 Aasiast, 102 Euroopast, 13 Põhja-Ameerikast ja 7 Austraaliast.Kasvuharjumuste andmete põhjal jaotati need materjalid kolme ökotüüpi;kevad (86 liitumist), talv (74 liitumist) ja pooltalv (428 liitumist).
Järjestus:Keskmiselt u.5× (vahemikus 3,37× kuni 7,71×)
Järjestusplatvorm:Illumina Hiseq 4000
Andmete tootmine:4,03 Tb puhtad andmed
SNP-kõne:BWA + GATK.Saadi 5 294 158 SNP-d ja 1 307 151 InDeli.
Tulemused
B. napuse päritolu
B. napusEuroopa naeri esivanemast arenes välja alamgenoom.Geenivoolu sündmus euroopa naerisest kuniB. napus Alamgenoom tekkis ~ 106–1170 aastat tagasi.B. napusC-alamgenoom võis areneda nende liinide ühisest esivanemast.EsivanemB. napuseraldunud nelja B. oleracea alamliigi ühisest esivanemast, kusjuures hiljutine geen voolab sisseB. napus~108–898 aastat tagasi.B. napusC-alamgenoomi päritolu on keerulisem kui A-alamgenoomil.Ajal tekkis mõlemas kahes subgenoomis tugev pudelikaelB. napusevolutsioon.Talv ja pooltalvB. napusökotüübid lahknesid ~60 aastat tagasi, talvel ja kevadelB. napuslahknesid ~416 aastat tagasi ning õliseemned ja mitteõliseemnedB. napuslahknes ~277 aastat tagasi.
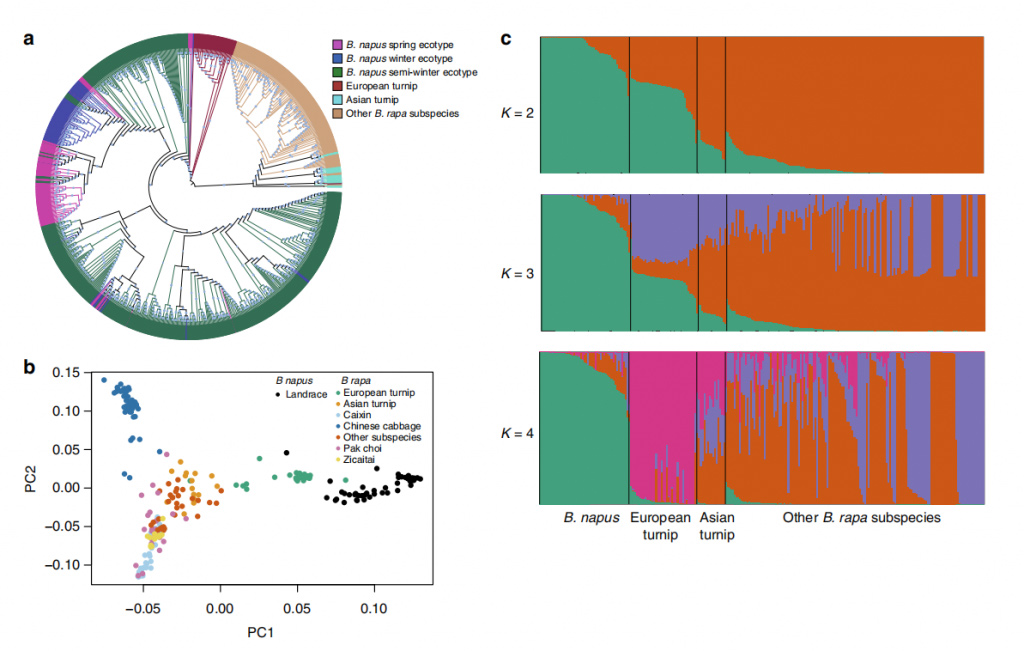
Joon. 2 588 B. napuse ja 199 B. rapa elaniku populatsiooni struktuur.
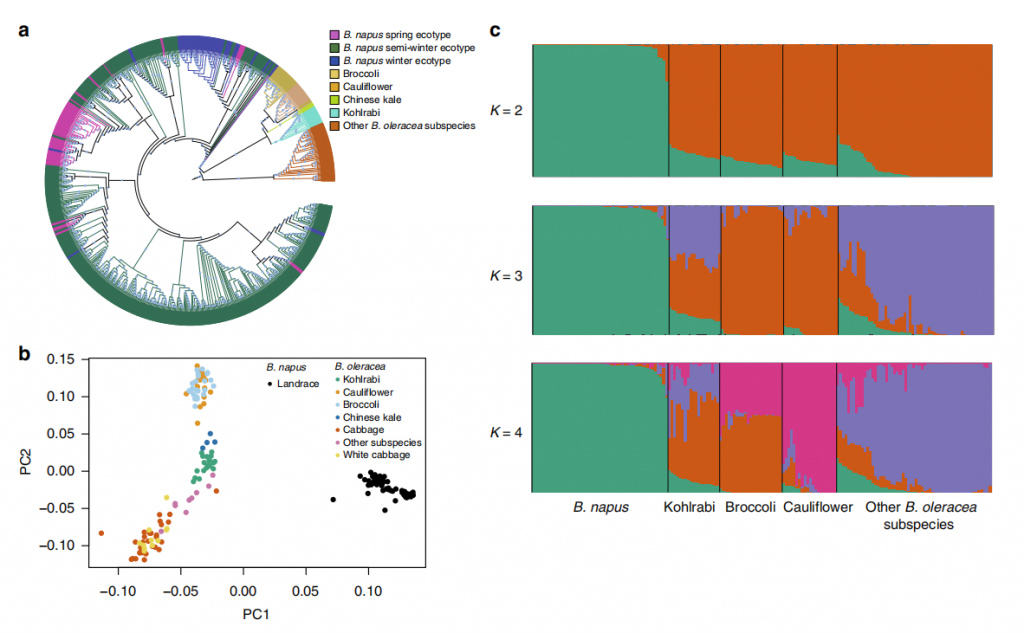
Joon. 3 588 B. napuse ja 119 B. oleracea nakatumise populatsiooni struktuur
Valikusignaalid ja kogu genoomi hõlmavad assotsiatsiooniuuringud.
Paranemise esimese etapi (FSI) ajal kadus B. napuse C subgenoomis suurem geneetiline mitmekesisus kui A-alamgenoomis.FSI ajal toimus vähem geneetilist diferentseerumist kui paranemise teises etapis (SSI).SSI-valiku signaalipiirkondade geene rikastati stressitaluvuse, arengu ja metaboolsete radadega.Tuvastati 60 lookust, mis olid märkimisväärselt seotud 10 sihtmärgiga, sealhulgas 5 oli seotud seemnesaagiga, 3 silikaalse pikkusega, 4 õlisisaldusega ja 48 seemne kvaliteediga.
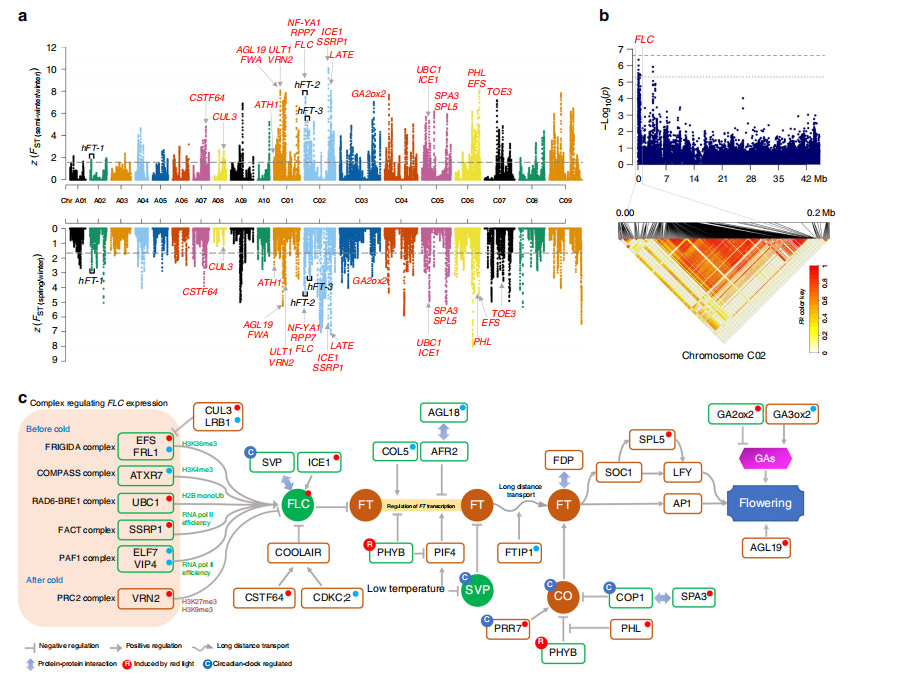
Joonis 4 Kogu genoomi hõlmav skaneerimine ja valitud piirkondade annotatsioonid B. napuse SSI ajal
Transkriptoomi analüüs
Suure õlisisaldusega ja topeltmadala kultivari ning madala õlisisaldusega ja topeltkõrge sordi identifitseeritud geenide RNAseq andmed, mis on seotud glükosinolaadi biosünteesiprotsessiga, olid oluliselt üleesindatud.
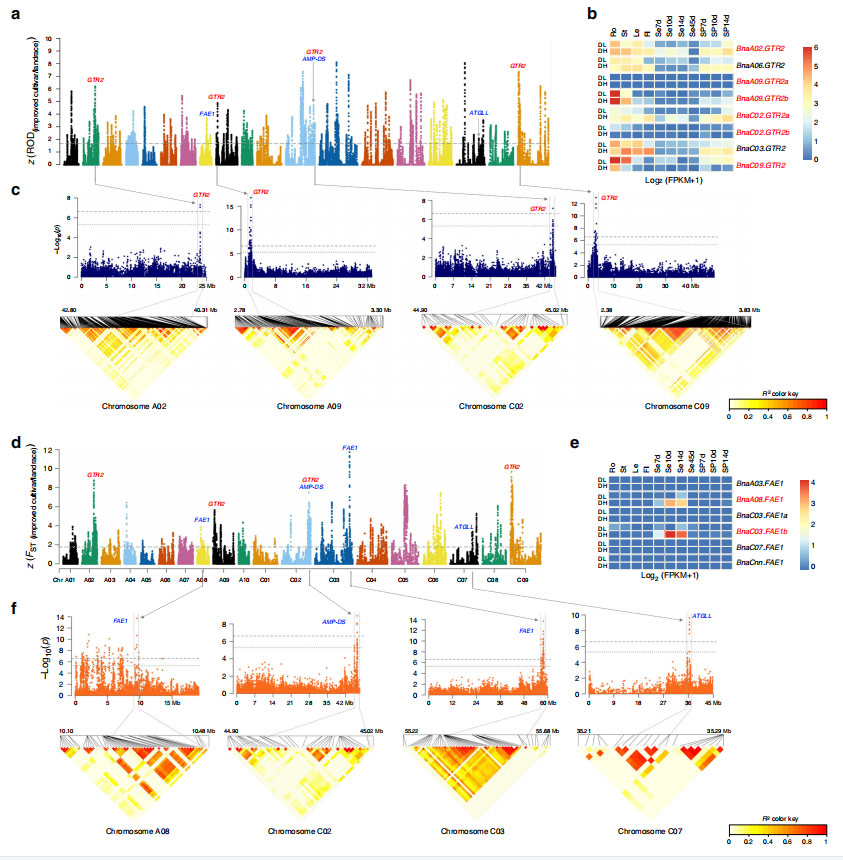
Joonis 5 Ülevaade õitsemisaja reguleerimisest B. napuse ökotüübi parandamise valikul
Arutelu
See uuring andis väärtusliku ressursi selle päritolu ja täiustamise ajaloo mõistmiseksB. napusja hõlbustab oluliste agrotehniliste komplekssete tunnuste geneetiliste aluste lahkamist.Soodsate variantide, selektsioonisignaalide ja kandidaatgeenidega seotud olulised SNP-d aitavad tulevikus suuresti kaasa, eriti selle hiljutise allopolüploidse põllukultuuri ja selle sugulaste saagikuse, seemnekvaliteedi, õlisisalduse ja kohanemisvõime parandamisel.
Viide
Terve genoomi resekveneerimine paljastab Brassica napuse päritolu ja selle paranemisega seotud geneetilised lookused [J].Looduskommunikatsioonid, 2019, 10(1).
Uudised ja tipphetked eesmärk on jagada uusimaid edukaid juhtumeid ettevõttega Biomarker Technologies, jäädvustada nii uudseid teadussaavutusi kui ka uuringu käigus kasutatud silmapaistvaid tehnikaid.
Postitusaeg: jaanuar 05-2022

