Secuenciación del exoma completo humano
Ventajas del servicio
● Región codificante de proteínas objetivo: al capturar y secuenciar la región codificante de proteínas, hWES se utiliza para revelar variantes relacionadas con la estructura de las proteínas;
● Alta precisión: con una alta profundidad de secuenciación, hWES facilita la detección de variantes comunes y raras con frecuencias inferiores al 1%;
● Rentable: hWES genera aproximadamente el 85 % de las mutaciones de enfermedades humanas a partir del 1 % del genoma humano;
● Cinco estrictos procedimientos de control de calidad que cubren todo el proceso con Q30>85% garantizado.
Especificaciones de muestra
| Plataforma
| Biblioteca
| Estrategia de captura de exones
| Recomendar estrategia de secuenciación
|
|
Plataforma Illumina NovaSeq
| PE150 | Agilent SureSelect Human All Exon V6 Panel híbrido IDT xGen Exome V2 | 5GB 10GB |
Requisitos de muestra
| Tipo de ejemplo
| Cantidad(Qubit®)
| Volumen
| Concentración
| Pureza (NanoDrop™) |
|
ADN genómico
| ≥ 300ng | ≥ 15 µL | ≥ 20 ng/μL | DO260/280=1,8-2,0 sin degradación, sin contaminación
|
Profundidad de secuenciación recomendada
Para trastornos mendelianos/enfermedades raras: profundidad de secuenciación efectiva superior a 50×
Para muestras de tumores: profundidad de secuenciación efectiva superior a 100×
Bioinformática


Flujo de trabajo del servicio

Entrega de muestra

Extracción de ADN

construcción de biblioteca

Secuenciación

Análisis de los datos

Entrega de datos

Servicios postventa
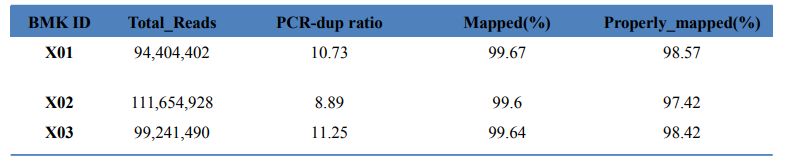
1.Estadísticas de alineación
Tabla 1 Estadísticas del resultado del mapa
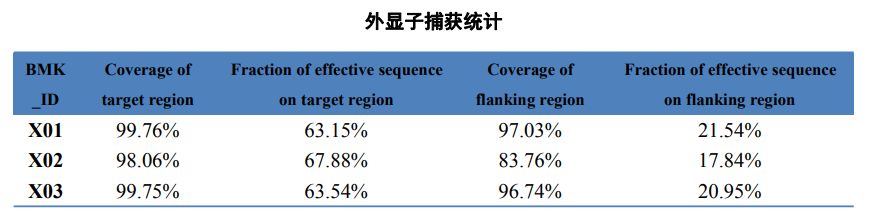
Tabla 2 Estadísticas de captura del exoma
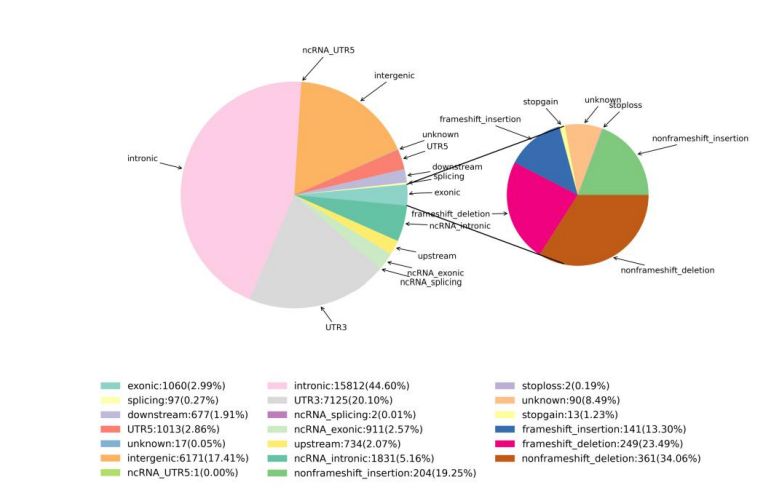
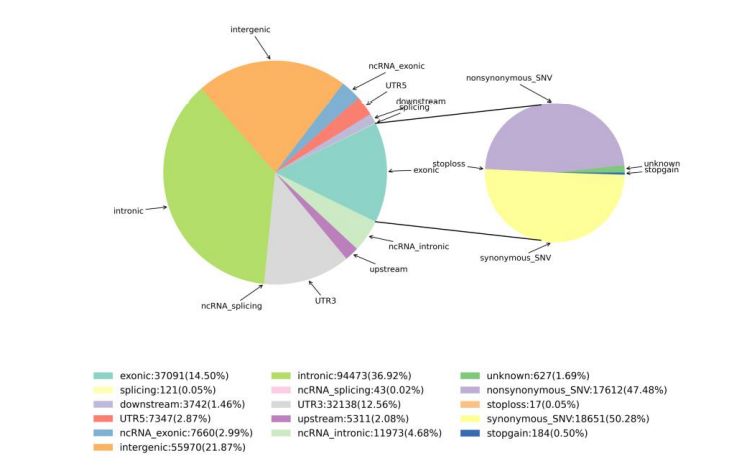
2.Detección de variaciones
Figura 1 Estadísticas de SNV e InDel
3.Análisis avanzado
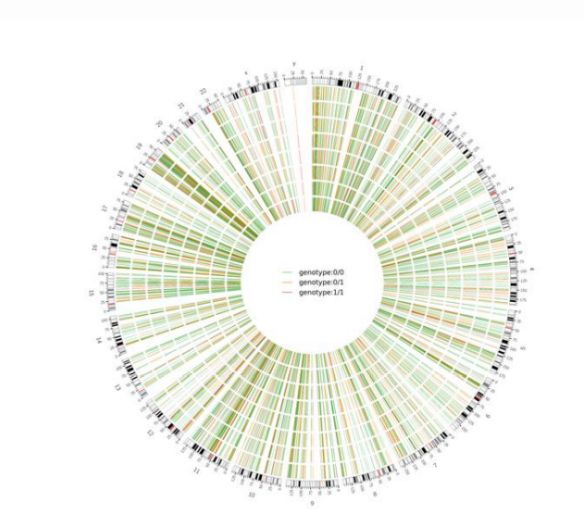
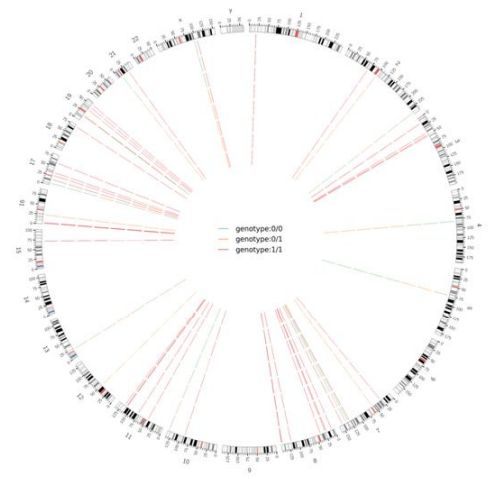
Figura 2 Gráfico Circos de SNV e InDel dañinos en todo el genoma
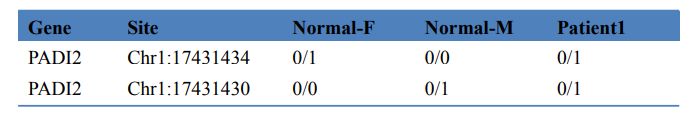
Tabla 3 Detección de genes causantes de enfermedades