Transcriptoma espacial BMKMANU S1000
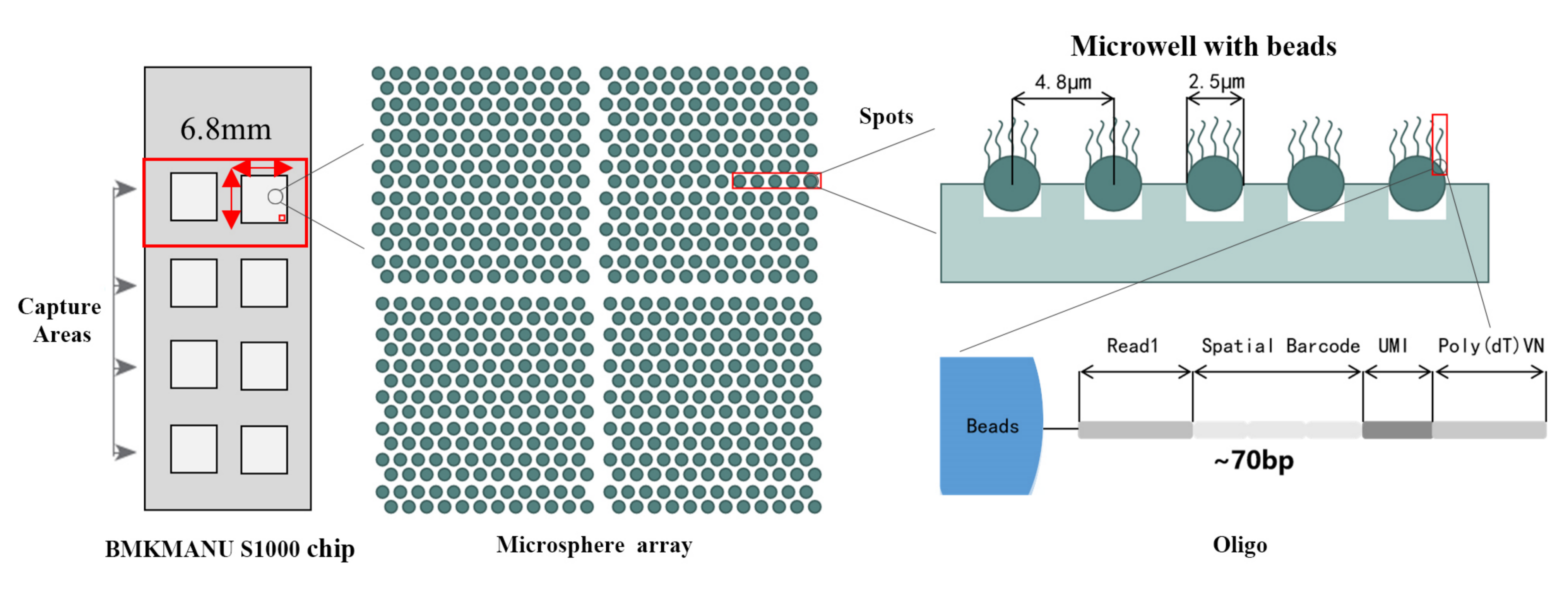
Esquema técnico del transcriptoma espacial BMKMANU S1000
Ventajas de BMKMANU S1000
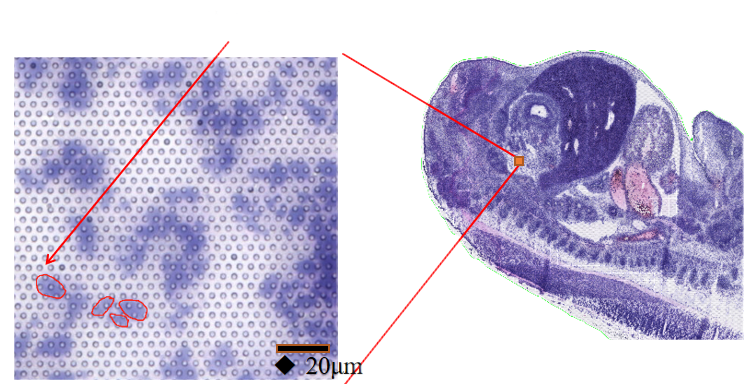
1) Resolución subcelular: cada área de captura contenía >2 millones de puntos espaciales con códigos de barras con un diámetro de 2,5 µm y un espacio de 5 µm entre los centros de los puntos, lo que permite el análisis del transcriptoma espacial con resolución subcelular (5 µm).
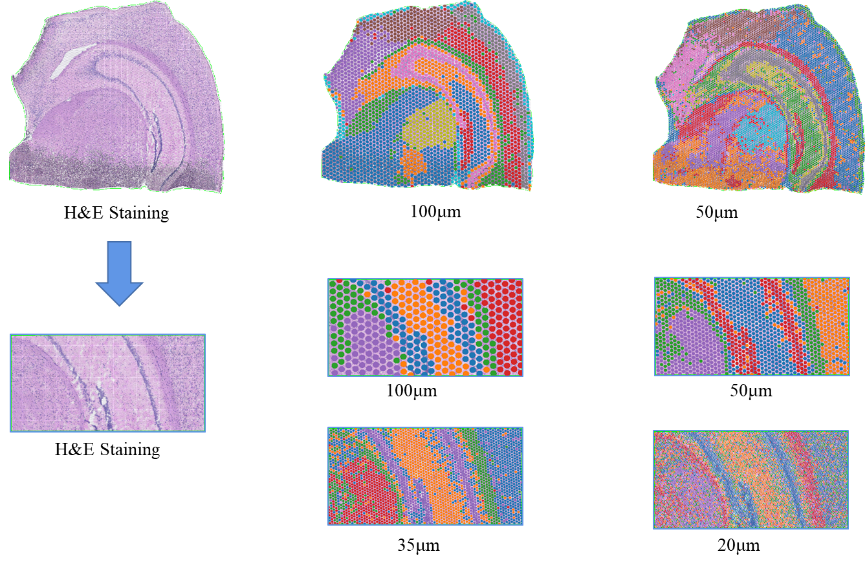
2) Análisis de resolución multinivel: análisis flexible de varios niveles que va desde 100 μm a 5 μm para resolver diversas características del tejido con una resolución óptima.
3) Perfiles completos del transcriptoma: las transcripciones capturadas de todo el portaobjetos de tejido se pueden analizar, sin restricción en el número de genes diana y el área objetivo.
Especificaciones de servicio
| Biblioteca | Estrategia de secuenciación | Salida de datos recomendada |
| Biblioteca de ADNc S1000 | BMKMANU S1000-Illumina PE150 | 60 Gb/muestra |
Requisitos de muestra
| Muestra | Número | Tamaño | Calidad del ARN |
| Bloque de tejido incluido en OCT | 2-3 bloques/muestra | Aprox.6,8x6,8x6,8mm3 | RIN≥7 |
Para obtener más detalles sobre la guía de preparación de muestras y el flujo de trabajo del servicio, no dude en hablar con unExperto en BMKGENE
Flujo de trabajo del servicio

Diseño de experimentos

Entrega de muestra

Pre-experimento
Código de barras espacial

construcción de biblioteca

Secuenciación

Análisis de los datos

Servicios postventa
Los datos generados por BMKMANU S1000 se analizan utilizando el software "BSTMatrix", diseñado de forma independiente por BMKGENE, que incluye:
1) Generación de matriz de expresión genética.
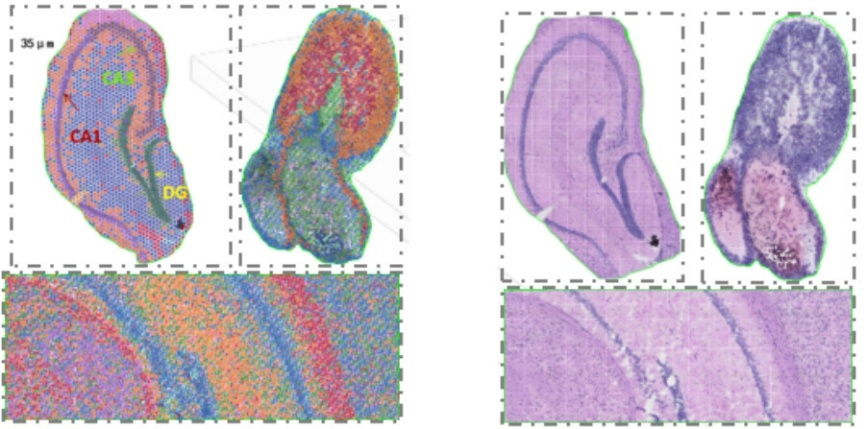
2) Procesamiento de imágenes HE
3) Compatible con software de terceros para análisis
4) El “BSTViewer” en línea ayuda a obtener resultados de visualización en diferentes resoluciones.
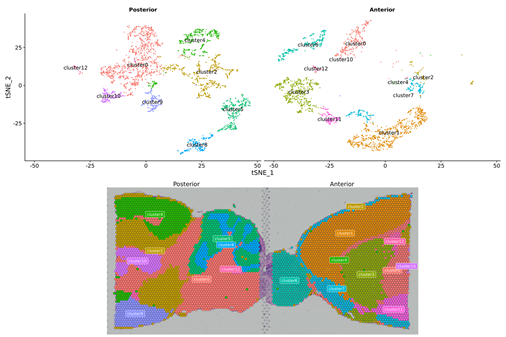
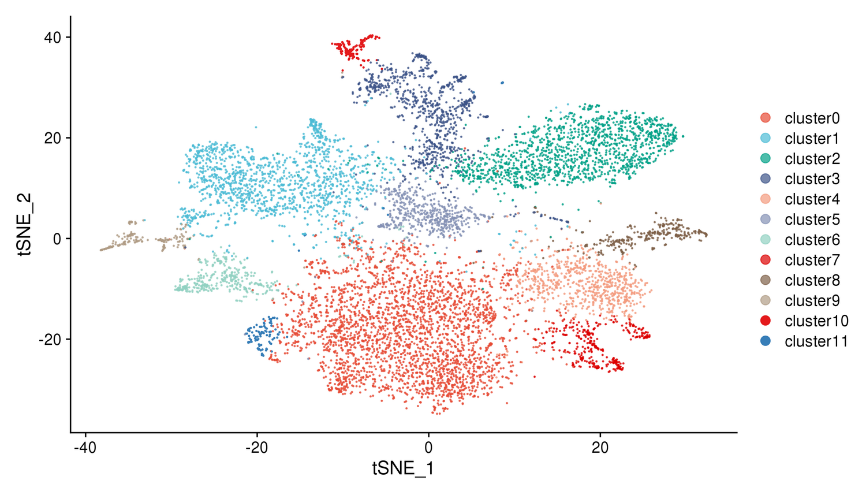
1.Agrupación puntual
2.Distribución espacial
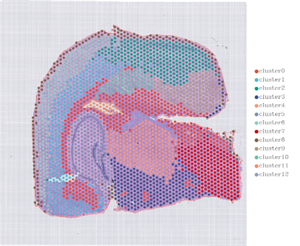
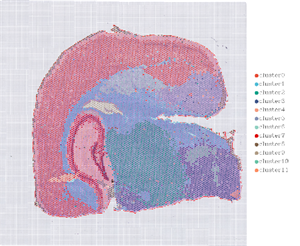
Nbeneficios según objetivos: Resoluciónnivel=13 (100 µm, izquierda); 7 (50 µm, bien)
3.Mapa de calor de agrupación de abundancia de expresión de marcadores
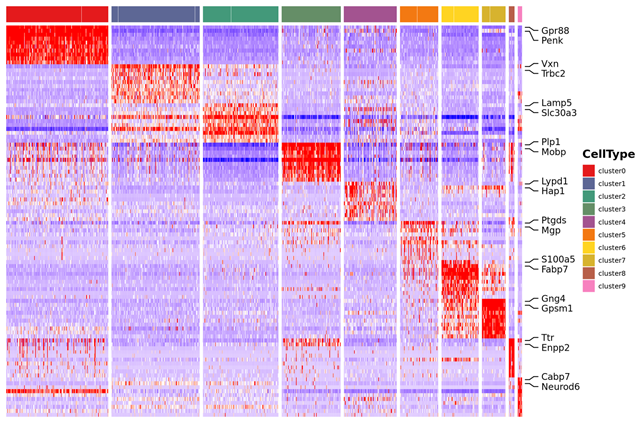
4.Análisis de datos entre muestras