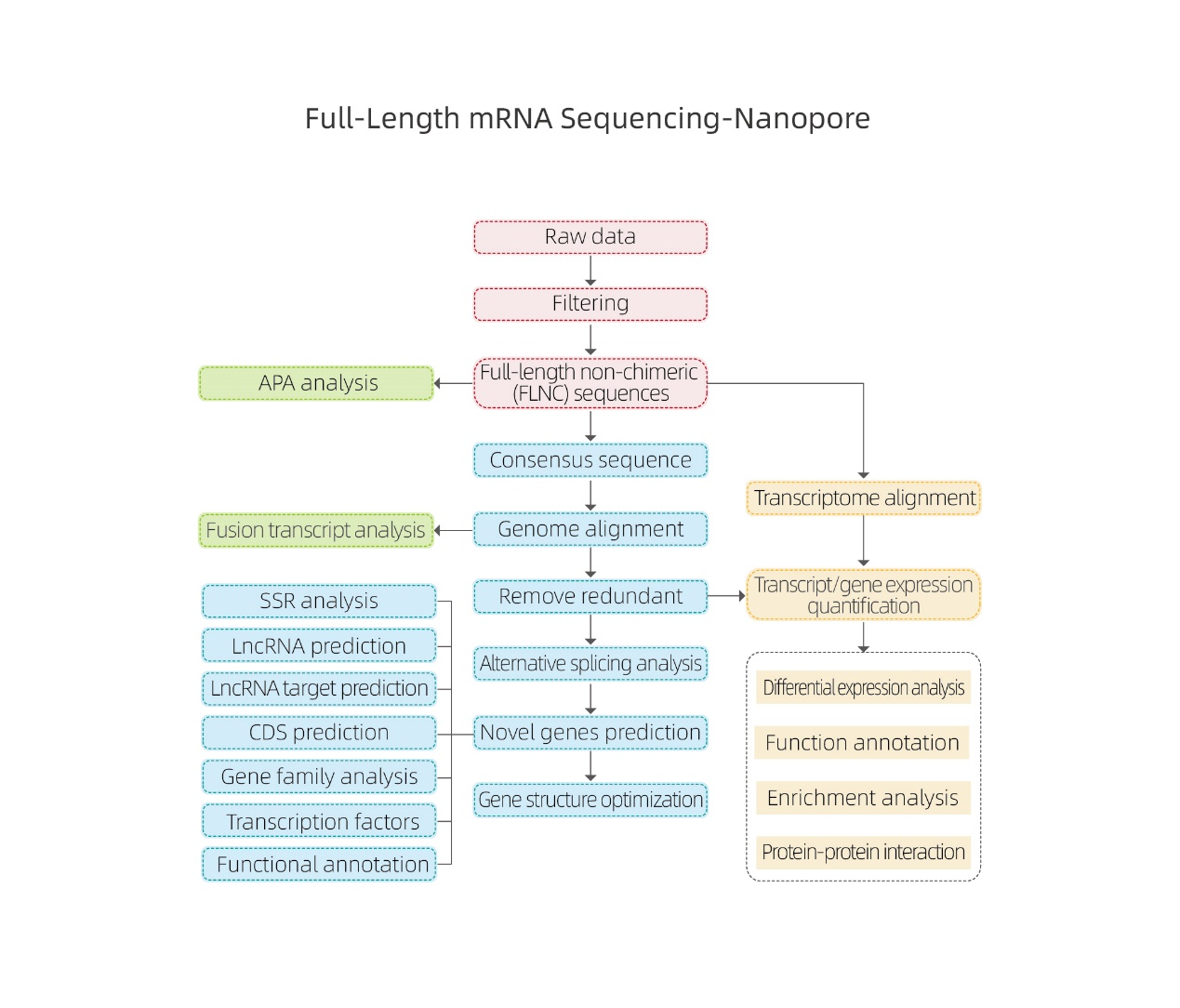
Plenlonga mRNA Sequencing-Nanopore
Servaj Avantaĝoj
● Malalta sekvenco biaso
● Malkaŝante plenlongajn cDNA-molekulojn
● Malpli da datumoj necesaj por kovri la saman nombron da transskribaĵoj
● Identigo de multoblaj izoformoj per geno
● Esprima kvantigo en izoforma nivelo
Specifoj de Servo
| Biblioteko | Platformo | Rekomendita datenrendimento (Gb) | Kontrolo de kvalito |
| cDNA-PCR (Riĉigita Poli-A) | Nanopore PromethION P48 | 6 Gb/provaĵo (Laŭ specio) | Plenlonga rilatumo> 70% Meza kvalita poentaro: Q10
|
Bioinformadikaj analizoj
●Kruda datumtraktado
● Transskriba identigo
● Alternativa splisado
● Esprim-kvantigo en gennivelo kaj izoforma nivelo
● Diferenca esprima analizo
● Funkcia komentario kaj riĉigo (DEG kaj DET)
Specimenaj Postuloj kaj Livero
Ekzemplaj Postuloj:
Nukleotidoj:
| Konk. (ng/μl) | Kvanto (μg) | Pureco | Integreco |
| ≥ 100 | ≥ 0,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Limigita aŭ neniu proteino aŭ DNA-poluado montrita sur ĝelo. | Por plantoj: RIN≥7.0; Por bestoj: RIN≥7.5; 5.0≥28S/18S≥1.0; limigita aŭ neniu bazlinia alteco |
Histo: Pezo (seka): ≥1 g
*Por histo pli malgranda ol 5 mg, ni rekomendas sendi fulmfrostitan (en likva nitrogeno) histo-provaĵo.
Ĉela suspendo: Ĉelkalkulo = 3×106- 1×107
*Ni rekomendas sendi frostitan ĉelan lizaton.Se tiu ĉelo nombras pli malgranda ol 5×105, fulmo frostigita en likva nitrogeno rekomendas, kio estas preferinda por mikro eltiro.
Sangaj specimenoj: Volumo≥1 ml
Rekomendita Specimena Livero
Ujo: 2 ml centrifuga tubo (Stana folio ne rekomendas)
Ekzempla etikedado: Grupo+replikato ekz. A1, A2, A3;B1, B2, B3... ...
Sendado: 2、Seka glacio: Specimenoj devas esti pakitaj en sakoj kaj enterigitaj en seka glacio.
- RNAstable-tuboj: RNA-provaĵoj povas esti sekigitaj en RNA-stabiliga tubo (ekz. RNAstable®) kaj senditaj en ĉambra temperaturo.
Serva Laborfluo
Nukleotidoj:

Specimena livero

Bibliotekokonstruo

Sekvencado

Analizo de datumoj

Post-vendaj servoj
Serva Laborfluo
histo:

Eksperimenta dezajno

Specimena livero

RNA-eltiro

Bibliotekokonstruo

Sekvencado

Analizo de datumoj

Post-vendaj servoj
1.Diferenciala esprimo analizo -Vulkanintrigo
Diferenciga esprimo-analizo povas esti prilaborita en kaj gennivelo por identigi diferencige esprimitajn genojn (DEGoj) kaj en izoforma nivelo por identigi diferencige.
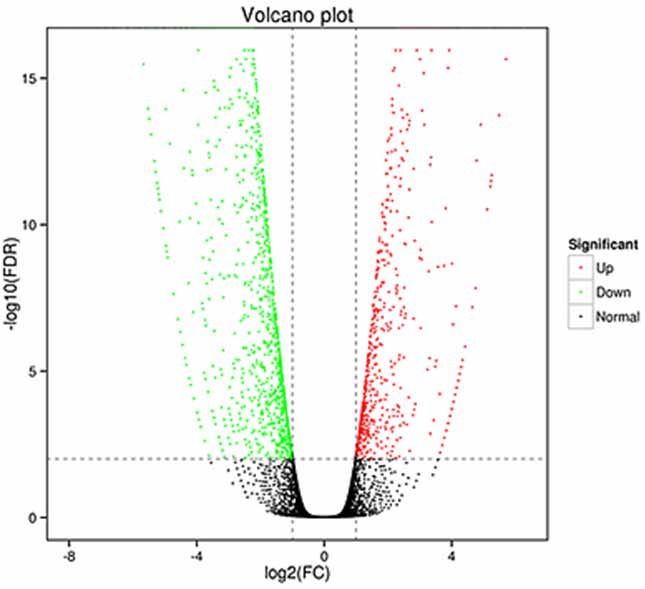
esprimitaj transskribaĵoj (DEToj)
2.Hierarkia grupiga varmomapo
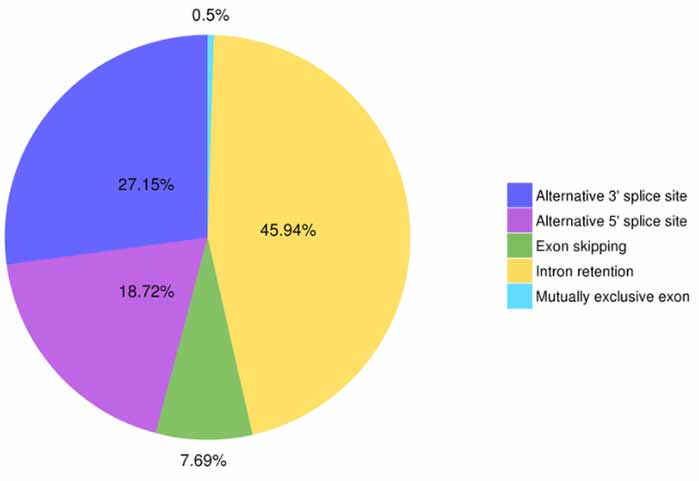
3.Alternativa splisanta identigo kaj klasifiko
Kvin specoj de alternativaj splisaj eventoj povas esti antaŭdiritaj de Astalavista.
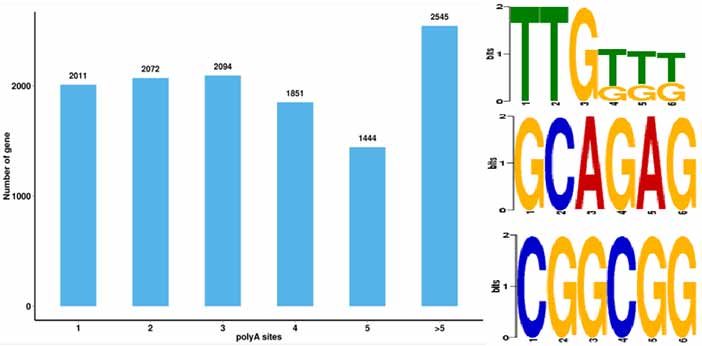
4.Alternativa poli-adenilation (APA) okazaĵidentigo kaj Motivo ĉe 50 bp kontraŭflue de poli-A
Kazo BMK
Alternativa splisada identigo kaj izoform-nivela kvantigo per nanopora plenlonga transkriptomsekvencado
Eldonita:Naturkomunikadoj, 2020
Sekvenca strategio:
Grupiĝo: 1. CLL-SF3B1(WT);2. CLL-SF3B1 (K700E mutacio);3. Normalaj B-ĉeloj
Sekvenca strategio: MinION 2D-biblioteko-sekvencado, PromethION 1D-biblioteko-sekvencado;mallonglegeblaj datumoj de samaj specimenoj
Sekvenca platformo: Nanopore MinION;Nanopore PromethION;
Ŝlosilaj rezultoj
1.Izoforma-nivela Alternativa Splisanta Identigo
Long-legataj sekvencoj povigas identigon de mutaciulo SF3B1K700E-ŝanĝitaj splisaj ejoj je izoforma nivelo.35 alternativaj 3′SS kaj 10 alternativaj 5′SS estis trovitaj esti signife diferencige splisitaj inter SF3B1K700Ekaj SF3B1WT.33 el la 35 ŝanĝoj estis lastatempe malkovritaj per longlegitaj sekvencoj.
2.Isoform-nivela Alternativa Splicing-kvantigo
Esprimo de intronreteno (IR) izoformoj en SF3B1K700Ekaj SF3B1WTestis kvantigitaj surbaze de nanoporsekvencoj, rivelante tutmondan malsupren-reguligon de IR-izoformoj en SF3B1K700E.
Referenco
Tang AD , Soulette CM , Baren MJV , et al.Plenlonga transskriba karakterizado de SF3B1-mutacio en kronika limfocita leŭkemio rivelas subreguligo de retenitaj intronoj [J].Naturkomunikadoj.